تسلسل زلق
التسلسل الزلق (بالإنجليزية: Slippery sequence) هو تسلسل قصير (عادة UUUAAAC) يتحكم في معدل واحتمال حدوث انزياح الإطار الريبوسومي. يتسبب التسلسل الزلق في انتقالٍ ريبوسوميٍ أسرع والذي يمكن أن يسبب بدوره «انزلاقا» لإطار القراءة الذي يقوم الريبوسوم بقراءته. يسمح هذا للرنا الناقل بالانزياح بقاعدة واحدة (-1) نحو النهاية 5' بعد أن يكون الكودون المضاد الخاص به قد ارتبط مع كودونٍ من الرنا الرسول وهو ما يغير إطار القراءة.[2][3][4][5][6] يُعرف انزياح الإطار -1 الذي يسببه هذا التسلسل بانزياح الإطار الريبوسومي -1 المبرمج. يلي هذا التسلسل الزلق منطقة فاصلة وبنية رنا ثانوية وتواجده شائع لدى الفيروسات التي تشفر عديدات البروتين.[1]
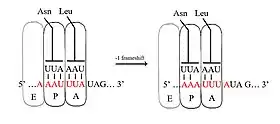
يحدث انزياح الإطار بسبب ترابط ووبل. تعطي طاقة غيبس الحرة الخاصة بالبنى الثانوية التي تلي التسلسل تلميحا وفكرة عن معدل حدوث انزياح الإطار.[7] يلعب التوتر الحاصل في جزيء الرنا الرسول دورا في الانزياح كذلك.[8] قائمة بالتسلسلات الزلقة الموجودة لدى الحيوانات متوفرة في ورقة هوانغ وزملائه.[9]
التسلسلات الزلقة التي تسبب انزياحا بـ -2 قاعدة (انزياح إطار -2) تم إنشاؤها اعتمادا على تسلسل فيروس عوز المناعة البشري UUUUUUA .[8]
مراجع
- Jacks T, Madhani HD, Masiarz FR, Varmus HE (نوفمبر 1988)، "Signals for ribosomal frameshifting in the Rous sarcoma virus gag-pol region"، Cell، 55 (3): 447–58، doi:10.1016/0092-8674(88)90031-1، PMC 7133365، PMID 2846182، S2CID 25672863.
- Green L, Kim CH, Bustamante C, Tinoco I (يناير 2008)، "Characterization of the mechanical unfolding of RNA pseudoknots"، Journal of Molecular Biology، 375 (2): 511–28، doi:10.1016/j.jmb.2007.05.058، PMC 7094456، PMID 18021801.
- Yu CH, Noteborn MH, Olsthoorn RC (ديسمبر 2010)، "Stimulation of ribosomal frameshifting by antisense LNA"، Nucleic Acids Research، 38 (22): 8277–83، doi:10.1093/nar/gkq650، PMC 3001050، PMID 20693527.
- "Dr Ian Brierley Research description"، Department of Pathology, University of Cambridge، مؤرشف من الأصل في 02 أكتوبر 2013، اطلع عليه بتاريخ 28 يوليو 2013.
- "Molecular Biology: Frameshifting occurs at slippery sequences"، Molecularstudy.blogspot.com، 16 أكتوبر 2012، مؤرشف من الأصل في 29 مارس 2021، اطلع عليه بتاريخ 28 يوليو 2013.
- Farabaugh PJ, Björk GR (مارس 1999)، "How translational accuracy influences reading frame maintenance"، The EMBO Journal، 18 (6): 1427–34، doi:10.1093/emboj/18.6.1427، PMC 1171232، PMID 10075915.
- Cao S, Chen SJ (مارس 2008)، "Predicting ribosomal frameshifting efficiency"، Physical Biology، 5 (1): 016002، Bibcode:2008PhBio...5a6002C، doi:10.1088/1478-3975/5/1/016002، PMC 2442619، PMID 18367782.
- Lin Z, Gilbert RJ, Brierley I (سبتمبر 2012)، "Spacer-length dependence of programmed -1 or -2 ribosomal frameshifting on a U6A heptamer supports a role for messenger RNA (mRNA) tension in frameshifting"، Nucleic Acids Research، 40 (17): 8674–89، doi:10.1093/nar/gks629، PMC 3458567، PMID 22743270.
- Huang X, Cheng Q, Du Z (2013)، "A genome-wide analysis of RNA pseudoknots that stimulate efficient -1 ribosomal frameshifting or readthrough in animal viruses"، BioMed Research International، 2013: 984028، doi:10.1155/2013/984028، PMC 3835772، PMID 24298557.
- بوابة علم الأحياء الخلوي والجزيئي
- بوابة الكيمياء الحيوية