سلالة الخلية
تشير سُلالَة الخلية (بالإنجليزية: Cell lineage) إلى التاريخ التطوري لنسيج أو عضو من الجنين المخصب.[1] يعتمد هذا على تتبع السلالة الخلوية للكائن الحي بسبب الانقسامات الخلوية والانتقال مع تقدم الوقت، ويبدأ هذا بالخلايا المنشئة وينتهي بخلية ناضجة لم تعد قادرة على الانقسام.[1]
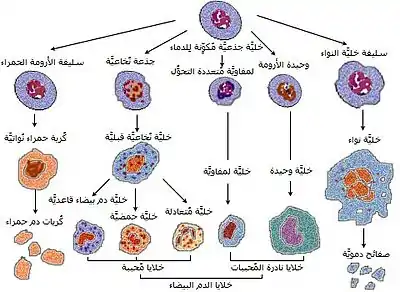
يمكن دراسة هذا النوع من النسب عن طريق تعليم الخلية (بجزيئات الفلورسنت أو غيرها من العلامات التي يمكن تتبعها) واتباع نسلها بعد انقسام الخلية. بعض الكائنات الحية، مثل ديدان الربداء الرشيقة، لديها نمط محدد مسبقاً من ذرية الخلية وسيتكون الذكر البالغ دائمًا من 1031 خلية؛ وذلك لأن الانقسام الخلوي في ديدان الربداء الرشيقة يتم تحديده وراثياً والمعروفة بإتلي.[2][3] يؤدي هذا إلى ارتباط سلالة الخلية ومصير الخلية ارتباطاً وثيقاً. الكائنات الحية الأخرى، مثل البشر، لها سلالات متغيرة وأعداد خلايا جسدية.
كائن نموذج: ديدان ربداء رشيقة
كواحد من الرواد الأوائل في سلالة الخلايا، بدأ الدكتور سيدني برينر في الستينيات في مراقبة تمايز الخلايا وتعاقبها في الديدان الخيطية الربداء الرشيقة. اختار الدكتور برينر هذا الكائن الحي بسبب جسمه الشفاف، والتكاثر السريع، وسهولة الوصول، والحجم الصغير مما جعله مثالياً لمتابعة سلالة الخلايا تحت المجهر.
بحلول عام 1976، كان الدكتور برينر وشريكه الدكتور جون سولستون، قد حددوا جزءاً من سلالة الخلية في الجهاز العصبي النامي للبكتيريا الرشيقة. أظهرت النتائج الأولية أن الديدان الخيطية كانت سهلة الانصهار (كل فرد لديه نفس مسارات التمايز)، لكن العمل الذي قام به سولستون وريتشارد هورفيتز أظهر أن العديد من الخلايا الضرورية للتكاثر تتمايز بعد الفقس. وتشمل هذه الخلايا خلايا الفرج وكذلك العضلات والخلايا العصبية. أدى هذا البحث أيضاً إلى الملاحظات الأولية لموت الخلايا المبرمج أوالاستماتة.[4]
بعد رسم خرائط لأقسام مختلفة من سلالة خلايا الربداء الرشيقة. حصلوا في وقت لاحق على جائزة نوبل لعام 2002 لعملهم في التنظيم الجيني لتطور الأعضاء وموت الخلايا المبرمج. نظراً لأن الربداء الرشيقة خنثى، هناك أعضاء من الذكور والإناث، حيث يخزنون الحيوانات المنوية ويكونون قادرين على الإخصاب الذاتي. تحتوي الربداء الرشيقة على 302 خلية عصبية و959 خلية جسدية، حيث تبدأ بالرقم 1031، حيث تخضع 72 خلية للاستماتة وهو موت الخلية المبرمج. هذا يجعل الربداء الرشيقة كائن نموذجي لدراسة سلالة الخلية والقدرة على مراقبة انقسامات الخلية بسبب النمط الظاهري الشفاف لها.[4]
تاريخ سلالة الخلية
أُجرِيَت إحدى الدراسات الأولى على سلالات الخلايا في سبعينيات القرن التاسع عشر بواسطة ويتمان الذي درس أنماط الانقسام في العلق واللافقاريات الصغيرة. وجد أن بعض المجموعات، مثل الديدان الخيطية والديدان الأسكيدية تشكل نمطًا من الانقسام الخلوي متطابقاً بين الأفراد وثابتاً. كان يُعتقد أن هذا الارتباط الكبير بين سلالة الخلية ومصير الخلية يتم تحديده من خلال عوامل الفصل داخل الخلايا المنقسمة. الكائنات الحية الأخرى لديها أنماط من الانقسام الخلوي وأنتجت سلالات فرعية كانت من ذرية خلايا سليفة معينة. يُعتقد أن مصائر الخلايا الأكثر تنوعاً ترجع إلى تفاعل الخلايا مع البيئة. بسبب الاختراقات الجديدة في تتبع الخلايا بدقة أكبر، ساعد هذا المجتمع البيولوجي حيث يتم الآن استخدام مجموعة متنوعة من الألوان في إظهار الخلايا الأصلية والقدرة على التتبع بسهولة. هذه الألوان مشعة ومميزة بالبروتينات عن طريق إعطاء الحقن لتتبع هذه الخلايا.[4]
تقنيات رسم خرائط المصير
يمكن تحديد نسب الخلية بطريقتين، إما من خلال الملاحظة المباشرة أو من خلال التحليل النسيلي. خلال أوائل القرن التاسع عشر، تم استخدام الملاحظة المباشرة ولكنها كانت محدودة للغاية حيث يمكن دراسة عينات شفافة صغيرة فقط. مع اختراع المجهر متحد البؤر، سمح هذا بدراسة كائنات أكبر وأكثر تعقيداً.[5]
ربما تكون الطريقة الأكثر شيوعاً لرسم خرائط مصير الخلية في العصر الجيني هي من خلال إعادة التركيب الخاص بالموقع بوساطة أنظمة Cre-Lox أو FLP-FRT. من خلال استخدام أنظمة إعادة التركيب Cre-Lox أو FLP-FRT، يتم تنشيط الجين المرشد (عادةً ما يشفر البروتين الفلوري) ويصنف بشكل دائم الخلية المعنية وخلايا نسلها، وبالتالي اسم تتبع نسب الخلية الاسمية.[5] باستخدام هذا النظام يمكن للباحثين التحقيق في وظيفة الجين المفضل لديهم في تحديد مصير الخلية عن طريق تصميم نموذج جيني حيث يتم تصميم حدث إعادة التركيب داخل الخلية لمعالجة الجين المعني بينما تم تصميم حدث إعادة التركيب الآخر لتنشيط الجين المرشد. تتمثل إحدى المشكلات الثانوية في أن حدثي إعادة التركيب قد لا يحدثان في وقت واحد وبالتالي يجب تفسير النتائج بحذر.[6] علاوة على ذلك، فإن بعض مراسلي الفلوريسنت لديهم عتبة إعادة تركيب منخفضة للغاية بحيث يمكنهم تصنيف مجموعات الخلايا في نقاط زمنية غير مرغوب فيها في غياب الحث.[6]
في الآونة الأخيرة، بدأ الباحثون في استخدام مناهج البيولوجيا التركيبية ونظام CRISPR/Cas9 لهندسة أنظمة وراثية جديدة تمكن الخلايا من تسجيل معلومات السلالة بشكل مستقل في الجينوم الخاص بها. تعتمد هذه الأنظمة على طفرة هندسية موجهة لعناصر وراثية محددة.[7][8] من خلال توليد تعديلات جينومية جديدة وعشوائية في كل جيل من الخلايا، تسهل هذه الأساليب إعادة بناء أشجار النسب. تَعِدْ هذه الأساليب بتقديم تحليل أكثر شمولاً لعلاقات النسب في الكائنات الحية النموذجية. طرق إعادة بناء الشجرة الحسابية يتم تطويرها أيضاً[9] لمجموعات البيانات التي تم إنشاؤها بواسطة هذه الأساليب.
عدم التناسق النمائي المبكر
في البشر بعد الإخصاب، تنقسم البيضة الملقحة إلى خليتين. يمكن استخدام الطفرات الجسدية التي تظهر مباشرة بعد تكوين البيضة الملقحة، وكذلك في وقت لاحق من التطور، كعلامات لتتبع سلالات الخلايا في جميع أنحاء الجسم. بدءاً من انقسامات الزيجوت، لوحظ أن السلالات تساهم بشكل غير متساوٍ في خلايا الدم. تم العثور على ما يصل إلى 90% من خلايا الدم مشتقة من واحد فقط من أول اثنين من القُسيم الأرومي. بالإضافة إلى ذلك، قد يؤدي التطور الطبيعي إلى خصائص غير متكافئة للأعضاء المتناظرة، مثل بين القشرة الدماغية اليمنى واليسرى الأمامية والقذالية. تم اقتراح أن كفاءة إصلاح الحمض النووي تساهم في عدم توازن النسب، حيث أن الوقت الإضافي الذي تقضيه الخلية في إصلاح الحمض النووي قد يقلل من معدل الانتشار.[10]
مراجع
- Collins English Dictionary - Complete & Unabridged 10th Edition.، HarperCollins Publishers.، مؤرشف من الأصل في 6 أكتوبر 2015، اطلع عليه بتاريخ 02 يونيو 2014.
- Sulston, JE؛ Horvitz, HR (1977)، "Post-embryonic cell lineages of the nematode, Caenorhabditis elegans"، Developmental Biology، 56 (1): 110–56، doi:10.1016/0012-1606(77)90158-0، PMID 838129.
- Kimble, J؛ Hirsh, D (1979)، "The postembryonic cell lineages of the hermaphrodite and male gonads in Caenorhabditis elegans"، Developmental Biology، 70 (2): 396–417، doi:10.1016/0012-1606(79)90035-6، PMID 478167.
- "The Nobel Prize in Physiology or Medicine for 2002 - Press Release"، www.nobelprize.org، مؤرشف من الأصل في 8 أبريل 2018، اطلع عليه بتاريخ 23 نوفمبر 2015.
- Chisholm, A D (2001)، "Cell Lineage" (PDF)، Encyclopedia of Genetics، ص. 302–310، doi:10.1006/rwgn.2001.0172، ISBN 9780122270802.[وصلة مكسورة]
- Liu, J؛ Willet, SG؛ Bankaitis, ED (2013)، "Non-parallel recombination limits Cre-LoxP-based reporters as precise indicators of conditional genetic manipulation"، Genesis، 51 (6): 436–42، doi:10.1002/dvg.22384، PMC 3696028، PMID 23441020.
- McKenna, Aaron؛ Findlay, Gregory M.؛ Gagnon, James A.؛ Horwitz, Marshall S.؛ Schier, Alexander F.؛ Shendure, Jay (29 يوليو 2016)، "Whole-organism lineage tracing by combinatorial and cumulative genome editing"، Science (باللغة الإنجليزية)، 353 (6298): aaf7907، doi:10.1126/science.aaf7907، ISSN 0036-8075، PMC 4967023، PMID 27229144.
- Frieda, Kirsten L.؛ Linton, James M.؛ Hormoz, Sahand؛ Choi, Joonhyuk؛ Chow, Ke-Huan K.؛ Singer, Zakary S.؛ Budde, Mark W.؛ Elowitz, Michael B.؛ Cai, Long (2017)، "Synthetic recording and in situ readout of lineage information in single cells"، Nature، 541 (7635): 107–111، Bibcode:2017Natur.541..107F، doi:10.1038/nature20777، PMC 6487260، PMID 27869821.
- Zafar, Hamim؛ Lin, Chieh؛ Bar-Joseph, Ziv (2020)، "Single-cell lineage tracing by integrating CRISPR-Cas9 mutations with transcriptomic data"، Nature Communications (باللغة الإنجليزية) (3055)، doi:10.1038/s41467-020-16821-5، PMC 7298005، PMID 32546686.
- Fasching L, Jang Y, Tomasi S, Schreiner J, Tomasini L, Brady MV, Bae T, Sarangi V, Vasmatzis N, Wang Y, Szekely A, Fernandez TV, Leckman JF, Abyzov A, Vaccarino FM. Early developmental asymmetries in cell lineage trees in living individuals. Science. 2021 Mar 19;371(6535):1245-1248. doi: 10.1126/science.abe0981. PMID 33737484
- بوابة علم الأحياء الخلوي والجزيئي