Comunidad microbiana
Un consorcio microbiano o comunidad microbiana[1] son dos o más grupos bacterianos o microbianos que viven simbióticamente.[2][3] Los consorcios pueden ser endosimbióticos o ectosimbióticos, o en ocasiones pueden ser ambos.
El protista Mixotricha paradoxa, en sí mismo un endosimbionte de la termita Mastotermes darwiniensis, siempre se encuentra como un consorcio de al menos un coco endosimbiótico, múltiples especies ectosimbióticas de bacterias flageladas o ciliadas y al menos una especie de bacteria Treponema helicoidal que forma la base de locomoción de los protistas mixotricha.[4]
El concepto de consorcio fue introducido por primera vez por Johannes Reinke en 1872,[5][6] y en 1877 se introdujo el término simbiosis y luego se amplió. La evidencia de simbiosis entre microbios sugiere que fue un precursor necesario de la evolución de las plantas terrestres y de su transición de las comunidades de algas en el mar a la tierra.[7]
Visión general
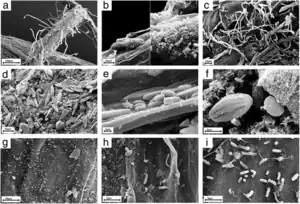
Los microbios tienen un potencial de aplicación prometedor para aumentar la eficiencia de los bioprocesos cuando se trata de sustancias resistentes a la descomposición.[8][9] Se ha aislado un gran número de microorganismos en función de su capacidad para degradar materiales recalcitrantes como la lignocelulosa y los poliuretanos.[10][11] En muchos casos de eficiencia de degradación, los consorcios microbianos han resultado superiores en comparación con las cepas individuales.[12] Por ejemplo, nuevos consorcios termofílicos de Brevibacillus spp. y Aneurinibacillus sp. se han aislado del medio ambiente para mejorar la degradación del polímero.[13]
Obtención de comunidades
Existen dos enfoques para obtener consorcios microbianos que involucran (i) un ensamblaje sintético desde cero mediante la combinación de varias cepas[13] o (ii) la obtención de comunidades microbianas complejas a partir de muestras ambientales.[14]
A partir de muestras ambientales, el proceso de enriquecimiento se utiliza a menudo para obtener los consorcios microbianos deseados.[15][16][17]
Por ejemplo, un consorcio derivado de termitas que mostraba una alta actividad xilanasa se enriqueció con paja de trigo crudo como única fuente de carbono, que pudo transformar la lignocelulosa en carboxilatos en condiciones anaeróbicas.[18]
Todavía se observan niveles de diversidad relativamente altos a pesar del uso de pasos de enriquecimiento cuando se trabaja con muestras ambientales,[17] probablemente debido a la alta redundancia funcional observada en las comunidades microbianas ambientales, siendo esto un elemento clave para su estabilidad funcional.[19][20] Esta diversidad intrínseca puede constituir un cuello de botella en los intentos de avanzar hacia la aplicación práctica debido a (i) la posible correlación negativa con la eficiencia,[21] (ii) los microbios tramposos cuya presencia no tiene impacto en la degradación, (iii) las amenazas a la seguridad planteadas por la presencia de patógenos conocidos o desconocidos, y (iv) el riesgo de perder las propiedades de interés si se apoya en especies raras.[22]
La utilización de consorcios microbianos con menos complejidad, pero con la misma eficiencia, puede conducir a procesos industriales más controlados y optimizados para el humano.[23] Por ejemplo, una gran proporción de genes funcionales se ha alterado notablemente para incrementar la eficiencia de la biodegradación del diesel al reducir la biodiversidad de una comunidad microbiana de suelos contaminados con diesel.[24] Por lo tanto, es crucial encontrar estrategias confiables para reducir la diversidad hacia consorcios microbianos optimizados obtenidos de muestras ambientales. Se han aplicado enfoques de detección reductiva para construir consorcios microbianos mínimos efectivos para la degradación de la lignocelulosa basados en diferentes grupos funcionales metabólicos. [23] Además, también se han empleado enfoques de selección artificial (dilución, toxicidad y calor) para obtener consorcios bacterianos.[25] Entre ellos, la dilución hasta la extinción, ha demostrado su eficacia para obtener consorcios microbianos funcionales a partir del agua de mar y el licor del rumen. [26][27][28] Se espera que la dilución hasta la extinción proporcione más ventajas en comparación con el aislamiento y el ensamblaje convencionales, ya que (i) genera muchas combinaciones microbianas listas para ser examinadas, (ii) incluye cepas del grupo microbiano inicial que podrían perderse debido al cultivo / aislamiento sesgos, y (iii) asegura que todos los microbios estén físicamente presentes e interactúen espontáneamente. [29][22]
Ejemplos de comunidades
Rizosfera
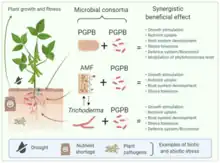
Aunque varios estudios han demostrado que un solo microorganismo puede ejercer efectos beneficiosos en las plantas, es cada vez más evidente que cuando se involucra un consorcio microbiano (dos o más microorganismos que interactúan), se pueden esperar resultados aditivos o sinérgicos. Esto ocurre, en parte, debido al hecho de que múltiples especies pueden realizar una variedad de tareas en un ecosistema como ocurre con la rizosfera de las raíces de las plantas. Los mecanismos benéficos de la estimulación del crecimiento de las plantas incluyen una mayor disponibilidad de nutrientes, modulación de fitohormonas control biológico, tolerancia al estrés biótico y abiótico) ejercida por diferentes agentes microbianos dentro de la rizosfera, como las bacterias promotoras del crecimiento de las plantas y hongos como Trichoderma y micorrizas.[30]
Los microorganismos de la rizosfera como las bacterias promotoras del crecimiento de las plantas, los hongos micorrízicos arbusculares y los hongos del género Trichoderma pueden establecer interacciones benéficas con las plantas, promoviendo el crecimiento y desarrollo de las plantas, aumentando el sistema de defensa de las plantas contra patógenos, promoviendo la absorción de nutrientes y mejorando la tolerancia a diferentes estreses ambientales. Los microorganismos de la rizosfera pueden influirse entre sí, y los consorcios resultantes de (por ejemplo, una bacteria fijadora de nitrógeno como Rhizobium spp. y Pseudomonas fluorescens), pueden tener efectos sinérgicos sobre el crecimiento y la aptitud de las plantas, proporcionando a la planta mayores beneficios para superar el estrés biótico y abiótico.[30]
La capacidad de los microbios para degradar materiales recalcitrantes se ha explorado ampliamente para la remediación ambiental y la producción industrial. Se han logrado logros significativos con cepas únicas, pero ahora el enfoque se dirige hacia el uso de consorcios microbianos que se apropien de su estabilidad funcional y eficiencia. Sin embargo, el ensamblaje de consorcios microbianos simplificados de comunidades ambientales complejas aún está lejos de ser útil, debido a la gran diversidad y el efecto de las interacciones bióticas.[22]
Las queratinas son materiales fibrosos recalcitrantes con componentes reticulados, que representan las proteínas más abundantes en las células epiteliales.[31] Se estima que tienen un valor económico considerable después de la biodegradación.[32] Un consorcio microbiano queratinolítico eficiente (KMCG6) se enriqueció previamente a partir de una muestra ambiental mediante cultivo en medio de queratina.[17] A pesar de reducir la diversidad microbiana durante el proceso de enriquecimiento, KMCG6 todavía incluía varias OTU dispersas entre siete géneros bacterianos.[22]
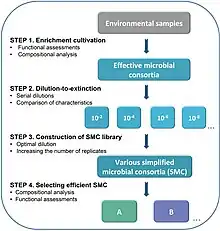
En 2020 Kang et al., Utilizando una estrategia basada en cultivos de enriquecimiento y dilución hasta la extinción, extrajeron de este consorcio original (KMCG6) un consorcio microbiano simplificado (SMC) con menos especies pero actividad queratinolítica similar.[22] Se realizaron diluciones en serie en un consorcio microbiano queratinolítico enriquecido previamente a partir de una muestra de suelo. Se seleccionó un régimen de dilución apropiado (10 9 ) para construir una biblioteca de SMC del consorcio microbiano enriquecido. El análisis de secuenciación adicional y los ensayos de actividad queratinolítica demostraron que las SMC obtenidas mostraban una diversidad microbiana reducida real, junto con diversas capacidades de composición taxonómica y biodegradación. Más importante aún, varios SMC poseían niveles equivalentes de eficiencia queratinolítica en comparación con el consorcio inicial, lo que demuestra que se puede lograr la simplificación sin pérdida de función y eficiencia. [22] el flujo de trabajo para este estudio incluyó cuatro pasos: (1) Enriquecimiento para los rasgos deseados, por ejemplo, actividad queratinolítica por selección en medio de queratina, donde la queratina es la única fuente de carbono. Este proceso se evaluó mediante evaluaciones funcionales (densidad celular, actividad enzimática y proporción del sustrato residual) y análisis de composición. (2) Se realizaron diluciones en serie para los consorcios microbianos eficaces enriquecidos. Se prepararon seis diluciones, desde la dilución 10 2 hasta 10 10 con 24 repeticiones. La diferencia entre las diluciones se evaluó mediante el cálculo de la distancia euclidiana según los criterios de evaluación funcional. (3) La construcción de la biblioteca se realizó a partir de la dilución que ofrece la disimilitud óptima entre las réplicas. Dilución 10 9 fue seleccionada para construir la biblioteca en este caso. (4) La selección más prometedora se basa en la caracterización funcional y composicional. [22]
Salud humana
Los consorcios se encuentran comúnmente en humanos, siendo los ejemplos predominantes el consorcio de la piel y el consorcio intestinal que brindan protección y ayuda en la nutrición humana. Además, se ha identificado que las bacterias existen dentro del cerebro (anteriormente se creía que eran estériles), con evidencia metagenómica que sugiere que las especies encontradas pueden ser de origen entérico. [33][34] Como las especies encontradas parecen estar bien establecidas, no tienen un impacto discernible en la salud humana y se sabe que las especies forman consorcios cuando se encuentran en el intestino, es muy probable que también hayan formado un consorcio simbiótico dentro del cerebro. [35]
Consorcios microbianos sintéticos
.tif.jpg.webp)
Los consorcios microbianos sintéticos (comúnmente llamados cocultivos) son sistemas de múltiples poblaciones que pueden contener una amplia gama de especies microbianas y son ajustables para servir una variedad de intereses industriales y ecológicos. Para la biología sintética, los consorcios llevan la capacidad de diseñar nuevos comportamientos celulares a un nivel de población. Los consorcios son más comunes que los que no lo son en la naturaleza y, en general, resultan más robustos que los monocultivos. [36] Hasta la fecha, se han cultivado e identificado poco más de 7.000 especies de bacterias. Muchas de las aproximadamente 1,2 millones de especies de bacterias que quedan aún no se han cultivado e identificado, en parte debido a la imposibilidad de cultivarlas axénicamente.[37] Al diseñar consorcios sintéticos o editar consorcios naturales, los biólogos sintéticos realizan un seguimiento del pH, la temperatura, los perfiles metabólicos iniciales, los tiempos de incubación, la tasa de crecimiento y otras variables pertinentes.[36]
Véase también
- Biofilm
- Inteligencia microbiana
- Bucle microbiano
- Estera microbiana
- Biología de población microbiana
- Sintético microbiano consortia
- Cooperación microbiana
- Hipótesis del rey rojo
- Interacciones biológicas
- Sucesión ecológica
Referencias
- OMS,OPS (ed.). «Microbiota». Descriptores en Ciencias de la Salud, Biblioteca virtual de Salud.
- Madigan, M; Bender, K; Buckley, D; Sattley, W; Stahl, D. (2019). Brock biology of microorganisms (Fifteenth, Global edición). New York, NY: Pearson. p. 173. ISBN 9781292235103.
- Mark, Martin (27 de abril de 2009). «Happy Together… Life of the Bacterial Consortium Chlorochromatium aggregatum». Small Things Considered - The Microbe Blog. American Society for Microbiology. Archivado desde el original el 1 de mayo de 2009. Consultado el 11 de enero de 2012. «Consortia are assemblages of different species of microbes in physical (and sometimes intricate biochemical) contact with one another, and are implicated in biological processes ranging from sewage treatment to marine nitrogen cycling to metabolic processes within the rumen. »
- Thompson, William Irwin (1991). Gaia 2 : emergence : the new science of becoming. Hudson, NY: Lindisfarne Press. pp. 51-58. ISBN 9780940262409.
- Reinke, Johannes 1872. Ueber die anatomischen Verhältnisse einiger Arten von Gunnera L. Nachrichten von der Königl. Gesellschaft der Wissenschaften und der Georg-Augusts-Universität zu Göttingen 9: 100–108.
- Kull, Kalevi 2010. Ecosystems are made of semiosic bonds: Consortia, umwelten, biophony and ecological codes. Biosemiotics 3(3): 347–357.
- Delaux, Pierre-Marc; Radhakrishnan, Guru V.; Jayaraman, Dhileepkumar; Cheema, Jitender; Malbreil, Mathilde; Volkening, Jeremy D.; Sekimoto, Hiroyuki; Nishiyama, Tomoaki et al. (27 de octubre de 2015). «Algal ancestor of land plants was preadapted for symbiosis». Proceedings of the National Academy of Sciences of the United States of America 112 (43): 13390-13395. Bibcode:2015PNAS..11213390D. PMC 4629359. PMID 26438870. doi:10.1073/pnas.1515426112.
- Subashchandrabose, Suresh R.; Ramakrishnan, Balasubramanian; Megharaj, Mallavarapu; Venkateswarlu, Kadiyala; Naidu, Ravi (2011). «Consortia of cyanobacteria/Microalgae and bacteria: Biotechnological potential». Biotechnology Advances 29 (6): 896-907. PMID 21801829. doi:10.1016/j.biotechadv.2011.07.009.
- Shong, Jasmine; Jimenez Diaz, Manuel Rafael; Collins, Cynthia H. (2012). «Towards synthetic microbial consortia for bioprocessing». Current Opinion in Biotechnology 23 (5): 798-802. PMID 22387100. doi:10.1016/j.copbio.2012.02.001.
- Brown, Margaret E.; Chang, Michelle CY (2014). «Exploring bacterial lignin degradation». Current Opinion in Chemical Biology 19: 1-7. PMID 24780273. doi:10.1016/j.cbpa.2013.11.015.
- Cregut, Mickael; Bedas, M.; Durand, M.-J.; Thouand, G. (2013). «New insights into polyurethane biodegradation and realistic prospects for the development of a sustainable waste recycling process». Biotechnology Advances 31 (8): 1634-1647. PMID 23978675. doi:10.1016/j.biotechadv.2013.08.011.
- Mikesková, H.; Novotný, Č.; Svobodová, K. (2012). «Interspecific interactions in mixed microbial cultures in a biodegradation perspectiva». Applied Microbiology and Biotechnology 95 (4): 861-870. PMID 22733114. doi:10.1007/s00253-012-4234-6.
- Skariyachan, Sinosh; Patil, Amulya A.; Shankar, Apoorva; Manjunath, Meghna; Bachappanavar, Nikhil; Kiran, S. (2018). «Enhanced polymer degradation of polyethylene and polypropylene by novel thermophilic consortia of Brevibacillus SPS. And Aneurinibacillus sp. Screened from waste management landfills and sewage treatment plants». Polymer Degradation and Stability 149: 52-68. doi:10.1016/j.polymdegradstab.2018.01.018.
- Skariyachan, Sinosh; Setlur, Anagha Shamsundar; Naik, Sujay Yashwant; Naik, Ashwini Amaresh; Usharani, Makam; Vasist, Kiran S. (2017). «Enhanced biodegradation of low and high-density polyethylene by novel bacterial consortia formulated from plastic-contaminated cow dung under thermophilic conditions». Environmental Science and Pollution Research 24 (9): 8443-8457. PMID 28188552. doi:10.1007/s11356-017-8537-0.
- Luo, Fei; Devine, Cheryl E.; Edwards, Elizabeth A. (2016). «Cultivating microbial dark matter in benzene-degrading methanogenic consortia». Environmental Microbiology 18 (9): 2923-2936. PMID 26549712. doi:10.1111/1462-2920.13121.
- Burniol-Figols, Anna; Varrone, Cristiano; Le, Simone Balzer; Daugaard, Anders Egede; Skiadas, Ioannis V.; Gavala, Hariklia N. (2018). «Combined polyhydroxyalkanoates (PHA) and 1,3-propanediol production from crude glycerol: Selective conversion of volatile fatty acids into PHA by mixed microbial consortia». Water Research 136: 180-191. PMID 29505919. doi:10.1016/j.watres.2018.02.029.
- Kang, Dingrong; Herschend, Jakob; Al-Soud, Waleed Abu; Mortensen, Martin Steen; Gonzalo, Milena; Jacquiod, Samuel; Sørensen, Søren J. (2018). «Enrichment and characterization of an environmental microbial consortium displaying efficient keratinolytic activity». Bioresource Technology 270: 303-310. PMID 30236907. doi:10.1016/j.biortech.2018.09.006.
- Lazuka, Adèle; Auer, Lucas; o'Donohue, Michael; Hernandez-Raquet, Guillermina (2018). «Anaerobic lignocellulolytic microbial consortium derived from termite gut: Enrichment, lignocellulose degradation and community dynamics». Biotechnology for Biofuels 11: 284. PMC 6191919. PMID 30356893. doi:10.1186/s13068-018-1282-x.
- Shade, Ashley; Peter, Hannes; Allison, Steven D.; Baho, Didier L.; Berga, Mercè; Bürgmann, Helmut; Huber, David H.; Langenheder, Silke et al. (2012). «Fundamentals of Microbial Community Resistance and Resilience». Frontiers in Microbiology 3: 417. PMC 3525951. PMID 23267351. doi:10.3389/fmicb.2012.00417.
- Awasthi, Ashutosh; Singh, Mangal; Soni, Sumit K.; Singh, Rakshapal; Kalra, Alok (2014). «Biodiversity acts as insurance of productivity of bacterial communities under abiotic perturbations». The ISME Journal 8 (12): 2445-2452. PMC 4260711. PMID 24926862. doi:10.1038/ismej.2014.91.
- Banerjee, Samiran; Kirkby, Clive A.; Schmutter, Dione; Bissett, Andrew; Kirkegaard, John A.; Richardson, Alan E. (2016). «Network analysis reveals functional redundancy and keystone taxa amongst bacterial and fungal communities during organic matter decomposition in an arable soil». Soil Biology and Biochemistry 97: 188-198. doi:10.1016/j.soilbio.2016.03.017.
- Kang, Dingrong; Jacquiod, Samuel; Herschend, Jakob; Wei, Shaodong; Nesme, Joseph; Sørensen, Søren J. (2020). «Construction of Simplified Microbial Consortia to Degrade Recalcitrant Materials Based on Enrichment and Dilution-to-Extinction Cultures». Frontiers in Microbiology 10: 3010. PMC 6968696. PMID 31998278. doi:10.3389/fmicb.2019.03010.
 Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License.
Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License. - Puentes-Téllez, Pilar Eliana; Falcao Salles, Joana (2018). «Construction of Effective Minimal Active Microbial Consortia for Lignocellulose Degradation». Microbial Ecology 76 (2): 419-429. PMC 6061470. PMID 29392382. doi:10.1007/s00248-017-1141-5.
- Jung, Jaejoon; Philippot, Laurent; Park, Woojun (2016). «Metagenomic and functional analyses of the consequences of reduction of bacterial diversity on soil functions and bioremediation in diesel-contaminated microcosms». Scientific Reports 6: 23012. Bibcode:2016NatSR...623012J. PMC 4789748. PMID 26972977. doi:10.1038/srep23012.
- Lee, Duu-Jong; Show, Kuan-Yeow; Wang, Aijie (2013). «Unconventional approaches to isolation and enrichment of functional microbial consortium – A review». Bioresource Technology 136: 697-706. PMID 23566469. doi:10.1016/j.biortech.2013.02.075.
- Ho, Kuo-Ling; Lee, Duu-Jong; Su, Ay; Chang, Jo-Shu (2012). «Biohydrogen from lignocellulosic feedstock via one-step process». International Journal of Hydrogen Energy 37 (20): 15569-15574. doi:10.1016/j.ijhydene.2012.01.137.
- Hoefman, Sven; Van Der Ha, David; De Vos, Paul; Boon, Nico; Heylen, Kim (2012). «Miniaturized extinction culturing is the preferred strategy for rapid isolation of fast-growing methane-oxidizing bacteria». Microbial Biotechnology 5 (3): 368-378. PMC 3821679. PMID 22070783. doi:10.1111/j.1751-7915.2011.00314.x.
- Sosa, Oscar A.; Gifford, Scott M.; Repeta, Daniel J.; Delong, Edward F. (2015). «High molecular weight dissolved organic matter enrichment selects for methylotrophs in dilution to extinction cultures». The ISME Journal 9 (12): 2725-2739. PMC 4817625. PMID 25978545. doi:10.1038/ismej.2015.68.
- Roger, Fabian; Bertilsson, Stefan; Langenheder, Silke; Osman, Omneya Ahmed; Gamfeldt, Lars (2016). «Effects of multiple dimensions of bacterial diversity on functioning, stability and multifunctionality». Ecology 97 (10): 2716-2728. PMID 27859115. doi:10.1002/ecy.1518.
- Santoyo, Gustavo; Guzmán-Guzmán, Paulina; Parra-Cota, Fannie Isela; Santos-Villalobos, Sergio de los; Orozco-Mosqueda, Ma. del Carmen; Glick, Bernard R. (2021). «Plant Growth Stimulation by Microbial Consortia». Agronomy 11 (2): 219. doi:10.3390/agronomy11020219.
 Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License.
Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License. - Coulombe, Pierre A.; Omary, M.Bishr (2002). «'Hard' and 'soft' principles defining the structure, function and regulation of keratin intermediate filaments». Current Opinion in Cell Biology 14 (1): 110-122. PMID 11792552. doi:10.1016/s0955-0674(01)00301-5.
- Korniłłowicz-Kowalska, Teresa; Bohacz, Justyna (2011). «Biodegradation of keratin waste: Theory and practical aspects». Waste Management 31 (8): 1689-1701. PMID 21550224. doi:10.1016/j.wasman.2011.03.024.
- Pennisi, Elizabeth (7 de mayo de 2020). «Meet the 'psychobiome': the gut bacteria that may alter how you think, feel, and act». Science Magazine. Consultado el 12 de diciembre de 2020.
- Rettner, Rachel (15 de noviembre de 2018). «Bacteria May Live (Harmlessly) in Your Brain» (en inglés). Live Science. Consultado el 12 de diciembre de 2020.
- Roberts, R. C.; Farmer, C. B.; Walker, C. K. (6 de noviembre de 2018). «The human brain microbiome; there are bacteria in our brains!». Psychiatry and Behavioral Neurobio., Univ. Of Alabama, Birmingham, Birmingham, AL. 2018 Neuroscience Meeting Planner. (Program No. 594.08). Consultado el 12 de diciembre de 2020.
- Hays, Stephanie G.; Ducat, Daniel C. (14 de febrero de 2014). «Engineering cyanobacteria as photosynthetic feedstock factories». Photosynthesis Research 123 (3): 285-295. PMC 5851442. PMID 24526260. doi:10.1007/s11120-014-9980-0.
- Stewart, Eric J. (15 de agosto de 2012). «Growing Unculturable Bacteria». Journal of Bacteriology (en inglés) 194 (16): 4151-4160. PMC 3416243. PMID 22661685. doi:10.1128/JB.00345-12.
Enlaces externos
- Esta obra contiene una traducción total derivada de «Microbial consortium» de Wikipedia en inglés, concretamente de esta versión, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.