Epigenoma
El epigenoma en el campo de la epigenética, es un término derivado del prefijo epi, que significa "por encima o sobre" el genoma.[1] El epigenoma es un conjunto de compuestos químicos en contacto con el ADN, que modifican, o marcan, el genoma de manera que le dice qué hacer, dónde hacerlo y cuándo hacerlo.[2] Células diferentes tienen diferentes marcas epigenéticas. Estas marcas epigenéticas, que no forman parte del propio ADN, pueden ser transmitidas de una célula a otra durante la división celular, y de una generación a la otra.[3]

compuesto por: ADN (hebra naranja) + proteína histona (cilindro azul) + elementos externos (esferas verdes).
El epigenoma consiste en un registro de los cambios químicos en el ADN y las proteínas nucleares histonas de un organismo; estos cambios pueden transmitirse a la descendencia de un organismo, a través de la Herencia epigenética transgeneracional varada. Los cambios en el epigenoma pueden provocar cambios en la estructura de la cromatina y por tanto cambios en la función del genoma.[4]
El epigenoma participa en la regulación de la expresión génica, el desarrollo, la diferenciación de tejidos y la supresión de elementos transponibles. A diferencia del genoma subyacente, que permanece en gran parte estático dentro de un individuo, el epigenoma puede ser alterado dinámicamente por las condiciones ambientales.[5]
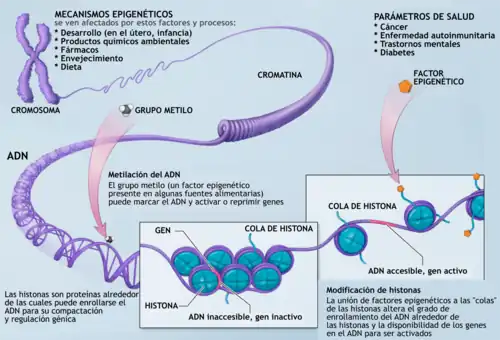
Cambios en el epigenoma
La regulación de la expresión de los genes, implica mecanismos de múltiples capas en los que las modificaciones epigenéticas, como la metilación del ADN y las modificaciones de la cola de las histonas, desempeñan un papel importante.
Las modificaciones postraduccionales de histonas en algunos de sus aminoácidos específicos, ayudan a determinar la estructura de la cromatina y, por tanto, la accesibilidad a los promotores de genes y las regiones reguladoras.[6]
Efectos de cambios en el epigenoma
Los cambios en el epigenoma, (los factores que son determinados por el ambiente celular y no por los genes), provocan cambios en la estructura de la cromatina y por tanto cambios en la función del genoma.[4] [7]
Las cosas que le pasan a alguien a lo largo de su vida pueden cambiar la forma en que se expresa su ADN, y ese cambio puede transmitirse a la próxima generación.[8][9]
Las exposiciones ambientales durante el desarrollo fetal y postnatal temprano, pueden conducir a una mayor incidencia de enfermedades de aparición tardía en la edad adulta. Esos factores ambientales incluyen: anomalías nutricionales, estrés y exposición a sustancias tóxicas.[10][9]
La exposición del feto a bisfenol A conduce a anormalidades inmunes. El tabaquismo materno conduce a un aumento de la enfermedad pulmonar en la edad adulta. Los defectos de nutrición conducen a la hipertensión en la descendencia y la exposición a drogas terapéuticas como la dexametasona conduce a defectos vasculares.
En la edad adulta se ha demostrado que los factores ambientales afectan a la próxima generación F2.[8] Los efectos sobre la generación F1 y F2 pueden deberse a la exposición multigeneracional directa al factor ambiental. Si una hembra gestante se define como la generación fundadora F0, entonces la descendencia fetal es la generación F1, y las células germinales presentes en esos fetos en desarrollo eventualmente se convertirán en los óvulos o espermatozoides que formarían la generación F2.[11]
Cáncer
La epigenética es un tema actualmente activo en la investigación del cáncer. Los tumores humanos sufren una alteración importante de los patrones de modificación de histonas y metilación del ADN. El paisaje epigenético aberrante de la célula cancerosa se caracteriza por una hipometilación genómica global, hipermetilación del promotor de isla CpG de genes supresores de tumores, un código de histona alterado para genes críticos y una pérdida global de histona H4 monoacetilada y trimetilada.
Proyectos de investigación de epigenomas
Como preludio de un posible Proyecto de Epigenoma Humano, el Proyecto Piloto de Epigenoma Humano tiene como objetivo identificar y catalogar las Posiciones Variables de Metilación (MVP) en el genoma humano.[12] Los avances en la tecnología de secuenciación ahora permiten analizar estados epigenómicos de todo el genoma mediante múltiples metodologías moleculares.[13] Se han construido o propuesto dispositivos a micro y nanoescala para investigar el epigenoma.[14]
Un esfuerzo internacional para analizar epigenomas de referencia comenzó en 2010 en forma del Consorcio Internacional de Epigenomas Humanos (IHEC).[15][16][17] Los miembros de IHEC tienen como objetivo generar al menos 1000 epigenomas humanos de referencia (línea de base) a partir de diferentes tipos de células humanas normales y relacionadas con enfermedades.[18][19]
Roadmap Epigenomics Project
Uno de los objetivos del Roadmap Epigenomics Project Archivado el 8 de abril de 2021 en Wayback Machine. de NIH es generar epigenomas humanos de referencia a partir de individuos normales y sanos a través de una gran variedad de líneas celulares, células primarias y tejidos primarios. Los datos producidos por el proyecto, que se pueden buscar y descargar del Atlas del Epigenoma Humano, se dividen en cinco tipos que analizan diferentes aspectos del epigenoma y los resultados de los estados epigenómicos (como la expresión génica):
- Modificaciones de histonas: la secuenciación de inmunoprecipitación de cromatina (ChIP-Seq) identifica patrones de modificaciones de histonas en todo el genoma utilizando anticuerpos contra las modificaciones.[20]
- Metilación del ADN: secuencia de bisulfito de genoma completo, secuencia de bisulfito de representación reducida (RRBS), secuenciación de inmunoprecipitación de ADN metilado (MeDIP-Seq) y secuenciación de enzimas de restricción sensibles a la metilación (MRE-Seq) identifican la metilación del ADN en porciones del genoma en diferentes niveles de resolución hasta el nivel de pares de bases.[21]
- Accesibilidad a la cromatina: sitios hipersensibles a la DNasa I La secuenciación (DNase-Seq) utiliza la enzima DNasa I para encontrar regiones abiertas o accesibles en el genoma.
- Expresión génica: las matrices de expresión y RNA-Seq identifican niveles de expresión o genes que codifican proteínas.
- Expresión de ARN pequeño: smRNA-Seq identifica la expresión de ARN no codificante pequeño, principalmente micro ARN.
Los epigenomas de referencia para individuos sanos permitirán el segundo objetivo del Roadmap Epigenomics Project, que es examinar las diferencias epigenómicas que ocurren en estados patológicos como la enfermedad de Alzheimer.
Véase también
Referencias
- «Epi». Wiktionary.
- OMS, OPS (ed.). «Epigenoma». Descriptores en Ciencias de la Salud, Biblioteca Virtual de Salud.
- «Epigenoma | NHGRI». Genome.gov. Consultado el 18 de octubre de 2021.
- Bernstein, Bradley E.; Meissner, Alexander; Lander, Eric S. (23 de febrero de 2007). «The mammalian epigenome». Cell 128 (4): 669-681. ISSN 0092-8674. PMID 17320505. doi:10.1016/j.cell.2007.01.033.
- Laura Elnitski. «Epigenoma». NIH. Consultado el 8 de abril de 2021.S.}}
- Figueroa M.E., Reimers M., Thompson R.F., Ye K., Li Y., Selzer R.R., Fridriksson J., Paietta E., Wiernik P., Green R.D., Greally J.M. (2008). «An Integrative Genomic and Epigenomic Approach for the Study of Transcriptional Regulation». PLoS ONE 3 (3): e1882. doi:10.1371/journal.pone.0001882.
- «Epigenómica». National Human Genome Research Institute. NIH. 2019. Consultado el 8 de abril de 2021.
- Martha Henriques (4 de mayo de 2019). «Qué es la epigenética y cómo explica que los hijos hereden los traumas de los padres». BBC-Future. Consultado el 8 de abril de 2021.
- Casavilca-Zambrano S., Cancino-Maldonado K., Jaramillo-Valverde L., Guio H. (2019). «Epigenética: la relación del medio ambiente con el genoma y su influencia en la salud mental.». Rev Neuropsiquiatr (Artículo de revisión) (Lima, Perú: SciELO) 82 (4). Consultado el 8 de abril de 2021.
- Environmental epigenomics and disease susceptibility (2007). «Jirtle R.L., Skinner M.K.». Review Nat Rev Genet (en inglés) 8 (4): 253-262. doi:10.1038/nrg2045. Consultado el 8 de abril de 2021.
- Nilsson E., Larsen G., Manikkam M., Guerrero-Bosagna C., Savenkova M.I., Skinner M.K. (2012). «Environmentally Induced Epigenetic Transgenerational Inheritance of Ovarian Disease». PLoS ONE (en inglés) 7 (5): e36129. doi:10.1371/journal.pone.0036129. Consultado el 8 de abril de 2021.
- «Human Epigenome Project». Archivado desde el original el 16 de julio de 2011. Consultado el 29 de junio de 2011.
- Milosavljevic, Aleksandar (June 2011). «Emerging patterns of epigenomic variation». Trends in Genetics 27 (6): 242-250. PMC 3104125. PMID 21507501. doi:10.1016/j.tig.2011.03.001.
- Aguilar, Carlos; Craighead, Harold (4 de octubre de 2013). «Micro- and nanoscale devices for the investigation of epigenetics and chromatin dynamics». Nature Nanotechnology 8 (10): 709-718. Bibcode:2013NatNa...8..709A. PMC 4072028. PMID 24091454. doi:10.1038/nnano.2013.195.
- «Time for the epigenome». Nature 463 (7281): 587. 4 de febrero de 2010. ISSN 1476-4687. PMID 20130607. doi:10.1038/463587a.
- Abbott, Alison (4 de febrero de 2010). «Project set to map marks on genome». Nature 463 (7281): 596-597. ISSN 1476-4687. PMID 20162836.
- Bae, JB (2013). «Perspectives of international human epigenome consortium». Genomics Inform 11 (1): 7-14. PMC 3630389. PMID 23613677. doi:10.5808/GI.2013.11.1.7.
- Eurice GmbH. "About IHEC".
- Kanai, Yae; Arai, Eri (2014). «Multilayer-omics analyses of human cancers: Exploration of biomarkers and drug targets based on the activities of the International Human Epigenome Consortium». Frontiers in Genetics 5: 24. PMC 3924033. PMID 24592273. doi:10.3389/fgene.2014.00024.
- Zhu, J. (2013). «Genome-wide chromatin state transitions associated with developmental and environmental cues». Cell 152 (3): 642-654. PMC 3563935. PMID 23333102. doi:10.1016/j.cell.2012.12.033.
- Harris, R Alan; Wang, Ting; Coarfa, Cristian; Nagarajan, Raman P; Hong, Chibo; Downey, Sara L et al. (19 de septiembre de 2010). «Comparison of sequencing-based methods to profile DNA methylation and identification of monoallelic epigenetic modifications». Nature Biotechnology 28 (10): 1097-1105. PMC 2955169. PMID 20852635. doi:10.1038/nbt.1682.
Enlaces externos
- Página de inicio del Consorcio de Mapeo de Epigenomas de Referencia
- Centro de epigenómica NCBI
- Epigenómica ómnibus de expresión génica de NCBI
- Atlas del epigenoma humano
- Roadmap Epigenomics Visualization Hub
- Roadmap Epigenomics Visualization Hub (centro de seguimiento de carga)
- Navegador de epigenoma humano en la Universidad de Washington
- Espejo UCSC del navegador Epigenome Archivado el 14 de febrero de 2021 en Wayback Machine.
- Proyecto Epigenoma Humano
- Investigación sobre el cáncer