Hidroximetilglutaril-CoA sintasa
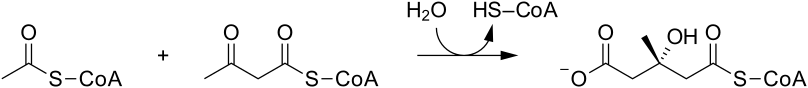
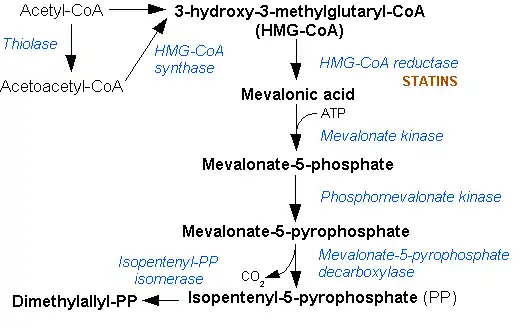
En biología molecular, la Hidroximetilglutaril-CoA sintasa o abreviadamente HMG-CoA sintasa (EC 2.3.3.10) es una enzima que cataliza la reacción en la que el acetil-CoA se condensa con el acetoacetil-CoA para formar 3-hidroxi-3-metilglutaril-CoA (HMG-CoA). Es la segunda reacción en la ruta de biosíntesis de isoprenoides dependiente de mevalonato. El HMG-CoA es un intermedio en la síntesis de colesterol y, por tanto, de la cetogénesis. Esta reacción es más activa en pacientes con diabetes mellitus tipo 1, si no se trata, debido a la deficiencia prolongada de la insulina y el agotamiento de los sustratos para la gluconeogénesis y el ciclo TCA, especialmente el oxalacetato. Esto resulta en la derivación del exceso de acetil-CoA por la vía de síntesis de cuerpos cetónicos a través de la HMG-CoA, que conduce al desarrollo de la cetoacidosis diabética.
| 3-hidroxi-3-metilglutaril-Coenzima A sintasa 1 (soluble) | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Símbolos | HMGCS1 (HGNC: 5007) HMGCS | |||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 2.3.3.10 | |||
| Locus | Cr. 5 p14-p13 | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
| 3-hidroxi-3-metilglutaril-Coenzima A sintasa 1 (mitocondrial) | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Símbolo | HMGCS2 (HGNC: 5008) | |||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 2.3.3.10 | |||
| Locus | Cr. 1 p13-p12 | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
| Hidroximetilglutaril-CoA sintasa | ||
|---|---|---|
 | ||
| Identificadores | ||
| Símbolo | HMG_CoA_synt_N | |
| Pfam | PF01154 | |
| InterPro | IPR013528 | |
| PROSITE | PDOC00942 | |
| Hidroximetilglutaril-CoA sintasa | ||
|---|---|---|
 3-hidroxi-3-metilglutaril-coa sintasa de Staphylococcus aureus | ||
| Identificadores | ||
| Símbolo | HMG_CoA_synt_C | |
| Pfam | PF08540 | |
| InterPro | IPR013746 | |
| PROSITE | PDOC00942 | |
Mecanismo
La HMG-CoA sintasa contiene un importante residuo catalítico de cisteína que actúa como un nucleófilo en la primera etapa de la reacción: la acetilación de la enzima por la acetil-CoA (su primer sustrato) para producir un tioéster acetil-enzima, liberando la coenzima A reducida. El ataque nucleofílico posterior sobre la acetoacetil-CoA (su segundo sustrato) conduce a la formación de la HMG-CoA.[1]
Localización en diferentes especies
Esta reacción es parte de varias rutas metabólicas en eucariotas, archaea y algunas bacterias.[2]
Organismos eucariotas
En los vertebrados, hay dos isoenzimas diferentes (citosólica y mitocondrial); en los seres humanos la forma citosólica sólo tiene una identidad de aminoácidos del 60,6% con la forma mitocondrial de la enzima. La HMG-CoA también se encuentra en otros eucariotas tales como insectos, plantas y hongos.[3]
HMG-CoA sintasa citosólica
La HMG-CoA sintasa citosólica (HMGCS1) es el punto de partida de la ruta del mevalonato, que lleva al colesterol y otros compuestos esterólicos e isoprenoides.
HMG-CoA sintasa mitocondrial
La isoenzima mitocondrial (HMGCS2) se localiza en el hígado y el riñón humano y es el único responsable de la biosíntesis de los cuerpos cetónicos. El gen para la forma mitocondrial de la enzima tiene tres elementos reguladores de esteroles en la región flanqueante 5 '[4] Estos elementos son responsables de la disminución de la transcripción del ARN mensajero responsable de la síntesis de la enzima cuando el nivel de colesterol de la dieta es alta en los animales. Las mutaciones en el gen HMGCS2 pueden conducir a una hipoglucemia severa.[5]
Organismos procariotas
En las bacterias, los precursores isoprenoides se sintetizan generalmente a través de una ruta alternativa, la ruta no-mevalonato, sin embargo, un número de patógenos Gram-positivos utilizan una vía del mevalonato que implica la HMG-CoA sintasa que es paralela a la que se encuentra en eucariotas.[6][7]
Referencias
- Theisen MJ, Misra I, Saadat D, Campobasso N, Miziorko HM, Harrison DH (noviembre de 2004). «3-hydroxy-3-methylglutaryl-CoA synthase intermediate complex observed in "real-time"». Proc. Natl. Acad. Sci. U.S.A. 101 (47): 16442-7. PMC 534525. PMID 15498869. doi:10.1073/pnas.0405809101.
- Bahnson BJ (noviembre de 2004). «An atomic-resolution mechanism of 3-hydroxy-3-methylglutaryl-CoA synthase». Proc. Natl. Acad. Sci. U.S.A. 101 (47): 16399-400. PMC 534547. PMID 15546978. doi:10.1073/pnas.0407418101.
- Bearfield JC, Keeling CI, Young S, Blomquist GJ, Tittiger C (abril de 2006). «Isolation, endocrine regulation and mRNA distribution of the 3-hydroxy-3-methylglutaryl coenzyme A synthase (HMG-S) gene from the pine engraver, Ips pini (Coleoptera: Scolytidae)». Insect Mol. Biol. 15 (2): 187-95. PMID 16640729. doi:10.1111/j.1365-2583.2006.00627.x.
- Goldstein J.L., Brown M.S. (1990) Regulation of the mevalonate pathway. Nature 343, 425-430
- Orphanet: 3-hydroxy 3-methylglutaryl-CoA (HMG) synthase deficiency
- Steussy CN, Robison AD, Tetrick AM, Knight JT, Rodwell VW, Stauffacher CV, Sutherlin AL (diciembre de 2006). «A structural limitation on enzyme activity: the case of HMG-CoA synthase». Biochemistry 45 (48): 14407-14. PMID 17128980. doi:10.1021/bi061505q.
- Steussy CN, Vartia AA, Burgner JW, Sutherlin A, Rodwell VW, Stauffacher CV (noviembre de 2005). «X-ray crystal structures of HMG-CoA synthase from Enterococcus faecalis and a complex with its second substrate/inhibitor acetoacetyl-CoA». Biochemistry 44 (43): 14256-67. PMID 16245942. doi:10.1021/bi051487x.