Inserción (genética)
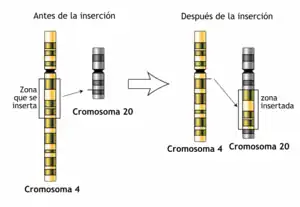
En genética, una inserción (también llamada mutación de inserción) es la adición de uno o más pares de bases de nucleótidos en una secuencia de ADN. Esto a menudo puede suceder en regiones de microsatélites debido al deslizamiento de la ADN polimerasa. Las inserciones pueden tener cualquier tamaño, desde un par de bases insertado incorrectamente en una secuencia de ADN hasta una sección de un cromosoma insertado en otro. Se cree que el mecanismo de las mutaciones de inserción de base única más pequeñas es a través de la separación de pares de bases entre las cadenas de plantilla y cebador, seguido por el apilamiento de bases no vecinas, que puede ocurrir localmente dentro del sitio activo de la ADN polimerasa.[1] A nivel cromosómico, una inserción se refiere a la inserción de una secuencia más grande en un cromosoma. Esto puede suceder debido a un cruce desigual durante la meiosis.
La adición de la región N es la adición de nucleótidos no codificados durante la recombinación por la desoxinucleotidil transferasa terminal.
La inserción de nucleótidos P es la inserción de secuencias palindrómicas codificadas por los extremos de los segmentos de genes recombinantes.
Las repeticiones de trinucleótidos se clasifican como mutaciones de inserción[2][3] y, a veces, como una clase separada de mutaciones.[4]
Efectos
Las inserciones pueden ser particularmente peligrosas si se producen en un exón, la región de codificación de aminoácidos de un gen. Una mutación de desplazamiento de lectura, una alteración en el marco de lectura normal de un gen, resulta si el número de nucleótidos insertados no es divisible por tres, es decir, el número de nucleótidos por codón. Las mutaciones de cambio de marco alterarán todos los aminoácidos codificados por el gen después de la mutación. Por lo general, las inserciones y la posterior mutación de desplazamiento de cuadros provocarán que la traducción activa del gen encuentre un codón de parada prematuro, lo que dará como resultado el final de la traducción y la producción de una proteína truncada. Las transcripciones que portan la mutación de desplazamiento de cuadros también pueden degradarse a través de la descomposición mediada sin sentido durante la traducción, lo que no da como resultado ningún producto proteico. Si se traducen, las proteínas truncadas con frecuencia son incapaces de funcionar correctamente o en absoluto, y posiblemente pueden dar lugar a cualquier cantidad de trastornos genéticos dependiendo del gen en el que ocurre la inserción. Se desarrollaron métodos para detectar errores de secuenciación de ADN.[5]
Las inserciones dentro del marco ocurren cuando el marco de lectura no se altera como resultado de la inserción; el número de nucleótidos insertados es divisible por tres. El marco de lectura permanece intacto después de la inserción y la traducción probablemente se completará si los nucleótidos insertados no codifican un codón de parada. Sin embargo, debido a los nucleótidos insertados, la proteína terminada contendrá, dependiendo del tamaño de la inserción, múltiples aminoácidos nuevos que pueden afectar la función de la proteína.
Véase también
- Indel
- Mutagénesis por inserción
- Mutaciones por pérdida de función
- Mutaciones de ganancia de función
Referencias
- «Mechanisms: Genetic Variation: Types of Mutations». Evolution 101: Understanding Evolution For Teachers. University of California Museum of Paleontology. Archivado desde el original el 14 de abril de 2009. Consultado el 19 de septiembre de 2009. ] Understanding Evolution For Teachers Home. Retrieved on September 19, 2009
- Brown, Terence A. (2007). «16 Mutations and DNA Repair». Genomes 3. Garland Science. p. 510. ISBN 978-0-8153-4138-3.
- Faraone, Stephen V.; Tsuang, Ming T.; Tsuang, Debby W. (1999). «5 Molecular Genetics and Mental Illness: The Search for Disease Mechanisms: Types of Mutations». Genetics of Mental Disorders: A Guide for Students, Clinicians, and Researchers. Guilford Press. p. 145. ISBN 978-1-57230-479-6.
- Shmilovici, Armin; Ben-Gal, Irad (1 de abril de 2007). «Using a VOM model for reconstructing potential coding regions in EST sequences». Computational Statistics (en inglés) 22 (1): 49-69. ISSN 1613-9658. doi:10.1007/s00180-007-0021-8.
Otras lecturas
- Pierce, Benjamin A. (2013). Genetics: A Conceptual Approach (5th edición). W. H. Freeman. ISBN 978-1-4641-5084-5.