Piruvato descarboxilasa
La piruvato descarboxilasa (EC 4.1.1.1) es una enzima homotetramérica que cataliza en el citoplasma la descarboxilación del ácido pirúvico a acetaldehído y dióxido de carbono.
| Piruvato descarboxilasa | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 4.1.1.1 | |||
| Número CAS | 9001-04-1 | |||
| Ortólogos | ||||
| Especies |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
A esta enzima también se le llama 2-oxoácido carboxilasa, α-carboxilasa, α-cetoácido carboxilasa y decarboxilasa pirúvica.[1] En condiciones anaerobias, esta enzima es parte del proceso de fermentación que produce etanol y que se produce en las levaduras, especialmente del género Saccharomyces.[2] La piruvato decarboxilasa depende de los cofactores tiamina pirofosfato (TPP) y magnesio. Esta enzima no debe ser confundida con la piruvato deshidrogenasa que cataliza la decarboxilación oxidativa del piruvato a acetil-CoA.
Función en las levaduras
En las levaduras, la piruvato decarboxilasa actúa independientemente durante la fermentación anaerobia y libera acetaldehído y dióxido de carbono. La piruvato decarboxilasa crea la forma de eliminación del CO2 que la célula libera. La enzima también crea etanol que es usado como antibiótico para eliminar organismos competidores.[3] La enzima es necesaria para ayudar en la decarboxilación de los α-cetoácidos ya que hay una acumulación de carga negativa que ocurre en el átomo de carbono carbonilo en el estado de transición proporcionando el ambiente adecuado para el la tiamina pirofosfato y el piruvato.[3]
Estructura
La piruvato decarboxilasa se presenta como un dímero de dímeros con dos sitios activos compartidos entre los monómeros de cada dímero. La enzima contiene una estructura beta-alfa-beta. Contiene 563 aminoácidos en cada dímero. La enzima tiene atracciones intermónomero fuertes, pero los dímeros interaccionan débilmente para formar el tetrámero.[3]
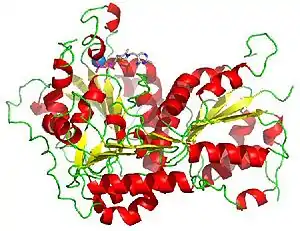
Residuos del sitio activo
La piruvato decarboxilasa es un homotetrámero y tiene cuatro sitios activos. Los sitios activos están dentro de una cavidad en el núcleo de la enzima en donde se pueden producir puentes de hidrógeno y en donde el piruvato reacciona con la tiamina pirofosfato (TPP). Cada sitio activo tiene 20 aminoácidos incluyendo el Glu-477 que contribuye a la estabilidad del anillo de la TPP, y el Glu-51 que ayuda en la unión del cofactor. Estos glutamatos también contribuyen a formar el dipolo neutro de la TPP, actuando como donante de protones al anillo aminopirimidina de la TPP. El microambiente alrededor de Glu-477 es muy no polar, contribuyendo a un pKa más alto de lo normal.[4]
Los residuos lipofílicos Ile-476, Ile-480 y Pro-26 contribuyen a la no polaridad del área alrededor de Glu-477. El otro residuo cargado negativamente separado de la coenzima TPP es el Asp-28, que también ayuda a incrementar el pKa de Glu-477. El medioambiente de la enzima debe permitir que la protonación del grupo γ-carboxilo del Glu-477 sea alrededor del pH 6.[4]
Una vez en su forma imina, el anillo aminopirimidina de la TPP actúa como una base para quitar el protón del carbono 2 de la TPP para formal el dipolo neutro nucleófilo.[3] Esto debe ocurrir porque la enzima no tiene cadenas laterales básicas para deprotononar el C2 de la TPP. Una mutación de estos glutamatos del sitio activo puede resultar en la ineficiencia o inactividad de la enzima. Esta inactividad se ha demostrado en experimentos en los que los grupos N1' y/o 4'-amino de la TPP no están. Mediante análisis NMR se ha determinado que cuando la TPP está unida a la enzima con el análogo del sustrato piruvamida, la velocidad de formación del dipolo neutro es superior que la velocidad normal de la enzima. También, la velocidad de mutación del Glu-51 a Gln reduce esta velocidad significativamente.[3]
También están incluidos en el sitio activo el Asp-444 y el Asp-28 que lo estabilizan. Estos actúan como estabilizadores para el ion Mg2+ que está presente en cada sitio activo. Para asegurar que solamente se une piruvato, los residuos Cys-221 e His-92 producen un cambio conformacional que inhibe o activa la enzima dependiendo del sustrato que interacciona con ella. Si el sustrato unido al sitio activo es piruvato, entonces la enzima es activada por un cambio conformacional en su sitio regulador.[5] El cambio conformacional supone una adición 1,2-nucleofílica. Esta reacción, la formación de un tiocetal, transforma la enzima desde su estado inactivo a su estado activo.
La inhibición del sitio activo es realizada por la clase XC6H4CH=CHCOCOOH de inhibidores/análogos de sustrato, así como por el producto de la decarboxilación de estos compuestos. Otros sitios potenciales nucleofílicos incluyen Cys-152, Asp-28. His-114, His-115 y Gln-477.[5]
La velocidad normal catalítica de la piruvato decarboxilasa es kcat = 10 s−1. La velocidad de la enzima con una mutación de Glu-51 a Gln es 1.7 s−1.[3]
Grupo prostético TPP
El cofactor tiamina pirofosfato TPP es necesario para este mecanismo de reacción, actúa como grupo prostético de la enzima. El átomo de carbono entre los átomos de azufre y nitrógeno en el anillo tiazol actúan como un carbanión que une al piruvato. La TPP tiene un H+ acídico en su carbono 2 que actúa como parte funcional del anillo tiazolium. Este anillo actúa como un sumidero de electrones, permitiendo a los electrones del carbanión ser estabilizados por resonancia.[2] La TPP entonces puede actuar como un nucleófilo con la pérdida del hidrógeno del C2, formando la forma dipolar neutra de la TPP. Esta forma puede atacar al piruvato que está sujetado por la enzima piruvato decarboxilasa. Durante la decarboxilación del piruvato, la TPP estabiliza los intermedios carbanión como un electrófilo por enlaces no covalentes.[3] Específicamente, el nitrógeno piridilo N1' y el grupo 4'-amino de la TPP son esenciales para la función catalítica del complejo enzima-TPP.[4]
Mecanismo
La enzima rompe al piruvato en dióxido de carbono y un fragmento de 2 carbonos que está unido al cofactor TPP por el anillo de cinco átomos en su forma de dipolo neutro.

Referencias
- «ENZYME entry: EC 4.1.1.1».
- Begley, Tadhg P.; McMurry, John (2005). Roberts and Co. Publishers, ed. The organic chemistry of biological pathways. pp. 179. ISBN 0-9747077-1-6. Consultado el 30 de octubre de 2011.
- PDB 1pyd ; Dyda F, Furey W, Swaminathan S, Sax M, Farrenkopf B, Jordan F (junio de 1993). «Catalytic centers in the thiamin diphosphate dependent enzyme pyruvate decarboxylase at 2.4-A resolution». Biochemistry 32 (24): 6165-70. PMID 8512926. doi:10.1021/bi00075a008.
- Lobell M, Crout DHG (1996). «Pyruvate Decarboxylase: A Molecular Modeling Study of Pyruvate Decarboxylation and Acyloin Formation». J. Am. Chem. Soc. 118 (8): 1867-1873. doi:10.1021/ja951830t.
- Baburina I, Dikdan G, Guo F, Tous GI, Root B, Jordan F (febrero de 1998). «Reactivity at the substrate activation site of yeast pyruvate decarboxylase: inhibition by distortion of domain interactions». Biochemistry 37 (5): 1245-55. PMID 9477950. doi:10.1021/bi9709912.