Preplasmiviricota
Preplasmiviricota es un filo de virus de ADN del dominio Varidnaviria y reino Bamfordvirae que incluye los virófagos, los adenovirus junto con algunas familias de virus procariotas. Estos últimos jugaron un papel en el desarrollo de la superfamilia de transposones de ADN polintones, los plásmidos mitocondriales y los plásmidos citoplasmáticos en los eucariotas a través de las infecciones virales, según los análisis filogenéticos de la ADN polimerasa. Contiene 8 familias virales y una esta en revisión.[1][2]
| Preplasmiviricota | ||
|---|---|---|
 | ||
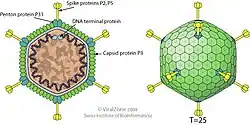 Viriones de un tectivirus y un adenovirus. | ||
| Taxonomía | ||
| Dominio: | Varidnaviria | |
| Reino: | Bamfordvirae | |
| Filo: | Preplasmiviricota | |
| Clasificación de Baltimore | ||
Este filo filogenéticamente es parafilético porque de virus pequeños de este conjunto se originarían los virus gigantes (filo Nucleocytoviricota), aumentando su genoma y el tamaño del virión mediante la duplicación y deleción de genes, la inclusión de elementos genéticos móviles y la adquisición masiva de genes del huésped y sus bacterias endosimbióticas, incluidos los genes para la traducción y los genes informáticos que se consideran los más resistentes a la transferencia horizontal.[3][4][5][6][7] Aunque sea un taxón parafilético puede emplearse para la taxonomía viral debido a que los taxones están emparentados entre sí y comparten caracteres únicos que los diferencia de los virus gigantes.
Taxonomía
La taxonomía establecida por el ICTV es la siguiente:[8]
- Filo Preplasmiviricota
- Familia Lavidaviridae
- Familia Adintoviridae
- Familia Finnlakeviridae[9]
- Clase Tectiliviricetes
- Familia Adenoviridae
- Familia Tectiviridae
- Familia Turriviridae
- Orden Vinavirales
- Familia Corticoviridae
- Familia Autolykiviridae
Referencias
- Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de octubre de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins» (docx). International Committee on Taxonomy of Viruses (en inglés). Consultado el 10 de junio de 2020.
- Kauffman KM, Hussain FA, Yang J, Arevalo P, Brown JM, Chang WK, VanInsberghe D, Elsherbini J, Sharma RS, Cutler MB, Kelly L, Polz MF (1 de febrero de 2018). «A Major Lineage of Non-Tailed dsDNA Viruses as Unrecognized Killers of Marine Bacteria». Nature 554 (7690): 118-122. PMID 29364876. doi:10.1038/nature25474.
- Natalya Yutin, Eugene Koonin (2019). Chapter Five - Evolution of the Large Nucleocytoplasmic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. Science Direct.
- Natalya Yutin, Yuri I.Wolf, Eugene V.Koonin (2014). Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. Science Direct. https://doi.org/10.1016/j.virol.2014.06.032
- Jonathan Filée (2013). Route of NCLDV evolution: the genomic accordion. Science Direct.
- David Moreira, Céline Brochier-Armanet (2008). Giant viruses, giant chimeras: The multiple evolutionary histories of Mimivirus genes. NCBI.
- Tom Williams, T Martin Embley, Eva Heinz (2011). Informational Gene Phylogenies Do Not Support a Fourth Domain of Life for Nucleocytoplasmic Large DNA Viruses. Researchgate.
- «Virus Taxonomy: 2019 Release» (html). International Committee on Taxonomy of Viruses (ICTV) (en inglés). October 2018. Consultado el 13 octobre 2019.
- Mart Krupovic, Valerian V. Dolja, Eugene V. Koonin (2020). The LUCA and its complex virome. Nature.