Unión de extremos no homólogos
La recombinación no homóloga o unión de extremos no homólogos (cuya sigla en inglés es NHEJ Non-homologous DNA end joining) es una de las vías para la reparación de roturas en la doble cadena de ADN (en inglés DBS "Double-strand break").
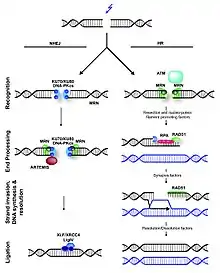
Consiste en 3 fases: fase de reconocimiento, fase de procesamiento y fase de terminación. El inicio del proceso se desencadena por la unión de un complejo proteico heterodimérico compuesto por las proteínas KU70 y KU80, el cual se une a ambos extremos rotos del ADN de doble cadena. La unión de este complejo heterodimérico KU a los extremos rotos del ADN es lo que ocurre inmediatamente después de originarse el DSB, esto es especialmente importante ya que sirve como base para la intervención de otras proteínas importantes que permitan la continuar el proceso de reparación, y así formar una estructura que finalmente logré la unión de los extremos, recordemos que el objetivo es el "alineamiento de los dos extremos terminales del ADN". El heterodímero KU se desliza hacia el interior de la doble hebra de ADN, permitiendo que DNA-PKcs (La subunidad catalítica de la proteína quinasa dependiente de ADN codificada en humanos por el gen PRKDC), interactúe con el extremo de ADN para activar su función quinasa, este desplazamiento hacia el interior permite que dos moléculas de DNA-PKcs interaccionen a lo largo del DSB formando un “puente” molecular o sinapsis entre los dos extremos del ADN, lo que permite la funcionalidad. La DNA-PKcs, tiene una actividad fundamental, ya que estabiliza los extremos del ADN y evita la resección gracias a que realiza reacciones de autofosforilación; en resumen, el complejo DNA-heterodímero KU permite la entrada de la importantísima proteína DNA-PKcs para continuar con el proceso. No está dilucidada completamente cuál es la importancia de estas reacciones de fosforilación en la recombinación homóloga. Lo que se ha podido observar en sistemas "in vivo" es que las autofosforilaciones de esta subunidad catalítica DNA-PKcs son indicadores de daños en el ADN. A toda esta estructura formada en la fase de reconocimiento podemos tomarla a como un complejo formado por los 3 componentes mencionados anteriormente (KU70, KU80 y la subunidad catalítica ADN-PKcs). Para que se lleve a cabo la unión "alineamiento de los dos extremos terminales del ADN" es necesario el complejo ARTEMIS/ADN-PKcs, con actividad nucleasa y el complejo XRCC4/liga-saIV, para la ligación en el paso final del proceso. Este mecanismo está sometido a errores frecuentes, ya que solamente se está uniendo los extremos, lo que implica por tanto muchas veces de partes de secuencias (Pérdida de nucleótidos) en el punto de reparación. Este mecanismo de reparación desarrollado bastante en varios eucariotas, principalmente mamíferos, aunque también se ha encontrado en organismos procariotas, lo que indica que es un proceso muy conservado.
Es denominada no homóloga porque los extremos rotos son directamente ligados sin la necesidad de un molde homólogo, en contraste con la recombinación homóloga, que requiere una secuencia homóloga para guiar la reparación. El término "unión de extremos no homólogos" fue acuñado en 1996 por Moore y Haber.[1]
NHEJ está conservada en todos los reinos de la vida y es la ruta de reparación del ADN de doble hebra predominante en muchos organismos, incluyendo eucariotas superiores como humanos y ratones. En levaduras gemantes (Saccharomyces cerevisiae), sin embargo, la recombinación homóloga domina cuando el organismo está creciendo bajo condiciones comunes de laboratorio. Aunque se pensaba que los organismos procariotas no presentaban mecanismos de este tipo, algunas especies de bacterias, como Mycobacterium tuberculosis o Escherichia coli tienen un sistema alternativo de unión de extremos no homólogos (A-EJ, del inglés alternative end joining[2]).
NHEJ típicamente utiliza secuencias cortas de ADN homólogo llamadas microhomologías para guiar la reparación. Estas microhomologías están a menudo presentes en salientes de hebra simple en los extremos de las roturas de la doble hebra. Cuando las salientes son perfectamente compatibles, NHEJ usualmente repara la rotura con precisión.[1][3][4][5] La reparación imprecisa también puede conducir a la pérdida de nucleótidos, pero es mucho más común que las salientes no son compatibles. NHEJ inapropiados pueden conducir a translocaciones y fusión de telómeros, sellos de las células cancerosas.[6]
Referencias
- Moore JK, Haber JE. Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae. Mol Cell Biol. 1996 May;16(5):2164-73. PMID 8628283
- Chang, Howard H. Y.; Pannunzio, Nicholas R.; Adachi, Noritaka; Lieber, Michael R. (17 de mayo de 2017). «Non-homologous DNA end joining and alternative pathways to double-strand break repair». Nature Reviews Molecular Cell Biology 18 (8): 495-506. ISSN 1471-0072. doi:10.1038/nrm.2017.48. Consultado el 21 de mayo de 2019.
- Boulton SJ, Jackson SP. Saccharomyces cerevisiae Ku70 potentiates illegitimate DNA double-strand break repair and serves as a barrier to error-prone DNA repair pathways. EMBO J. 1996 Sep 16;15(18):5093-103. PMID 8890183
- Wilson, T. E., and Lieber, M. R. Efficient processing of DNA ends during yeast nonhomologous end joining. Evidence for a DNA polymerase beta (Pol4)-dependent pathway. (1999) J. Biol. Chem. 274, 23599–23609. PMID 10438542
- Budman J, Chu G. Processing of DNA for nonhomologous end-joining by cell-free extract. EMBO J. 2005 Feb 23;24(4):849-60. PMID 15692565
- Espejel S, Franco S, Rodriguez-Perales S, Bouffler SD, Cigudosa JC, Blasco MA. Mammalian Ku86 mediates chromosomal fusions and apoptosis caused by critically short telomeres. EMBO J. 2002 May 1;21(9):2207-19. PMID 11980718