Clamp (ADN)
En biologie moléculaire, on appelle clamp à ADN un motif structurel caractéristique de certaines protéines et enzymes intervenant dans la réplication de l'ADN et favorisant la promotion et la processivité de ce processus. Constituant indispensable du réplisome formé par l'holoenzyme de l'ADN polymérase III chez les bactéries, le clamp se lie à l'ADN polymérase et l'empêche de se dissocier du brin d'ADN à répliquer. Les interactions clamp-polymérase sont plus intenses et plus spécifiques que les interactions directes entre la polymérase et le brin d'ADN modèle. Cette dernière étant l'un des paramètres limitants de la synthèse de l'ADN, la présence d'une telle pince à ADN accroît considérablement le nombre de nucléotides qu'une polymérase est susceptible d'ajouter à un brin d'ADN en croissance avant de se dissocier du brin modèle, jusqu'à trois ordres de grandeur (facteur 1000) par rapport à une polymérase non processive[3].
Pour les articles homonymes, voir Clamp (homonymie).
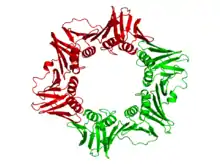
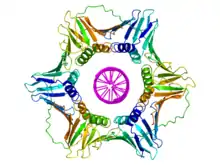
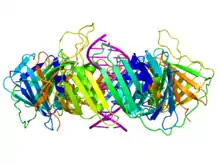
Le clamp est constitué d'un ensemble d'hélices α et de feuillets β assemblés en formant une structure polymérique qui encercle complètement la double hélice d'ADN pendant que la polymérase ajoute des nucléotides sur le brin en cours de croissance[4]. Cette structure s'assemble sur l'ADN au niveau de la fourche de réplication et glisse le long de l'ADN au fur et à mesure que la polymérase progresse, une couche de molécules d'eau située entre l'ADN et la protéine facilitant la progression de l'ensemble. La double hélice d'ADN ne peut se dégager du clamp sans dissocier celui-ci en monomères.
On trouve de tels clamps chez les bactéries, les archées, les eucaryotes et certains virus. Chez les bactéries, il s'agit d'un homodimère constitué de deux sous-unités β de l'ADN polymérase III, de sorte qu'on l'appelle clamp β. Chez les archées[5] et les eucaryotes, il s'agit d'un trimère constitué de trois molécules de PCNA. Le phage T4 utilise également un clamp, appelé gp45, qui est un trimère dont la structure est semblable à celle du PCNA mais dont la séquence est cependant différente à la fois du PCNA et du clamp β[4].
| Domaine | Protéine de clamp | Degré d'agrégation | ADN polymérase associée |
|---|---|---|---|
| Bactéries | Sous-unité β de l'ADN polymérase III | Dimère | ADN polymérase III |
| Archées | PCNA archéen | Trimère | ADN polymérase ε |
| Eucaryotes | PCNA | Trimère | ADN polymérase δ |
| Virus | gp43 / gp45 | Trimère | Pol RB69 / Pol T4 |
Les clamps sont chargés sur l'ADN à répliquer par des protéines spécialisées appelées « chargeurs de clamp », qui assurent également leur désassemblage une fois la réplication terminée. Les sites de liaison pour ces protéines d'initiation se superposent aux sites de liaison avec l'ADN polymérase, de sorte que le clamp ne peut se lier en même temps au chargeur de clamp et à la polymérase. C'est la raison pour laquelle le clamp ne peut être désassemblé par le chargeur de clamp tant qu'il demeure lié à la polymérase.
Les clamps peuvent également se lier à d'autres facteurs intervenant dans l'homéostase génomique, tels que les facteurs d'assemblage des nucléosomes, les ADN ligases des fragments d'Okazaki et les protéines de réparation de l'ADN. Toutes ces protéines partagent un site de liaison sur le clamp qui se superpose à celui du chargeur de clamp, de sorte que le clamp ne peut être dissocié tant que l'une de ces enzymes demeure active sur l'ADN. Le fonctionnement du chargeur de clamp requiert l'hydrolyse d'une molécule d'ATP pour refermer le clamp autour de l'ADN.
Notes et références
- (en) George Kontopidis, Su-Ying Wu, Daniella I. Zheleva, Paul Taylor, Campbell McInnes, David P. Lane, Peter M. Fischer et Malcolm D. Walkinshaw, « Structural and biochemical studies of human proliferating cell nuclear antigen complexes provide a rationale for cyclin association and inhibitor design », Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no 6, , p. 1871–1876 (PMID 15681588, PMCID 548533, DOI 10.1073/pnas.0406540102, Bibcode 2005PNAS..102.1871K, lire en ligne)
- (en) A. J. Oakley, P. Prosselkov, G. Wijffels, J. L. Beck, M. C. J. Wilce et N. E. Dixon, « Flexibility revealed by the 1.85 Å crystal structure of the β sliding-clamp subunit of Escherichia coli DNA polymerase III », Acta Crystallographica Section D – Biological Crystallography, vol. 59, no Part 7, , p. 1192-1199 (PMID 12832762, DOI 10.1107/S0907444903009958, lire en ligne)
- (en) V. Mizrahi, R. N. Henrie, J. F. Marlier, K. A. Johnson et S. J. Benkovic, « Rate-limiting steps in the DNA polymerase I reaction pathway », Biochemistry, vol. 24, no 15, , p. 4010-4018 (PMID 3902078, DOI 10.1021/bi00336a031, lire en ligne)
- (en) Irina Bruck et Mike O'Donnell, « The ring-type polymerase sliding clamp family », Genome Biology, vol. 2, no 1, , reviews3001-reviews3001.3 (PMID 11178284, PMCID 150441, DOI 10.1186/gb-2001-2-1-reviews3001, lire en ligne)
- (en) Shigeki Matsumiya, Yoshizumi Ishino et Kosuke Morikawa, « Crystal structure of an archaeal DNA sliding clamp: Proliferating cell nuclear antigen from Pyrococcus furiosus », Protein Science, vol. 10, no 1, , p. 17-23 (PMID 11266590, PMCID 2249843, DOI 10.1110/ps.36401, lire en ligne)
- Portail de la biologie cellulaire et moléculaire