DBSCAN
DBSCAN (density-based spatial clustering of applications with noise) est un algorithme de partitionnement de données proposé en 1996 par Martin Ester, Hans-Peter Kriegel, Jörg Sander et Xiaowei Xu[1]. Il s'agit d'un algorithme fondé sur la densité dans la mesure qui s’appuie sur la densité estimée des clusters pour effectuer le partitionnement.
Principe général
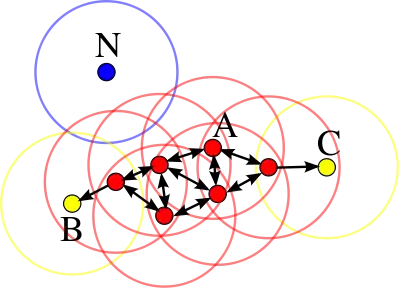
L'algorithme DBSCAN utilise 2 paramètres : la distance et le nombre minimum de points devant se trouver dans un rayon pour que ces points soient considérés comme un cluster. Les paramètres d'entrées sont donc une estimation de la densité de points des clusters. L'idée de base de l'algorithme est ensuite, pour un point donné, de récupérer son -voisinage et de vérifier qu'il contient bien points ou plus. Ce point est alors considéré comme faisant partie d'un cluster. On parcourt ensuite l'-voisinage de proche en proche afin de trouver l'ensemble des points du cluster.
Algorithme
DBSCAN(D, eps, MinPts)
C = 0
pour chaque point P non visité des données D
marquer P comme visité
PtsVoisins = epsilonVoisinage(D, P, eps)
si tailleDe(PtsVoisins) < MinPts
marquer P comme BRUIT
sinon
C++
etendreCluster(D, P, PtsVoisins, C, eps, MinPts)
etendreCluster(D, P, PtsVoisins, C, eps, MinPts)
ajouter P au cluster C
pour chaque point P' de PtsVoisins
si P' n'a pas été visité
marquer P' comme visité
PtsVoisins' = epsilonVoisinage(D, P', eps)
si tailleDe(PtsVoisins') >= MinPts
PtsVoisins = PtsVoisins U PtsVoisins'
si P' n'est membre d'aucun cluster
ajouter P' au cluster C
epsilonVoisinage(D, P, eps)
retourner tous les points de D qui sont à une distance inférieure à epsilon de P
Structure des solutions[2]
Les points du jeu de données sont séparés en 3 types :
- Les points centraux (core points)
- Les points frontières (border points)
- Les points aberrants (noise points)
Les points centraux
Un point du jeu de données est dit central si :
- Son voisinage est dense
Ces points forment des composantes connexes indépendantes de l'ordre d'exploration du jeu données.
Les points frontières
Un point du jeu de données est dit frontière si :
- Ce n'est pas un point central
- Il appartient au voisinage d'un point central
Ces points viennent s'agréger autour des composantes connexes pour former des groupes. Ces groupes sont couramment appelées par leur nom anglais : Clusters.
Contrairement aux composantes connexes, la formation des clusters est dépendante de l'ordre d'exploration du jeu de données. En effet un point frontière est assigné au premier cluster rencontré lors de l'étape d'expansion. En plus de l'ordre d'exploration, ces points sont sensibles aux différentes implémentations de l'algorithme DBSCAN.
Les points aberrants
Un point du jeu de données est dit aberrant si :
- Ce n'est pas un point central
- Ce n'est pas un point frontière
Ces points sont donc tous les autres points du jeu de données.
Attention, le nom donné à ces points peut être trompeur car leur désignation dépend des paramètres choisis.
Concepts mathématiques sous-jacent
Voisinage d'un point
La notion de voisinage est le concept élémentaire à la base de la méthode DBSCAN. Il permet de définir mathématiquement les voisinages denses qui sont utilisés pour la localisation des points centraux et l'expansion des clusters.
Distance entre points
En mathématiques, on appelle distance sur un ensemble E toute application d définie sur le produit E2 = E×E et à valeurs dans l'ensemble ℝ+ des réels positifs,
vérifiant les propriétés suivantes
| Nom | Propriété |
|---|---|
| symétrie | |
| séparation | |
| inégalité triangulaire |
Le choix de la distance entre points est un paramètre implicite à la méthode DBSCAN.
Dans le cas de DBSCAN, c'est communément la distance euclidienne qui est utilisée.
Boule ouverte de rayon de epsilon
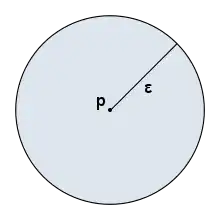
Dans l'espace usuel comme dans n'importe quel espace métrique :
La boule ouverte est l'ensemble des points de l'espace dont la distance au point est strictement inférieure à :
Le point est alors appelé centre de la boule ouverte et est appelé le rayon de la boule ouverte .
Les caractéristiques des boules dépendent de deux éléments :
- La forme des boules est liée à la distance (paramètre implicite à DBSCAN).
- L'espace couvert dépend du rayon choisie (paramètre explicite à DBSCAN).
Ci contre une boule ouverte de centre le point et de rayon est représentée dans un espace à deux dimensions pour une distance euclidienne. La surface bleu représente la boule ouverte : Celle-ci correspond à la zone de recherche des voisins pour l'algorithme DBSCAN.
Epsilon-voisinage
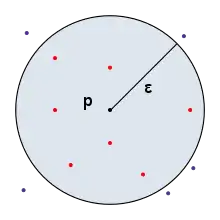
L'-voisinage d'un point , est l'ensemble des points du jeu de données situés dans la boule ouverte centrée en et de rayon :
Les points du jeu de données sélectionnés reposent sur les boules ouvertes et sont donc dépendants des paramètres suivants :
- La distance entre les points et
- Le rayon de recherche autour du point
Ci-contre un -voisinage du point a été représenté dans un espace de dimension 2 avec une distance euclidienne. Les points rouges sont les points du jeu de données qui appartiennent à l' -voisinage du point , et les points de couleur indigo représentent les points du jeu de données qui n'appartiennent pas à l' -voisinage du point . Pour l'algorithme DBSCAN, c'est le nombre de points rouges qui est important.
Epsilon-voisinage dense
Un -voisinage est dit dense si son cardinal est supérieur ou égal à .
Cette définition est la première qui dépend des 3 paramètres de DBSCAN :
- Le nombre minimum de points pour qu'un voisinage soit désigné dense.
- Le rayon du voisinage autour du point considéré.
- La distance entre points.
En reprenant l'illustration précédente, est fixé. Si est fixé à 3 points alors le voisinage considéré est clairement dense. À l’inverse si est fixé à 50 points alors le voisinage n'est clairement pas dense.
Relations basées sur la densité
Les relations binaires qui suivent sont utilisés pour effectuer des démonstrations sur la méthode DBSCAN, et plus généralement en apprentissage non supervisé basé sur la densité.
Directement accessible par densité
Un point du jeu de données est directement accessible par densité depuis un autre point si :
- est dense
Autrement dit, les points directement accessible par densité sont les points d'un voisinage dense.
Une telle relation est graphiquement représentée par un flèche qui part du point vers le point .
Accessible par densité
Un point du jeu de données est accessible par densité depuis un autre point s'il existe une séquence ordonnée de points tel que :
- est directement accessible par densité depuis
Un point est accessible par densité depuis un autre points s'il existe un chemin fléché vers de vers .
Densément connecté
Un point du jeu de données est densément connecté à un autre point si :
- est accessible par densité depuis
- est accessible par densité depuis
Un point et un point sont densément connecté s'il existe deux chemins fléché qui partent d'un même point pour aller respectivement vers les points et .
Estimation des paramètres
L'estimation des paramètres et n'est pas un problème facile, car ces deux valeurs sont intrinsèquement liées à la topologie de l'espace à partitionner. Une trop faible valeur de et/ou une trop grande valeur de peuvent empêcher l'algorithme de propager les clusters. À l'inverse, une trop grande valeur pour et/ou une trop faible valeur pour peuvent conduire l'algorithme à ne renvoyer que du bruit. Une heuristique[3] permettant de déterminer conjointement et pour un certain espace pourrait être donnée par :
- : calculer pour chaque point de l'espace la distance à son plus proche voisin. Prendre tel qu'une part « suffisamment grande » des points aient une distance à son plus proche voisin inférieure à ;
- : calculer pour chaque point le nombre de ses voisins dans un rayon de taille (la taille de son -voisinage). Prendre tel qu'une part « suffisamment grande » des points aient plus de points dans leur -voisinage.
Par « suffisamment grand » on entend, par exemple, % ou % des points.
Une autre heuristique pour les cas en 2D (définie dans l'article original de DBSCAN[1]) consiste à fixer la valeur de à 4, et à tracer la courbe (triée dans l'ordre décroissant) des distances de chaque point à leur 4ème plus proche voisin. On fixe alors à la valeur du "point seuil" repéré sur le graphe. Si ce seuil n'est pas clairement identifiable, l'utilisateur peut le fixer en estimant le pourcentage de bruit dans le jeu de données : est donc tel que seuls les termes de bruit ont une distance à leur 4ème plus proche voisin plus grande que .
Avantages et inconvénients
L'algorithme est très simple et ne nécessite pas qu'on lui précise le nombre de clusters à trouver. Il est capable de gérer les données aberrantes en les éliminant du processus de partitionnement. Les clusters n'ont pas pour obligation d'être linéairement séparables (tout comme pour l'algorithme des k-moyennes par exemple). Cependant, il n'est pas capable de gérer des clusters de densités différentes.
Articles connexes
Références
- M. Ester, H.-P. Kriegel, J. Sander, and X. Xu, “A density-based algorithm for discovering clusters in large spatial databases with noise,” in Proceedings of the 2nd International Conference on Knowledge Discovery and Data mining, 1996, pp. 226–231.
- (en) Michael Hahsler, Matthew Piekenbrock et Derek Doran, « dbscan : Fast Density-Based Clustering with R », Journal of Statistical Software, vol. 91, no 1, (ISSN 1548-7660, DOI 10.18637/jss.v091.i01, lire en ligne, consulté le )
- alitouka, spark_dbscan: DBSCAN clustering algorithm on top of Apache Spark, (lire en ligne)
- Portail de l’informatique