FreeSurfer
FreeSurfer est un logiciel d'imagerie des IRM de cerveau développé par Athinoula A. Martinos Center for Biomedical Imaging[2] du Massachusetts General Hospital avec le support du CorTechs Labs, Inc, La Jolla, CA[3]. C'est un outil important d'imagerie neurofonctionnelle, et facilite la visualisation des régions d'activations fonctionnelles du cortex cérébral, jusque-là rendue difficile par le nombre important de plis de celui-ci. FreeSurfer permet à la fois des analyses surfaciques et volumiques, qui utilisent prioritairement la surface de la matière blanche[4]. Sont également inclus des outils pour une reconstruction topologiquement correct et géométriquement précise de la surface piale et de l'interface matière grise/blanche. Mesurer l'épaisseur cortical, les propriétés des surfaces et des plis corticaux et effectuer des registrations (trouver la transformation linéaire ou non-linéaire entre le cerveau d'un sujet particulier et un cerveau qui sert d'atlas) basées sur un modèle cortical sont des autres fonctionnalités de FreeSurfer. De plus, le logiciel est capable de séparer automatiquement 35 régions non corticales.
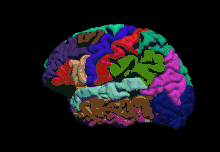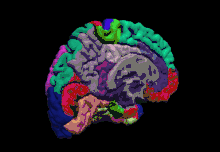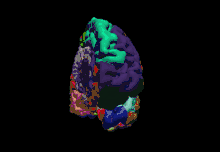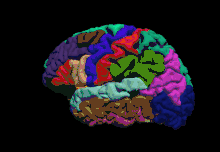
| Dernière version | 7.1.1 ()[1] |
|---|---|
| Dépôt | github.com/freesurfer/freesurfer |
| Système d'exploitation | MacOS et Linux |
| Formats lus | NII (d) |
| Licence | Licence à sources ouvertes (en) |
| Documentation | www.freesurfer.net/fswiki/FreeSurferWiki |
| Site web | www.freesurfer.net |
Usage
En pratique, le flot de traitement d'une image est appelé recon-all[5], qui transforme les fichiers sources IRM en un format facilement lisible pour les analyses statistiques morphométriques et fonctionnelles réalisées avec FsFast. FreeSurfer permet, grâce à ses outils de registration, de réaliser des analyses fonctionnelles de groupe sur des projections sphériques de cerveau. Tkmedit et Tksurfer sont les outils d'édition et de visualisation des images IRM mais seront bientôt remplacés par Scuba. Le logiciel permet également la parcellisation automatique de la surface du cerveau en un certain nombre de régions d'intérêt standard que le Center for Morphometric Analysis a développé. Ce projet a d'autres applications encore comme la rétinotopie ou l'analyse d'épaisseur corticale.
Compatibilité
FreeSurfer est facilement compatible avec FMRIB Software Library (FSL), qui est une bibliothèque d'analyse d'images et d'outils statistiques développée par le groupe Functional MRI of the Brain (FMRIB) à Oxford, Royaume-Uni. Ainsi, les résultats fonctionnels d'activation obtenus soit avec FreeSurfer Functional Analysis Stream (FS-FAST) ou FSL peuvent être superposés sur les surfaces corticales de FreeSurfer, soit enflées, soit sous forme sphériques, voire aplaties. Le logiciel utilise également des outils provenant d'autres bibliothèques comme MNI MINC, VXL, Tcl/Tk/Tix/BLT et Qt qui sont incluses lors du téléchargement. D'autres logiciels de neuro-imagerie comme Caret et 3D Slicer peuvent importer des données générées par FreeSurfer.
Téléchargement
FreeSurfer fonctionne sur Mac OS et Linux. L'inscription et l'installation sont gratuites. La documentation se trouve sur la page Wiki de FreeSurfer et une assistance gratuite est en place de la part des développeurs et de la communauté des utilisateurs, via la liste de diffusion FreeSurfer.
Voir aussi
Articles connexes
- Cartographie statistique paramétrique
- FMRIB Software Library
Liens externes
- Caret Van Essen Lab, Washington University in St. Louis
- LONI Pipeline Laboratory of Neuro Imaging, UCLA
- MNI MINC McConnell Brain Imaging Center, Montreal Neurological Institute, McGill University
- FreeSurfer Articles Overview and Contents Graham Wideman fournit une revue complète du flot de travail de FreeSurfer avec une description du format des fichiers
- Résume de FreeSurfer sur le site du Martinos Center
- Page d'accueil de FreeSurfer
- Page wiki de FreeSurfer
Références
- « Release 7.1.1 », (consulté le )
- Athinoula A. Martinos Center for Biomedical Imaging
- CorTechs Labs, Inc, La Jolla, CA
- (en) [PDF] erreur modèle {{Lien archive}} : renseignez un paramètre «
|titre=» Dale, A. M., B. Fischl, et al. (1999). "Cortical surface-based analysis: Segmentation and surface reconstruction." NeuroImage 9(2): 179-94. - (en) Tableau d'aide à la reconstruction
- Portail de l’imagerie numérique