Glissière à leucine
Une glissière à leucine, ou leucine zipper en anglais, est un domaine fonctionnel tridimensionnel courant des protéines. On les considère comme des cas particuliers des superhélices, comme celles de la kératine α. De telles structures ont été décrites pour la première fois par Landschulz et al. en 1988[1] en projetant sur une hélice α la séquence particulière d'une segment d'une trentaine de résidus d'acides aminés d'un activateur de protéine de liaison : un résidu sur sept était de la leucine sur huit tours d'hélice. Il a alors été proposé que ces segments contenant des répétitions régulières de résidus de leucine forment des structures α-hélicoïdales le long desquelles les chaînes latérales de leucine sont alignées d'un côté de l'hélice et peuvent interagir avec d'autres structures semblables pour faciliter la dimérisation des complexes protéiques ainsi formés.
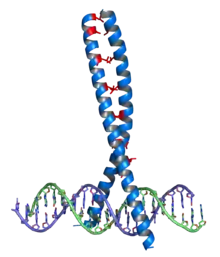
On trouve des glissières à leucine dans le domaine de dimérisation des facteurs de transcription d'eucaryotes de la classe des bZIP (basic-region leucine zipper)[2]. Ces domaines bZIP sont longs de 60 à 80 résidus d'acides aminés avec un domaine de liaison à l'ADN très conservé et une région de dimérisation davantage diversifiée contenant une glissière à leucine[3]. La localisation des résidus de leucine le long des hélices α est déterminante pour la liaison des protéines à l'ADN. On trouve des glissières à leucine dans les protéines régulatrices des procaryotes comme des eucaryotes, mais elles sont surtout caractéristiques de ces derniers. On les retrouve également dans d'autres protéines que les facteurs de transcription et on pense qu'elles constituent l'un des modes d'interaction protéine-protéine[4].
Le mécanisme de la régulation de la transcription par les protéines bZIP a été étudié en détail. La plupart de ces protéines ont une affinité élevée pour les motifs ACGT, comprenant les séquences CACGTG (boîte G), GACGTC (boîte C), TACGTA (boîte A), AACGTT (boîte T), et un motif GCN4 correspondant à TGA(G/C)TCA[1],[3],[5]. Un petit nombre de facteurs bZIP tels que OsOBF1 sont également capables de reconnaître les séquences palindromiques[6]. Cependant, les autres protéines bZIP, dont LIP19, OsZIP-2a et OsZIP-2b, ne se lient pas à des séquences d'ADN, mais se lient à d'autres protéines bZIP pour réguler les processus de transcription[6],[7].
Séquence et structure
Les dimères bZIP interagissent avec l'ADN au niveau de l'extrémité N-terminale des deux monomères en hélice α. Des résidus de lysine et d'arginine, très basiques, qui se trouvent dans cette région, interagissent avec le grand sillon de la double hélice d'ADN, à la base des interactions spécifiques des séquences nucléotidiques. La glissière à leucine se trouve du côté C-terminal, formant une hélice α amphiphile dont le côté hydrophobe de deux hélices voisines provoque la dimérisation de celles-ci.
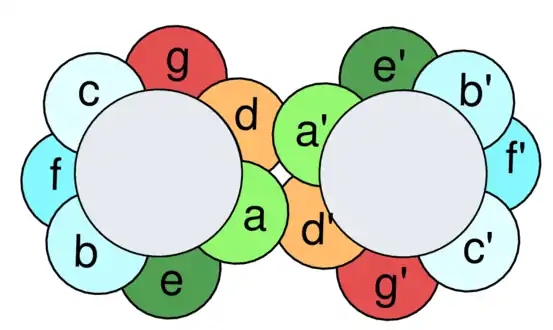
Les glissières à leucine sont considérées comme des cas particuliers des superhélices, qui sont formées d'au moins deux hélices α qui s'enroulent les unes autour des autres. Leur séquence en acides aminés présent des motifs répétés tous les sept résidus, formant des heptades dans lesquelles les résidus sont conventionnellement désignés par les lettres a à g : dans ce cadre, les résidus désignés par les lettres a et d sont généralement hydrophobes et distribués en zigzag d'un côté de l'hélice en formant des bosses et des creux complémentaires qui s'emboîtent étroitement entre deux hélices dimérisées.
Dans le cas des glissières à leucine, les résidus de leucine sont prédominants en position d de l'heptade constituant le motif répété. Ces résidus interagissent les uns avec les autres un tour d'hélice sur deux, et la région hydrophobe est complétée, entre deux positions d consécutives, par les résidus en position a, qui sont généralement également hydrophobes. Ce type de structure est appelé superhélice dans le cas général, mais on fait plus spécifiquement référence au domaine bZIP dès lors que celui-ci revêt une importance déterminante pour la fonction biochimique de la protéine[8].
Références
- (en) W. H. Landschulz, P.F. Johnson et S. L. McKnight, « The leucine zipper: a hypothetical structure common to a new class of DNA binding proteins », Science, vol. 240, no 4860, , p. 1759-1764 (PMID 3289117, DOI 10.1126/science.3289117, Bibcode 1988Sci...240.1759L, lire en ligne)
- (en) C. R. Vinson, P. B. Sigler et S. L. McKnight, « Scissors-grip model for DNA recognition by a family of leucine zipper proteins », Science, vol. 246, no 4932, , p. 911-916 (PMID 2683088, DOI 10.1126/science.2683088, Bibcode 1989Sci...246..911V, lire en ligne)
- (en) Z.G. E, Y. P. Zhang, J. H. Zhou et L. Wang, « Mini review – Roles of the bZIP gene family in rice », Genetics and Molecular Research, vol. 13, no 2, , p. 3025-3036 (PMID 24782137, DOI 10.4238/2014.April.16.11, lire en ligne)
- (en) Toshio Hakoshima, « Leucine Zippers », eLS, (DOI 10.1002/9780470015902.a0005049.pub2, lire en ligne)
- (en) Aashima Nijhawan, Mukesh Jain, Akhilesh K. Tyagi et Jitendra P. Khurana, « Genomic Survey and Gene Expression Analysis of the Basic Leucine Zipper Transcription Factor Family in Rice », Plant Physiology, vol. 146, no 2, , p. 333-350 (PMID 18065552, PMCID 2245831, DOI 10.1104/pp.107.112821, lire en ligne)
- (en) Hidekazu Shimizu, Kazuhito Sato, Thomas Berberich, Atsushi Miyazaki, Rei Ozaki, Ryozo Imai et Tomonobu Kusano, « LIP19, a Basic Region Leucine Zipper Protein, is a Fos-like Molecular Switch in the Cold Signaling of Rice Plants », Plant & Cell Physiology, vol. 46, no 10, , p. 1623-1634 (PMID 16051676, DOI 10.1093/pcp/pci178, lire en ligne)
- (en) André Nantel et Ralph S. Quatrano, « Characterization of Three Rice Basic/Leucine Zipper Factors, Including Two Inhibitors of EmBP-1 DNA Binding Activity », Journal of Biological Chemistry, vol. 271, no 49, , p. 31296-31305 (PMID 8940135, DOI 10.1074/jbc.271.49.31296, lire en ligne)
- (en) Dmitry Krylov, Charles R. Vinson, « Leucine Zipper », eLS, (DOI 10.1038/npg.els.0003001, lire en ligne)
- Portail de la biochimie