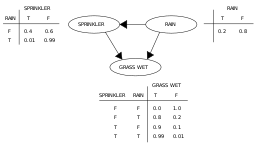
Réseau bayésien
En informatique et en statistique, un réseau bayésien est un modèle graphique probabiliste représentant un ensemble de variables aléatoires sous la forme d'un graphe orienté acyclique. Intuitivement, un réseau bayésien est à la fois :
- un modèle de représentation des connaissances ;
- une « machine à calculer » des probabilités conditionnelles
- une base pour des systèmes d'aide à la décision[1]
Pour un domaine donné (par exemple médical[1]), on décrit les relations causales entre variables d'intérêt par un graphe. Dans ce graphe, les relations de cause à effet entre les variables ne sont pas déterministes, mais probabilisées. Ainsi, l'observation d'une cause ou de plusieurs causes n'entraîne pas systématiquement l'effet ou les effets qui en dépendent, mais modifie seulement la probabilité de les observer.
L'intérêt particulier des réseaux bayésiens est de tenir compte simultanément de connaissances a priori d'experts (dans le graphe) et de l'expérience contenue dans les données.
Les réseaux bayésiens sont surtout utilisés pour le diagnostic (médical et industriel), l'analyse de risques, la détection des spams et le data mining.
Intuition
Un exemple dans la modélisation des risques
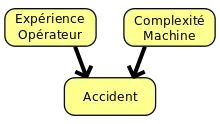
Un opérateur travaillant sur une machine risque de se blesser s’il l’utilise mal. Ce risque dépend de l’expérience de l’opérateur et de la complexité de la machine. « Expérience » et « Complexité » sont deux facteurs déterminants de ce risque (fig. 1).
Bien sûr, ces facteurs ne permettent pas de créer un modèle déterministe. Quand bien même l’opérateur serait expérimenté et la machine simple, un accident reste possible. D’autres facteurs peuvent jouer : l’opérateur peut être fatigué, dérangé, etc. La survenance du risque est toujours aléatoire, mais la probabilité de survenance dépend des facteurs identifiés.
La figure 1 ci-dessous représente la structure de causalité de ce modèle (graphe).
La figure 2 représente la probabilisation de la dépendance : on voit que la probabilité d'accident augmente si l'utilisateur est peu expérimenté ou la machine complexe.
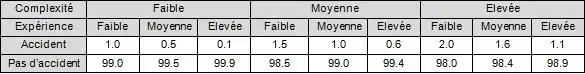
On voit ici comment intégrer des connaissances d'expert (les facteurs déterminants) et des données (par exemple, la table de probabilité d'accident en fonction des déterminants peut venir de statistiques).
Construction de réseaux bayésiens
Construire un réseau bayésien, c'est donc :
- définir le graphe du modèle ;
- définir les tables de probabilité de chaque variable, conditionnellement à ses causes.
Le graphe est aussi appelé la « structure » du modèle, et les tables de probabilités ses « paramètres ». Structure et paramètres peuvent être fournis par des experts, ou calculés à partir de données, même si en général, la structure est définie par des experts et les paramètres calculés à partir de données expérimentales.
Utilisation d'un réseau bayésien
L'utilisation d'un réseau bayésien s'appelle « inférence ». Le réseau bayésien est alors véritablement une « machine à calculer des probabilités conditionnelles ». En fonction des informations observées, on calcule la probabilité des données non observées. Par exemple, en fonction des symptômes d'un malade, on calcule les probabilités des différentes pathologies compatibles avec ces symptômes. On peut aussi calculer la probabilité de symptômes non observés, et en déduire les examens complémentaires les plus intéressants.
Définition formelle
Loi de probabilité jointe
Il existe plusieurs façons de définir un réseau bayésien. La plus simple exprime la loi de probabilité jointe sur l'ensemble des variables aléatoires modélisées dans le réseau. Un réseau bayésien est un graphe orienté acyclique G = (V, E) avec V l'ensemble des nœuds du réseaux et E l'ensemble des arcs. À chaque nœud x appartenant à V du graphe est associé la distribution de probabilité conditionnelle suivante :
- où pa(x) représente les parents immédiats de x dans V
L'ensemble V est donc un ensemble discret de variables aléatoires. Chaque nœud de V est conditionnellement indépendant de ses non-descendants, étant donné ses parents immédiats. Il est ainsi possible de factoriser les distributions de probabilité conditionnelles sur l'ensemble des variables en en faisant le produit[2],[3] :
Ce résultat est parfois noté JPD, pour distribution de probabilité jointe. Cette définition peut être retrouvée dans l'article Bayesian Networks: A Model of Self-Activated Memory for Evidential Reasoning, où Judea Pearl introduit le terme « réseau bayésien »[4].
Dans l'exemple donné figure 1, la probabilité jointe est égale à :
- .
Propriété de Markov globale
Une autre façon de définir un réseau bayésien est d'évaluer si un graphe orienté acyclique G = (V, E) vérifie la propriété de Markov globale (dite orientée ici, en raison de la nature de G) étant donné une loi de probabilité P sur l'ensemble des variables V. Soit X, Y, S trois sous-ensemble de V, tels que X et Y sont d-séparés par S, noté (X|S|Y) : la propriété de Markov globale exprime l'indépendance conditionnelle entre X et Y, c'est-à-dire que X est indépendant de Y conditionnellement à S. Formellement, la propriété dans un graphe orienté s'énonce ainsi[5] :
Il convient de définir la d-séparation, pour « séparation dirigée » ou « séparation orientée », dans un graphe orienté. Les sous-ensembles X et Y sont d-séparés par S si et seulement si toute chaîne de nœuds de X à Y est « bloquée » par S. Pour trois nœuds x appartenant à X, y appartenant à Y et s appartenant à S, une chaîne est bloquée dans les deux cas suivants[6] :
- soit la chaîne contient une séquence ou ;
- soit la chaîne contient une séquence où z n'appartient pas à S et aucun descendant de z appartient à S.
L'intuition est que S « bloque » l'information entre X et Y.
L'indépendance conditionnelle enfin, notée ci-dessus , exprime que X est conditionnellement indépendant de Y étant donné S. Formellement :
Dans l'exemple donné plus haut (figure 1), on peut écrire que l'expérience de l'opérateur est indépendante de la complexité de la machine, mais qu'elle est conditionnellement dépendante de la complexité de la machine étant donné le risque d'accident[8].
En conclusion, un graphe orienté acyclique G = (V, E) est un réseau bayésien si et seulement s'il vérifie la propriété de Markov globale orientée étant donné une loi de probabilité P sur l'ensemble des variables V.
Inférence
Définition et complexité
L'inférence dans un réseau bayésien est le calcul des probabilités a posteriori dans le réseau, étant donné des nouvelles informations observées. Il s'agit d'un problème de calcul car, grâce aux opérations sur les probabilités et au théorème de Bayes, toutes les probabilités a posteriori possibles dans un réseau peuvent être calculées[9]. Ainsi, étant donné un ensemble d'évidences (de variables instanciées) Y, le problème de l'inférence dans G=(V, E) est de calculer avec [10],[11]. Si Y est vide (aucune évidence), cela revient à calculer P(X). Intuitivement, il s'agit de répondre à une question de probabilité sur le réseau.
Bien que le problème d'inférence dépende de la complexité du réseau (plus la topologie du réseau est simple, plus l'inférence est facile), il a été montré par G. F. Cooper en 1987 que dans le cas général, il s'agit d'un problème NP-difficile. Par ailleurs, Dagum et Luby ont également montré en 1993 que trouver une approximation d'un problème d'inférence dans un réseau bayésien est également NP-difficile. À la suite de ces résultats, deux grandes catégories d'algorithmes d'inférence viennent naturellement : les algorithmes d'inférence exacte, qui calculent les probabilités a posteriori généralement en temps exponentiel, et les heuristiques qui fournissent plutôt une approximation des distributions a posteriori, mais avec une complexité computationnelle moindre[12]. Plus rigoureusement, les algorithmes d'inférence exacte fournissent un résultat et la preuve que le résultat est exact, tandis que les heuristiques fournissent un résultat sans preuve de sa qualité (c'est-à-dire que selon les instances, une heuristique peut aussi bien trouver la solution exacte qu'une approximation très grossière).
La classification du problème d'inférence comme NP-difficile est donc un résultat primordial. Pour établir sa preuve, Cooper considère le problème général P(X=x)>0 : la probabilité que la variable X prenne la valeur x est-elle supérieure à zéro dans un réseau bayésien ? Il s'agit d'un problème de décision (la réponse est oui ou non). Cooper montre tout d'abord que si le problème P(X=x)>0 est NP-complet, alors P(X=x) est NP-difficile, et donc le problème de l'inférence dans les réseaux bayésiens est NP-difficile. Pour prouver la NP-complétude de P(X=x)>0, il réduit polynomialement le problème 3-SAT (un problème NP-complet classique) au problème P(X=x)>0 : premièrement, il montre que la construction d'un réseau bayésien à partir d'une instance de 3-SAT, notée C, peut être faite avec une complexité polynomiale ; ensuite, il montre que C est satisfaite si et seulement si P(X=x)>0 est vrai. Il en résulte que P(X=x)>0 est NP-complet[11].
Apprentissage automatique
L'apprentissage automatique désigne des méthodes ayant pour but d'extraire de l'information contenue dans des données. Il s'agit de processus d'intelligence artificielle, car ils permettent à un système d'évoluer de façon autonome à partir de données empiriques. Dans les réseaux bayésiens, l'apprentissage automatique peut être utilisé de deux façons : pour estimer la structure d'un réseau, ou pour estimer les tables de probabilités d'un réseau, dans les deux cas à partir de données.
Apprentissage de la structure
Il existe deux grandes familles d'approches pour apprendre la structure d'un réseau bayésien à partir de données :
- Les approches basées sur les contraintes. Le principe en est de tester les indépendances conditionnelles, et de chercher une structure de réseau cohérente avec les dépendances et indépendances observées.
- Les approches utilisant une fonction de score. Dans ces approches, un score est associé à chaque réseau candidat, mesurant l'adéquation des (in)dépendances encodées dans le réseau avec les données. On cherche alors un réseau maximisant ce score.
Variantes
Réseau bayésien dynamique
Un réseau bayésien dynamique ou temporel (souvent noté RBN, ou DBN pour Dynamic Bayesian Network) est une extension d'un réseau bayésien qui permet de représenter l'évolution des variables aléatoires en fonction d'une séquence discrète, par exemple des pas temporels[13]. Le terme dynamique caractérise le système modélisé, et non le réseau qui lui ne change pas. Par exemple, partant du réseau donné en figure 1, le système pourrait être rendu dynamique en exprimant que le risque futur d'accident dépend du risque d'accident passé et présent. L'intuition est que plus le nombre d'utilisateurs peu expérimentés se succèdent, plus le risque d'accident augmente ; au contraire, une succession d'utilisateurs expérimentés fait décroitre ce risque dans le temps. Dans cet exemple, la variable « accident » est dite dynamique, temporelle ou persistante.
Formellement, un réseau bayésien dynamique se définit comme un couple . est un réseau bayésien classique représentant la distribution a priori (ou initiale) des variables aléatoires ; dit plus intuitivement, il s'agit du temps 0. est un réseau bayésien dynamique a deux pas de temps décrivant la transition du pas de temps t-1 au pas de temps t, c'est-à-dire pour tout nœud appartenant à , dans un graphe orienté acyclique comme introduit plus haut. La probabilité jointe d'un pas de temps s'écrit alors[14],[15] :
Les parents d'un nœud, notés pour mémoire , peuvent ainsi être soit un parent direct dans le réseau au temps t, soit un parent direct au temps t-1.
La loi de probabilité jointe factorisée se calcule en « déroulant » le réseau sur la séquence temporelle, à condition de connaître sa longueur, que l'on va noter ici . Formellement, si est la probabilité jointe du réseau initial , donc au pas de temps 0, on peut écrire[14],[15] :
Un réseau bayésien dynamique respecte ainsi la propriété de Markov, qui exprime que les distributions conditionnelles au temps t ne dépendent que de l'état au temps t-1 dans un processus stochastique. Les réseaux bayésiens dynamiques sont une généralisation des modèles probabilistes de séries temporelles de type modèle de Markov caché, filtre de Kalman[15]...
Classifieur bayésien naïf
Un classifieur bayésien naïf est un type de classifieur linéaire qui peut au contraire être défini comme une simplification des réseaux bayésiens. Leur structure est en effet composée de seulement deux niveaux : un premier ne comportant qu'un seul nœud, noté par exemple , et le second plusieurs nœuds ayant pour seul parent . Ces modèles sont dits naïfs car ils font l'hypothèse que tous les fils sont indépendants entre eux. Soit les fils de notés , la loi de probabilité jointe d'un classifieur bayésien s'écrit[16]:
Ces modèles sont particulièrement adaptés pour les problèmes de classification automatique, où représente les classes possibles non observées d'un domaine et des variables observées caractérisant chaque classe de [16]. Un exemple serait de trier dans une population les individus en deux groupes ou classes, « sains » et « malades », en fonction de symptômes observés, comme la présence de fièvre, etc.
Diagramme causal
Un diagramme causal est un réseau bayésien dans lequel les liens entre les nœuds représentent des relations de causalité. Les diagrammes causaux permettent de faire de l'inférence causale[17].
Outils logiciels
- Logiciels
- Bayesia (commercial)
- Elvira (open source)
- GeNie (gratuit et code propriétaire ; GeNie est une interface graphique pour la bibliothèque SMILE, également gratuite et propriétaire)
- Hugin (commercial)
- Netica (commercial)
- OpenBUGS (open source)
- ProBayes (commercial)
- IBM SPSS (commercial)
- WinBUGS (gratuit)
- Weka (gratuit et open source)
- Bibliothèques
- Bayesian Network tools in Java (open source, bibliothèque pour JAVA)
- bnlearn et deal (open source, bibliothèques pour l'apprentissage sous R)
- Bayes Net Toolbox pour Matlab (code libre pour l'environnement commercial Matlab)
- Structure Learning Package (package pour l'apprentissage de structure en supplément du Bayes Net Toolbox)
- SMILE (gratuit et code propriétaire, C++ ou JAVA)
- Visual Numerics (bibliothèque pour IMSL)
- aGrUM/pyAgrum : modélisation, inférence , apprentissage, analyse causale de réseaux Bayesiens et extensions (gratuit et opensource c++ et python)
Annexes
Articles connexes
Bibliographie
- Patrick Naïm, Pierre-Henri Wuillemin, Philippe Leray, Olivier Pourret et Anna Becker, Réseaux bayésiens, Eyrolles, , 3e éd., 424 p. (ISBN 978-2-212-04723-3, lire en ligne)
- (en) Adnan Darwiche, Modeling and Reasoning with Bayesian Networks, Cambridge University Press, , 548 p. (ISBN 978-0-521-88438-9, lire en ligne)
- (en) Uffe B. Kjaerulff et Anders L. Madsen, Bayesian Networks and Influence Diagrams : A Guide to Construction and Analysis, New York, Springer, , 318 p. (ISBN 978-0-387-74100-0, lire en ligne)
- (en) Timo Koski et John Noble, Bayesian Networks : An Introduction, John Wiley & Sons, , 2e éd., 366 p. (ISBN 978-1-119-96495-7, lire en ligne)
- (en) Richard E. Neapolitan, Learning Bayesian networks, Pearson Prentice Hall, (ISBN 978-0-13-012534-7, lire en ligne)
- (en) Judea Pearl, Probabilistic Reasoning in Intelligent Systems : Networks of Plausible Inference, Morgan Kaufmann, , 552 p. (ISBN 978-1-55860-479-7, lire en ligne)
- Stuart Jonathan Russell et Peter Norvig (trad. de l'anglais par Fabrice Popineau), Intelligence artificielle, Paris, Pearson Education France, , 3e éd., 1198 p. (ISBN 978-2-7440-7455-4, lire en ligne)
Références
- Lepage, E., Fieschi, M., Traineau, R., Gouvernet, J., & Chastang, C. (1992). Système d'aide à la décision fondé sur un modèle de réseau bayesien application à la surveillance transfusionnelle. Nouvelles Méthodes de Traitement de l’Information en Médecine, 5, 76-87.
- Jean-Jacques Boreux, Éric Parent et Jacques Bernier, Pratique du calcul bayésien, Paris/Berlin/Heidelberg etc., Springer, , 333 p. (ISBN 978-2-287-99666-5, lire en ligne), p. 38-39
- Naïm et al. 2011, p. 90-91
- (en) Judea Pearl, « Bayesian Networks: A Model of Self-Activated Memory for Evidential Reasoning », Proceedings of the 7th Conference of the Cognitive Science Society, , p. 329–334 (lire en ligne)
- Naïm et al. 2011, p. 86-87
- David Bellot, Fusion de données avec des réseaux bayésiens pour la modélisation des systèmes dynamiques et son application en télémédecine, université Nancy-I, (lire en ligne), p. 68 (thèse)
- Naïm et al. 2011, p. 354-356
- Russell et Norvig 2010, p. 532
- Naïm et al. 2011, p. 93-94
- (en) Nevin Lianwen Zhang, « A simple approach to Bayesian network computations », Proceedings of the 10th Canadian conference on artificial intelligence, (lire en ligne)
- (en) Gregory F. Cooper, « Probabilistic Inference Using Belief Networks Is NP-Hard », Knowledge Systems Laboratory (report), (lire en ligne) ; (en) Gregory F. Cooper, « The computational complexity of probabilistic inference using Bayesian belief networks », Artificial Intelligence, vol. 42, nos 2-3, , p. 393-405 (lire en ligne)
- Kjaerulff et Madsen 2007, p. 9
- (en) Thomas Dean et Keiji Kanazawa, « A model for reasoning about persistence and causation », Computational Intelligence, vol. 5, no 2, , p. 142-150 (lire en ligne)
- (en) Kevin Patrick Murphy, Dynamic Bayesian Networks : Representation, Inference and Learning, université de Californie à Berkeley (lire en ligne), p. 14-15 (thèse)
- Roland Donat, Philippe Leray, Laurent Bouillaut et Patrice Aknin, « Réseaux bayésiens dynamiques pour la représentation de modèles de durée en temps discret », Journées francophone sur les réseaux bayésiens, (lire en ligne)
- (en) Nir Friedman et Moises Goldszmidt, « Building Classifiers using Bayesian Networks », Proceedings of the thirteenth national conference on artificial intelligence, (lire en ligne)
- (en) Judea Pearl, The Book of Why : The New Science of Cause and Effect, Penguin Books, , 432 p. (ISBN 978-0-14-198241-0 et 0141982411).
- Portail des probabilités et de la statistique
- Portail de l'informatique théorique