Sous-fonctionnalisation (évolution)
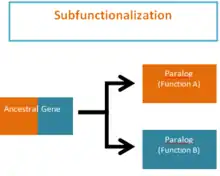
La sous-fonctionnalisation (ou subfonctionnalisation) est un mécanisme, proposé par Stoltzfus en 1999[1] ainsi que Force et ses collaborateurs la même année[2], constituant l'une des issues possibles de la divergence fonctionnelle ayant lieu à la suite d'un événement de duplication génique, pendant lequel une paire de gènes provenant d'une duplication (paralogues), prennent en charge des fonctions distinctes[3],[4]. La sous-fonctionnalisation est un processus mutationnel neutre, ce qui signifie qu'aucune adaptation nouvelle n'est formée[5],[4]. Lors du processus de duplication génique, les paralogues subissent simplement une "division du travail" en conservant différentes parties (sous-fonctions) de la fonction ancestrale originelle[6]. Cet événement de séparation a lieu en raison de l'extinction génique segmentaire conduisant à la formation de paralogues qui ne sont plus des duplicons, car chaque gène conserve seulement une fonction[4]. Il est important de noter que le gène ancestral est à la base capable de prendre en charge les deux fonctions, et les gènes dupliqués descendants ne peuvent désormais prendre en charge qu'une des fonctions ancestrales originelles[4].
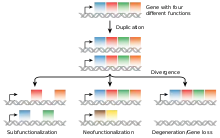
Hypothèse alternative
On pense que la sous-fonctionnalisation faisant suite à la duplication génique constitue l'exemple le plus récemment découvert de divergence fonctionnelle[7]. Avant 1910, les chercheurs ne savaient pas que les gènes étaient capables de multifonctionnalisation[4]. On pensait au départ que chaque gène pouvait prendre en charge une fonction, alors qu'en réalité les gènes possèdent des régions mutables indépendantes les unes des autres, ainsi que la capacité à se sous-fonctionnaliser[4],[8]. On pense que la néo-fonctionnalisation, dans laquelle l'une des copies des paralogues dérive une nouvelle fonction à la suite d'une duplication génique, constitue un modèle classique de divergence fonctionnelle. Néanmoins, en raison de son processus mutationnel neutre, la sous-fonctionnalisation semble présenter une explication plus parcimonieuse pour la rétention de duplicons au sein d'un génome[3],[9],[10].
Spécialisation
La spécialisation est un modèle unique de sous-fonctionnalisation, dans laquelle les paralogues se divisent en plusieurs zones spécialisées plutôt qu'en une fonction chacun. Dans ce modèle, les deux copies géniques prennent en charge exactement la même fonction ancestrale. Par exemple, alors que le gène ancestral pouvait réaliser sa fonction dans tous les tissus, stades de développement et conditions environnementales, les gènes paralogues deviennent eux spécialistes, se divisant entre les différents tissus, stades de développement et conditions environnementales[11]. Par exemple, si le gène ancestral est responsable des processus régulatoires à la fois dans les systèmes digestif et lymphatique, après la duplication génique l'un des paralogues revendiquerait la responsabilité de la régulation lymphatique et l'autre celle de la régulation digestive. La spécialisation est également unique dans le sens où c'est un processus mutatif positif plutôt que neutre[4]. Lorsqu'un gène se spécialise parmi entre différents tissus, stades de développement ou conditions environnementales, il acquiert une amélioration dans sa fonction. Les isozymes en sont un bon exemple car ce sont des produits de gènes paralogues qui catalysent la même réaction biochimique. Cependant, différents membres ont acquis au cours de l'évolution des adaptations particulières aux différents tissus ou à différents stades de développement, qui améliorent la mise au point physiologique de la cellule[4].
Partage de gènes
Le partage de gènes (gene sharing en anglais) a lieu lorsqu'un gène acquiert une fonction secondaire au cours du processus évolutif. Ce partage de gènes est unique car le gène conserve et prend en charge à la fois sa fonction ancestrale et sa fonction acquise. La duplication génique n'est pas nécessaire dans ce modèle, car l'apparition d'une nouvelle fonction peut avoir lieu avant, ou plus souvent pendant celle-ci. Ce partage est un phénomène assez courant, le plus souvent observé chez les enzymes réalisant plusieurs sous-fonctions telles que la transduction du signal et la régulation transcriptionnelle[4]. L'exemple le plus notable de partage génique est qu'on a trouvé que les cristallines (les protéines responsables de la transparence et de la diffraction dans la lentille oculaire, au sein du cristallin) sont également capables de servir d'enzymes métaboliques dans un autre tissu[4].
S'échapper du conflit adaptatif
Le conflit adaptatif a lieu lors du partage de gènes quand l'amélioration d'une fonction d'un gène altère gravement une autre fonction. Cela a lieu car les contraintes sélectives sont particulièrement rigoureuses dans le cas du partage génique[4]. Il est très difficile pour une autre fonction de subir des changements morphologiques, dû au fait qu'à la fois la fonction ancestrale et la nouvelle fonction sont nécessaires. En conséquence de cette double fonction, le gène est sujet à deux ou plusieurs ensembles indépendants de pression de sélection[4]. Cela signifie que la sélection positive des améliorations pour une fonction aura tendance à causer un effet préjudiciable pour l'autre fonction. Il y a deux solutions pour résoudre la situation délicate du conflit adaptatif : le gène peut soit complètement perdre sa nouvelle fonction, soit subir une duplication génique suivie d'une sous-fonctionnalisation[4], ce qu'on appelle aussi "division de fonction" (function splitting en anglais)[12].
Duplication - Dégénérescence - Complémentation
Dans le modèle Duplication - Dégénérescence - Complémentation (DDC) de la sous-fonctionnalisation, les deux copies du gène ancestral sont nécessaires pour prendre en charge la fonction du gène ancestral originel[7]. Dans ce modèle, après un événement de duplication, les deux paralogues subissent des mutations délétères menant à une dégradation fonctionnelle. Cette dégradation est si importante qu'aucune copie génique ne peut réaliser la fonction ancestrale ni aucun des sous-ensembles de cette fonction indépendamment. Afin d'être fonctionnels, les paralogues doivent fonctionner ensemble afin de prendre en charge la fonction ancestrale. Ce travail d'équipe au sein des paralogues est possible car la sous-fonction perdue dans un duplicon est complémentée dans l'autre duplicon[4]. Ce partage fonctionnel ne serait pas possible si les deux paralogues avaient perdu des sous-fonctions identiques. Les processus de dégénération et de complémentation font du modèle DDC un processus de mutation sélectivement neutre. Les mutations accumulées dans les deux paralogues auraient été délétères si elles n'avaient pas été complémentées par l'autre copie[4]. Un exemple du modèle DDC est lorsque des paralogues fonctionnellement similaires sont exprimés à des niveaux si faibles que les deux copies sont nécessaires pour produire des quantités suffisantes du produit génique originel[4].
Prévention de ségrégation
La prévention de ségrégation (segregation avoidance en anglais) a lieu lorsqu'un crossing-over inégal amène une duplication de locus contenant deux allèles hétérogènes créant une situation comparable à une hétérozygotie permanente[4]. Cela a lieu principalement dans des situations de sélection surdominante où l'hétérozygote a augmenté la fitness mais où des homozygotes moins complémentaires sont encore maintenus au sein de la population[4]. La prévention de ségrégation aborde la question de la charge ségrégationnelle, où la fitness moyenne de la population est inférieure à la fitness la plus forte possible. Le crossing-over inégal et la duplication conséquente d'un locus contenant des allèles hétérogènes assure la fitness la plus forte possible. En évitant les allèles homogènes, les organismes d'une population peuvent bénéficier des avantages que les deux allèles peuvent leur fournir. Un excellent exemple est le locus ace-1 chez le moustique domestique Culex pipiens[13]. À cause de la prévention de ségrégation, ce moustique est capable de bénéficier de l'allèle ace-1R conférant une résistance lorsqu'il est exposé à un pesticide, ainsi que de l'allèle ace-1S sauvage lorsqu'il n'y a pas d'exposition à un pesticide[4]. Cette dualité est particulièrement utile car l'allèle mutant diminue la fitness lors des périodes de non-exposition au pesticide[13].
Exemple de l'hémoglobine
L'hémoglobine humaine fournit une certaine variété d'exemples de sous-fonctionnalisation. Par exemple, le gène codant la chaîne a de l'hémoglobine est sans aucun doute dérivé d'un duplicon de la chaîne ß[4]. Cependant, aucune des deux chaînes ne peut fonctionner indépendamment pour former une molécule monomérique d'hémoglobine, c'est-à-dire une molécule composée entièrement de chaînes a ou de chaînes ß. Réciproquement, l'hémoglobine est composée des deux types de chaînes, avec l'enchaînement a2-ß2 étant parmi les formes les plus efficaces de l'hémoglobine dans le génome humain. Cela constitue un des premiers exemples de sous-fonctionnalisation. Un autre bon exemple est l'émergence de l'hémoglobine fœtale à partir de l'hémoglobine embryonnaire à la suite de la duplication de la chaîne ?. Cet exemple de sous-fonctionnalisation illustre la façon dont les différentes formes d'hémoglobine sont présentes à divers stades de développement. En réalité, on retrouve une forme d'hémoglobine distincte à chaque stade de développement : ?2-e2 et a2-e2 au stade embryon, a2-?2 au stade fœtus, et a2-ß2 et a2-d2 au stade adulte[4]. Chaque type d'hémoglobine confère des avantages particuliers adaptés au stade de développement dans lequel elle se développe. Par exemple les hémoglobines fœtale et embryonnaire ont une affinité plus grande à l'oxygène que l'hémoglobine adulte, ce qui leur donne une fonctionnalité améliorée dans des environnements hypoxiques comme l'est l'utérus[4].
Notes et références
- (en) Arlin Stoltzfus, « On the Possibility of Constructive Neutral Evolution », Journal of Molecular Evolution, vol. 49, no 2, , p. 169–181 (ISSN 0022-2844 et 1432-1432, DOI 10.1007/PL00006540, lire en ligne, consulté le )
- (en) Allan Force, Michael Lynch, F. Bryan Pickett et Angel Amores, « Preservation of Duplicate Genes by Complementary, Degenerative Mutations », Genetics, vol. 151, no 4, , p. 1531–1545 (ISSN 0016-6731 et 1943-2631, PMID 10101175, lire en ligne, consulté le )
- Shruti Rastogi et David A. Liberles, « Subfunctionalization of duplicated genes as a transition state to neofunctionalization », BMC Evolutionary Biology, vol. 5, , p. 28 (ISSN 1471-2148, DOI 10.1186/1471-2148-5-28, lire en ligne, consulté le )
- Bernard Conrad et Stylianos E. Antonarakis, « Gene Duplication: A Drive for Phenotypic Diversity and Cause of Human Disease », Annual Review of Genomics and Human Genetics, vol. 8, no 1, , p. 17–35 (PMID 17386002, DOI 10.1146/annurev.genom.8.021307.110233, lire en ligne, consulté le )
- (en) Susumu Ohno, Evolution by Gene Duplication, Springer, (lire en ligne), p. 59-87
- (en) Marie Sémon et Kenneth H. Wolfe, « Preferential subfunctionalization of slow-evolving genes after allopolyploidization in Xenopus laevis », Proceedings of the National Academy of Sciences, vol. 105, no 24, , p. 8333–8338 (ISSN 0027-8424 et 1091-6490, PMID 18541921, DOI 10.1073/pnas.0708705105, lire en ligne, consulté le )
- Riet De Smet et Yves Van de Peer, « Redundancy and rewiring of genetic networks following genome-wide duplication events », Current Opinion in Plant Biology, genome studies molecular genetics, vol. 15, no 2, , p. 168–176 (DOI 10.1016/j.pbi.2012.01.003, lire en ligne, consulté le )
- (en) J. Graham Ruby, Alexander Stark, Wendy K. Johnston et Manolis Kellis, « Evolution, biogenesis, expression, and target predictions of a substantially expanded set of Drosophila microRNAs », Genome Research, vol. 17, no 12, , p. 1850–1864 (ISSN 1088-9051 et 1549-5469, PMID 17989254, DOI 10.1101/gr.6597907, lire en ligne, consulté le )
- (en) Adam Eyre-walker, « Fundamentals of Molecular Evolution (2nd edn) », Heredity, vol. 84, no 6, , p. 735–735 (ISSN 0018-067X, DOI 10.1046/j.1365-2540.2000.0728d.x, lire en ligne, consulté le )
- (en) Grigoris D. Amoutzias, Ying He, Jonathan Gordon et Dimitris Mossialos, « Posttranslational regulation impacts the fate of duplicated genes », Proceedings of the National Academy of Sciences, vol. 107, no 7, , p. 2967–2971 (ISSN 0027-8424 et 1091-6490, PMID 20080574, DOI 10.1073/pnas.0911603107, lire en ligne, consulté le )
- (en) Hideki Innan, « Population genetic models of duplicated genes », Genetica, vol. 137, no 1, , p. 19 (ISSN 0016-6707 et 1573-6857, DOI 10.1007/s10709-009-9355-1, lire en ligne, consulté le )
- (en) Lee Altenberg, Evolution and Biocomputation, Berlin/Heidelberg/New York, Springer, Berlin, Heidelberg, coll. « Lecture Notes in Computer Science », , 277 p. (ISBN 3-540-59046-3, DOI 10.1007/3-540-59046-3_11, lire en ligne), p. 205–259
- (en) Austin L. Hughes, Adaptive Evolution of Genes and Genomes, Oxford University Press, , 270 p. (ISBN 978-0-19-511626-7, lire en ligne)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Subfunctionalization » (voir la liste des auteurs).
Voir aussi
Articles connexes
- Portail origine et évolution du vivant
- Portail de la biologie