علم التشخيص الجزيئي
علم التشخيص الجزيئي (أ) هو مجموعة من التقنيات المستخدمة لتحليل الواصمات الحيوية في الجينوم والبروتيوم - الشيفرة الوراثية للفرد وكيف تعبّر الخلايا عن جيناتها كبروتينات - من خلال تطبيق البيولوجيا الجزيئية على الاختبارات الطبية. تستخدم هذه التقنية لتشخيص ومراقبة المرض، والكشف عن المخاطر، وتحديد نوع العلاج الأمثل حسب الحالة الفردية للمريض.[1][2]
علم التشخيص الجزيئي
|

من خلال تحليل تفاصيل المريض ومرضه، يوفر التشخيص الجزيئي الأساس لاحتمال نجاح مبدأ الطب الشخصي.[3]
هذه الاختبارات مفيدة في مجموعة من التخصصات الطبية، بما في ذلك الأمراض المعدية، وعلم الأورام، ومستضد الكريات البيضاء البشرية (التي تحقق وتتكهن بالوظيفة المناعية)، والتخثر، وعلم الصيدلة الجيني- التنبؤ الجيني الذي ستعمل به الأدوية على أفضل وجه.[4] كما أنها تتداخل مع الكيمياء السريرية (اختبارات طبية على سوائل الجسم).
التاريخ
نما مجال البيولوجيا الجزيئية في أواخر القرن العشرين، وكذلك تطبيقه السريري. في عام 1980، اقترح Yuet Wai Kan وآخرون في مجموعة بحثه اختبارًا وراثيًا سابقًا للثلاسيميا لم يعتمد على اختبار تسلسل الحمض النووي في مرحلة الطفولة، ولكن على إنزيمات الاقتطاع التي تقطع سلسلة الحمض النووي عند التعرّف على تسلسلات قصيرة محددة، مما خلق أطوالًا مختلفة من شريط حمض نووي ريبوزي منقوص الأكسجين حسب نوع الأليل (التنوع الجيني) الذي يمتلكه الجنين.[5] في الثمانينات، تم استخدام العبارة في أسماء شركات مثل Molecular Diagnostics Incorporated [6] وBethseda Research Laboraties Molecular Diagnostics.[7][8]
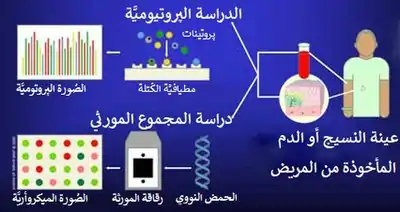
خلال التسعينيات، أدى تحديد الجينات المكتشفة حديثًا والتقنيات الجديدة لتسلسل الحمض النووي إلى ظهور مجال متميز من الطب المخبري الجزيئي والجينومي. في عام 1995، تم تشكيل جمعية علم الأمراض الجزيئية (AMP)؛ وفي عام 1999، شاركت جمعية علم الأمراض الجزيئية في تأسيس مجلة علم التشخيص الطبي The Journal of Medical Diagnostics.[9] أطلقت شركة إنفورما للرعاية الصحية خدمة تعليقات الخبراء في تلك الدورية العلمية في عام 2001.[1] منذ عام 2002 فصاعدا، قام مشروع HapMap بتجميع المعلومات عن اختلافات الحرف الواحد الجينية والتي تتكرر بين البشر - تعدد الأشكال النوكليوتيدية الوحيدة - وعلاقتها بالمرض.[2] في عام 2012، تم استخدام تقنيات التشخيص الجزيئي للثلاسيميا اختبارات التهجين الوراثي لتحديد تعدد أشكال النوكليوتيدات المفرد الذي يسبب مرضاً للفرد.[10]
كما أصبح التطبيق التجاري لعلم التشخيص الجزيئي أكثر أهمية، وكذلك النقاش حول براءات الاختراع للاكتشافات الجينية في صميمها. في عام 1998، صدر التوجيه الخاص بالاتحاد الأوروبي،(ب) الذي سمح ببراءات الاختراع على تسلسلات الحمض النووي.[11] في عام 2010، في الولايات المتحدة، رفع AMP دعوى قضائية ضد شركة مايرياد جينيتكس للطعن في براءات الاختراع الأخيرة بشأن جينات، BRCA1 BRCA2، والتي ترتبط بسرطان الثدي. في عام 2013، وافقت المحكمة العليا في الولايات المتحدة، جزئيًا، على الحكم بأنه لا يمكن تسجيل براءة اختراع على تسلسل جيني يحدث بشكل طبيعي.[12][13]
التقنيات

جعلت عملية تصنيع أدوات اختبار البيولوجيا الجزيئية استخدامها في العيادات عمليا؛ [2] كما أن التصغير في جهاز واحد محمول يمكن أن يجلب التشخيص الطبي إلى العيادة وإلى المكتب أو المنزل.[2] يتطلب المختبر السريري معايير عالية من الموثوقية؛ بالإضافة إلى ذلك قد تتطلب التشخيصات اعتمادًا من هيئات موثوقة أو أن تكون مدرجة تحت لوائح الأجهزة الطبية.[14] لذلك واعتبارًا من عام 2011، استخدمت بعض المختبرات الطبية الأمريكية فحوصًا تم بيعها «للاستخدام البحثي فقط».[15] يجب أن تلتزم العمليات المخبرية باللوائح الناظمة، من الأمثلة على ذلك في الولايات المتحدة: «تعديلات تحسين المختبرات السريرية» (CLIA) و«قانون قابلية التأمين الصحي وقابلية التأمين» (HIPAA) و«الممارسات المختبرية الجيدة» (GLP) ومواصفات «إدارة الأغذية والأدوية» (FDA). تساعد أنظمة إدارة المعلومات المخبرية في ذلك من خلال تتبع تلك العمليات؛[16] كما يمكن لعملية الأتمتة ومسح العينات بالباركود أن يوسع من الإنتاجية وأن يقلل من إمكانية الخطأ أو التلوث أثناء المعالجة اليدوية وإصدار تقارير عن النتائج. تتوفر الآن أجهزة مفردة لإجراء الفحص من البداية إلى النهاية.[14]
يستخدم علم التشخيص الجزيئي مقايسات بيولوجية في المختبر مثل PCR-ELISA أو التهجين الموضعي المتألق.[17][18] يكشف فحص المقايسة جزيئًا، غالبًا بتركيزات منخفضة، والذي هو مؤشر لمرض أو خطر في عينة مأخوذة من مريض. الحفاظ على العينة قبل التحليل أمر بالغ الأهمية؛ حيث يجب تقليل التعامل اليدوي.[19] كما يطرح جزيء الـ RNA الهش تحديات معينة: فكجزء من العملية الخلوية للتعبير عن الجينات كبروتينات، فإنها تقدم مقياسًا للتعبير الجيني ولكنها عرضة للتحلل المائي وللتجزئة من خلال إنزيمات RNAse دائمة الوجود. يمكن أن تكون العينات مجمدة في النتروجين السائل أو محتضنة في عوامل حافظة.[2]
نظرًا لأن أساليب التحليل الجزيئي يمكنها اكتشاف الواسمات الحساسة، فإن هذه الاختبارات أقل تطفلاً من تلك التقليدية، مثل أخذ خزعة. فعلى سبيل المثال، بسبب وجود الأحماض النووية الخالية من الخلايا في البلازما البشرية، يمكن أن تكون عينة دم بسيطة كافية لاستحصال المعلومات الوراثية من الأورام أو الأعضاء المزروعة أو من الأجنة.[2] إن معظم طرق علم التشخيص الجزيئي المعتمدة على الأحماض النووية تستخدم تفاعل البلمرة المتسلسل (PCR) لزيادة عدد جزيئات الحمض النووي إلى حد كبير، وبالتالي تضخّم التسلسل الهدف في عينة المريض.[2] يعتمد مبدأ PCR على تضخيم قالب حمض نووي ريبوزي منقوص الأكسجين باستخدام البادئات الاصطناعية (برايمر)، وبوليميراز الحمض النووي، وdNTPs. يتم تدوير الخليط بين درجتي حرارة متباينتين على الأقل: درجة حرارة عالية من أجل تغيير شكل الدنا ذي اللولب المزدوج إلى جزيئات أحادية الجديلة؛ ودرجة حرارة منخفضة من أجل التمهيد للتهجين إلى القالب ولتمكين إنزيم البوليميراز من تطويل وتضخيم البادئ (البرايمر). تعمل كل دورة درجة حرارة نظرياً على مضاعفة كمية التسلسل الهدف. إن الكشف عن الاختلافات في التسلسل باستخدام PCR يتضمن غالباً تصميم واستخدام كواشف قليل النوكليوتيد التي تعمل على تضخيم المتغيّر المطلوب، مما يعطي نتيجة أكثر كفاءة من مجرّد إجراء عملية تضخيم غير منضبطة للتسلسل. PCR هو حاليا الطريقة الأكثر استخداما للكشف عن تسلسل الحمض النووي.[20] قد يؤدي اكتشاف الوسم إلى استخدام PCR في الوقت الفعلي، والتسلسل المباشر،[2] ورقائق ميكروأراي، وهي رقائق مسبقة الصنع تختبر العديد من الواسمات في وقت واحد، [2] أو MALDI-TOF[21] نفس الشيء ينطبق مبدأ على البروتين والجينوم. يمكن لصفائف البروتين عالية الإنتاجية استخدام الحمض النووي المتمم أو الأجسام المضادة للارتباط، وبالتالي يمكن الكشف عن العديد من البروتينات في نفس الوقت.[22] تختلف الاختبارات التشخيصية الجزيئية اختلافًا كبيرًا في الحساسية، ووقت الإنجاز، والتكلفة، والتغطية، والموافقة التشريعية. كما تختلف في مستوى المصادقة المطبقة في المختبرات التي تستخدمها. وبالتالي، فإن التحقق المحلي القوي وفقًا للمتطلبات التنظيمية واستخدام الضوابط المناسبة مطلوب خاصةً عند استخدام النتيجة لإبلاغ قرار علاج المريض.[23]
التطبيقات
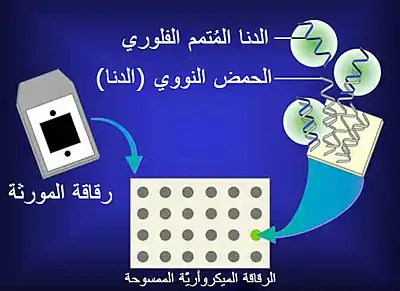
قبل الولادة
تعتمد الاختبارات التقليدية السابقة للولادة للكشف عن تشوهات الكروموسومات مثل متلازمة داون على تحليل عدد الكروموسومات ومظهرها - أي على النمط النووي. الاختبارات التشخيصية الجزيئية مثل اختبار ميكروأراي التهجين الجينومي المقارن تعمل على اختبار عينة من الحمض النووي بدلا من ذلك. وبسبب الحمض النووي الخالي من الخلايا في البلازما، يمكن أن تكون العمليات أكثر سلاسة، ولكن لا يزال (سنة 2013) ذلك بمثابة ملحق للاختبارات التقليدية.[24]
العلاج
يمكن لبعض أشكال تعدد الأشكال النوكليوتيدية الوحيدة للمريض - وهي اختلافات طفيفة في حمضه النووي - أن تساعد في التنبؤ بمدى سرعة أيضها لعقاقير معينة؛ وهذا ما يسمى علم الصيدلة الجيني.[25] على سبيل المثال، يُوَيِّض الإنزيم CYP2C19 العديد من العقاقير، مثل عقار كلوبيدوغريل المضاد للتخثر، إلى أشكاله النشطة. يكون تعدد الأشكال لبعض المرضى في أماكن محددة على جين 2C19، الأمر الذي يجعل أيض تلك العقاقير ضعيفاً؛ يمكن للأطباء اختبار هذه الأشكال المتعددة ومعرفة ما إذا كانت الأدوية فعالة بشكل كامل لهذا المريض.[26] وقد ساعد التقدم في علم الأحياء الجزيئي في إظهار أن بعض المتلازمات التي كانت تُصنف سابقًا كمرض واحد هي في الواقع أنواع فرعية متعددة ذات أسباب وعلاجات مختلفة تمامًا. يمكن أن تساعد التشخيصات الجزيئية في تشخيص النوع الفرعي - على سبيل المثال، للإصابات والأمراض السرطانية - أو التحليل الجيني لمرض ذي مكون موروث، مثل متلازمة سيلفر-روسل.[1][27]
الأمراض المعدية
يستخدم علم التشخيص الجزيئي لتحديد الأمراض المعدية مثل الكلاميديا، [28] وفيروس الإنفلونزا [29] والسل، [30] أو سلالات محددة مثل فيروس الإنفلونزا أ H1N1.[31] يمكن أن يكون التعرف الجيني سريعًا. على سبيل المثال اختبار «تضخيم متساوي الحرارة بوساطة حلقة» (LAMP) يقوم بتشخيص طفيلي الملاريا، وهو متين بما فيه الكفاية بالنسبة للبلدان النامية.[32] لكن على الرغم من هذه التطورات في تحليل الجينوم، في عام 2013 كان لا تزال العدوى أكثر تحديدًا بوسائل أخرى - مثل مظهر البروتيوم، أو جراثيم العاثية، أو نتائج تحاليل الاستشراب.[33] كما يستخدم علم التشخيص الجزيئي لفهم سلالة معينة من مسببات الأمراض - على سبيل المثال من خلال الكشف عن الجينات المقاومة للعقاقير التي تمتلكها - وبالتالي العلاجات التي يجب تجنبها.[33]
إدارة مخاطر الأمراض
قد يشتمل جينوم المريض على طفرة موروثة أو عشوائية تؤثر على احتمال تطور مرض في المستقبل.[25] على سبيل المثال، تعتبر متلازمة لينش مرضًا وراثيًا يعرّض المرضى لسرطان القولون والمستقيم وغيره من أنواع السرطان. يمكن للاكتشاف المبكر أن يؤدي إلى المراقبة الدقيقة التي تحسن فرص المريض في الحصول على نتائج جيدة.[34] يشار إلى المخاطر القلبية الوعائية بواسطة واسمات بيولوجية ويمكن للفحص أن يقيس خطر ولادة طفل بمرض وراثي مثل التليف الكيسي.[35] يعتبر الاختبار الوراثي معقدًا أخلاقيًا: فقد لا يرغب المرضى في الضغط النفسي المصاحب عند معرفة المخاطر المحدقة بهم.[36] في البلدان التي لا توجد فيها رعاية صحية شاملة، قد تؤدي المخاطر المعروفة إلى زيادة أقساط التأمين.[37]
السرطان
السرطان هو تغير في العمليات الخلوية التي تتسبب في نمو ورم خارج عن السيطرة.[25] تحتوي الخلايا السرطانية في بعض الأحيان على طفرات في الجينات الورمية، مثل KRAS وCTNNB1 (cat-catenin).[38] إن تحليل التوقيع الجزيئي للخلايا السرطانية - الدنا ومستويات التعبير عبر الرنا المرسال - تمكّن الأطباء من وصف السرطان واختيار أفضل علاج لمرضاهم.[25] واعتبارًا من عام 2010، فإن المقايسات التي تتضمن مجموعة من الأجسام المضادة ضد جزيئات محددة من واسمات البروتين هي تكنولوجيا ناشئة؛ هناك آمال لهذه المقايسات المتعددة التي يمكن أن تقيس العديد من الوسوم في وقت واحد.[39] ومن الواسمات الحيوية المستقبلية المحتملة الأخرى جزيئات الحمض النووي الريبي الدقيقة، التي تعبر الخلايا السرطانية عنها أكثر من الخلايا السليمة.[40]
السرطان هو مرض ذو أسباب جزيئية مفرطة وتطور مستمر. هناك أيضًا عدم تجانس للمرض حتى في الفرد. أثبتت الدراسات الجزيئية للسرطان أهمية الطفرات القائدة في نمو وانبثاث الأورام.[41] وقد تم تطوير العديد من التقنيات للكشف عن اختلافات التسلسل لأبحاث السرطان. يمكن تجميع هذه التقنيات عمومًا في ثلاث طرق: تفاعل البوليميراز المتسلسل (PCR)، والتهجين، وتسلسل الجيل التالي NGS (NGS).[20] حاليا، تمت الموافقة على الكثير من مقايسات PCR والتهجين من قبل FDA كما في التشخيص المخبري.[42] لكن اختبارات NGS لا تزال في مرحلة مبكرة من التشخيص السريري.[43]
للقيام باختبار التشخيص الجزيئي للسرطان، واحدة من القضايا الهامة هي الكشف عن اختلاف تسلسل الحمض النووي. تحتوي عينات خزعات الورم المستخدمة للتشخيص دائمًا على 5٪ من المتغير المستهدف بالمقارنة مع التسلسل غير المستهدف؛ أي أنها متميزة عن النوع غير النمطي للطفرات. أيضًا، بالنسبة لأخذ عينات من الدم المحيطي أو البول، يجب أن يكون اختبار الحمض النووي محددًا بما يكفي لاكتشاف الطفرات عند ترددات الأليل المختلفة التي تقل عن 0.1٪.[20]
في الوقت الحالي، من خلال تحسين PCR التقليدي، هناك اختراع جديد، وهو «نظام طفرة التضخيم الحراري» (ARMS)، وهو طريقة للكشف عن متغيرات تسلسل الحمض النووي في السرطان. المبدأ الذي يكمن وراء هذه الطريقة هو أن نشاط تمديد الأنزيمات في إنزيم DNA بوليميراز هو حساس للغاية لعدم التطابق قرب نهاية 3 من البادئ (برايمر).[20] طورت العديد من الشركات المختلفة اختبارات تشخيصية مبنية على بادئات ARMS PCR. على سبيل المثال، قامت شركات كياجين ثيراسكرين Qiagen therascreen، [44] روش كوباس Roche cobas [45] وBiomerieux THxID [46] بتطوير اختبارات PCR المعتمدة من FDA للكشف عن سرطان الرئة، وسرطان القولون، وطفرات الورم الميلانيني الانبثاثي في جينات KRAS، EGFR وBRAF. تم التحقق من صحة مجموعات IVD الخاصة بهم بشكل أساسي على الحمض النووي الجيني المستخرج من أنسجة FFPE.
هناك أيضا وحدات ميكروأراي تستخدم آلية التهجين للقيام بتشخيص السرطان. يمكن تركيب أكثر من مليون من المجسات المختلفة على مصفوفة مع تقنية Genechip من Affymetrix مع حد كشف من 1-10 نسخ من mRNA لكل بئر (بئر: وحدة حيز من المكان على الشريحة).[20] تعتبر وحدات ميكروأراي الدقيقة المحسنة قادرة بالعادة على تقديم نتائج تحديد كمية قابلة للتكرار لأهداف مختلفة.[47] في الوقت الحالي، وافقت إدارة الأغذية والأدوية FDA بالفعل على عدد من فحوصات التشخيص التي تستخدم المصفوفات الدقيقة ميكروأراي: يمكن لمقايسات MammaPrint من Agendia أن تخبر خطر تكرار سرطان الثدي عن طريق تمييز التعبير عن 70 جينة مرتبطة بسرطان الثدي؛ [48] يمكن لفحص "Autogenomics INFNITI CYP2C19" توصيف الأشكال الجينية، التي تؤثر على الاستجابة العلاجية لمضادات الاكتئاب بشكل كبير؛ [49] ويمكن لـ CytoScan Dx من Affymetrix تقييم الإعاقات الذهنية والاضطرابات الخلقية عن طريق تحليل طفرة الكروموسومات.[50]
في المستقبل، من المرجح أن تركز أدوات التشخيص للسرطان على تسلسل الجيل التالي (NGS). من خلال استخدام تسلسل الحمض النووي الريبوزي DNA وRNA لإجراء تشخيص للسرطان، ستتطور التكنولوجيا في مجال أدوات التشخيص الجزيئي بشكل أفضل. على الرغم من أن إنتاجية وسعر NGS قد انخفضت بشكل كبير خلال السنوات العشر الماضية بنحو 100 ضعف، إلا أننا ما زلنا على مسافة 6 درجات على الأقل بعيدًا عن إجراء التسلسل العميق على مستوى الجينوم بأكمله.[51] حاليا، طورت Ion Torrent بعض لوحات NGS على أساس AmpliSeq متعدي، على سبيل المثال، الفحص الشامل Oncomine.[52] حيث يركزون على استخدام التسلسل العميق للجينات المتعلقة بالسرطان لاكتشاف المتغيرات التسلسلية النادرة.
يمكن استخدام أداة التشخيص الجزيئي لتقييم مخاطر السرطان. على سبيل المثال، يقيّم اختبار BRCA1 / 2 من قبل شركة ميرياد جينيتكس مخاطر إصابة النساء بسرطان الثدي مدى الحياة.[20] أيضًا، لا تعمل بعض أنواع السرطان دائمًا مع أعراض واضحة. من المفيد تحليل الناس عندما لا تظهر عليهم أعراض واضحة، وبالتالي يمكن الكشف عن السرطان في المراحل المبكرة. على سبيل المثال، يمكن استخدام اختبار ColoGuard لفحص الأشخاص الذين تزيد أعمارهم عن 55 عامًا لسرطان القولون والمستقيم.[53] السرطان هو مرض على المدى الطويل مع خطوات التقدم المختلفة، ويمكن استخدام أدوات التشخيص الجزيئي لتوقعات تطور السرطان. على سبيل المثال، اختبار OncoType Dx بواسطة Genomic Health يمكنه تقدير خطر الإصابة بسرطان الثدي. يمكن لتقنيتهم إبلاغ المرضى بالبحث عن العلاج الكيماوي عند الضرورة عن طريق فحص مستويات التعبير عن الرنا في نسيج خزعة الثدي.[54]
مع ارتفاع الدعم الحكومي في التشخيص الجزيئي للحمض النووي، من المتوقع أن يتوافر قريبًا عدد متزايد من فحوصات اكتشاف الحمض النووي السريري للسرطانات. في الوقت الحالي، يتطور البحث في تشخيص السرطان بسرعة مع أهداف بتكلفة أقل، واستهلاك أقل للوقت وأساليب أبسط للأطباء والمرضى.
هوامش
- أ. مصطلح علم التشخيص هو ترجمة diagnostics حسب المعجم الطبي الموحد.[55]
- ب. وهو التوجيه (98/44/EC)، صدر في الشهر السادس من عام 1998م، دخل حيز التنفيذ بعد عامين.[56]
المراجع
- Poste G (May 2001). "Molecular diagnostics: a powerful new component of the healthcare value chain". Expert Review of Molecular Diagnostics. 1 (1): 1–5. doi:10.1586/14737159.1.1.1. PMID 11901792.
- Burtis CA, Ashwood ER, Bruns DE (2012). Tietz Textbook of Clinical Chemistry and Molecular Diagnostics. Elsevier. ISBN 1-4557-5942-2.
- Hamburg MA, Collins FS (July 2010). "The path to personalized medicine". The New England Journal of Medicine. 363 (4): 301–4. doi:10.1056/NEJMp1006304. PMID 20551152.
- Grody WW, Nakamura RM, Strom CM, Kiechle, Frederick L. (2010). Molecular Diagnostics: Techniques and Applications for the Clinical Laboratory. Boston MA: Academic Press Inc. ISBN 978-0-12-369428-7.
- Kan YW, Lee KY, Furbetta M, Angius A, Cao A (January 1980). "Polymorphism of DNA sequence in the beta-globin gene region. Application to prenatal diagnosis of beta 0 thalassemia in Sardinia". The New England Journal of Medicine. 302 (4): 185–8. doi:10.1056/NEJM198001243020401. PMID 6927915.
- Cohn DV, Elting JJ, Frick M, Elde R (June 1984). "Selective localization of the parathyroid secretory protein-I/adrenal medulla chromogranin A protein family in a wide variety of endocrine cells of the rat". Endocrinology. 114 (6): 1963–74. doi:10.1210/endo-114-6-1963. PMID 6233131.
- Persselin JE, Stevens RH (August 1985). "Anti-Fab antibodies in humans. Predominance of minor immunoglobulin G subclasses in rheumatoid arthritis". The Journal of Clinical Investigation. 76 (2): 723–30. doi:10.1172/JCI112027. PMC 423887 Freely accessible. PMID 3928684.
- Kaplan G, Gaudernack G (October 1982). "In vitro differentiation of human monocytes. Differences in monocyte phenotypes induced by cultivation on glass or on collagen". The Journal of Experimental Medicine. 156 (4): 1101–14. doi:10.1084/jem.156.4.1101. PMC 2186821 Freely accessible. PMID 6961188.
- Fausto N, Kaul KL (1999). "Presenting the Journal of Molecular Diagnostics". The Journal of Molecular Diagnostics. 1: 1. doi:10.1016/S1525-1578(10)60601-0. PMC 1906886 Freely accessible.
- Atanasovska B, Bozhinovski G, Chakalova L, Kocheva S, Karanfilski O, Plaseska-Karanfiska D (December 2012). "Molecular Diagnostics of β-Thalassemia". Balkan Journal of Medical Genetics. 15 (Suppl): 61–5. doi:10.2478/v10034-012-0021-z (inactive 2017-01-15). PMC 3776673 Freely accessible. PMID 24052746.
- Sharples A (2011-03-23). "Gene Patents in Europe Relatively Stable Despite Uncertainty in the U.S." Genetic Engineering and Biotechnology News. Retrieved 2013-06-13.
- Bravin J, Kendall B (13 June 2013). "Justices Strike Down Gene Patents". The Wall Street Journal. Retrieved 15 June 2013.
- Barnes R, Brady D (13 June 2013). "Supreme Court rules human genes may not be patented". The Washington Post. Retrieved 15 June 2013.
- Gibbs JN (1 August 2008). "Regulatory pathways for molecular diagnosis. Detailing the various options available and what each requires". 24 (14). Genetic Engineering & Biotechnology News. Retrieved 4 September 2013.
- Gibbs JN (1 April 2011). "Uncertainty persists with RUO products. FDA may be considering more restrictive approach with research use only assays". 31 (7). Genetic Engineering & Biotechnology News. Retrieved 4 September 2013.
- Tomiello K (21 February 2007). "Regulatory compliance drives LIMS". Design World. Retrieved 7 November 2012.
- Molecular Diagnostics: Current Technology and Applications (Horizon Bioscience). 7 July 2006. p. 97. ISBN 1-904933-19-X.
- van Ommen GJ, Breuning MH, Raap AK (June 1995). "FISH in genome research and molecular diagnostics". Current Opinion in Genetics & Development. 5 (3): 304–8. doi:10.1016/0959-437X(95)80043-3. PMID 7549423.
- Hammerling JA (2012). "A Review of Medical Errors in Laboratory Diagnostics and Where We Are Today". Laboratory Medicine. 43 (2): 41–44. doi:10.1309/LM6ER9WJR1IHQAUY.
- Khodakov D, Wang C, Zhang DY (October 2016). "Diagnostics based on nucleic acid sequence variant profiling: PCR, hybridization, and NGS approaches". Advanced Drug Delivery Reviews. 105 (Pt A): 3–19. doi:10.1016/j.addr.2016.04.005. PMID 27089811.
- doi.org/10.1371/journal.pone.0100566
- Walter G, Büssow K, Lueking A, Glökler J (June 2002). "High-throughput protein arrays: prospects for molecular diagnostics". Trends in Molecular Medicine. 8 (6): 250–3. doi:10.1016/S1471-4914(02)02352-3. PMID 12067604.
- Sherwood JL, Brown H, Rettino A, Schreieck A, Clark G, Claes B, Agrawal B, Chaston R, Kong BS, Choppa P, Nygren AO, Deras IL, Kohlmann A (2017-09-01). "Key differences between 13 KRAS mutation detection technologies and their relevance for clinical practice". ESMO Open. 2 (4): e000235. doi:10.1136/esmoopen-2017-000235. PMID 29018576.
- "Advances in Prenatal Molecular Diagnostics Conference (Introduction)". HealthTech. 2013. Retrieved 2013-09-28.
- "Molecular Diagnostics - National Cancer Institute". Cancer.gov. 2005-01-28. Retrieved 2013-09-26.
- Desta Z, Zhao X, Shin JG, Flockhart DA (2002). "Clinical significance of the cytochrome P450 2C19 genetic polymorphism". Clinical Pharmacokinetics. 41 (12): 913–58. doi:10.2165/00003088-200241120-00002. PMID 12222994.
- Eggermann T, Spengler S, Gogiel M, Begemann M, Elbracht M (June 2012). "Epigenetic and genetic diagnosis of Silver-Russell syndrome". Expert Review of Molecular Diagnostics. 12 (5): 459–71. doi:10.1586/erm.12.43. PMID 22702363.
- Tong CY, Mallinson H (May 2002). "Moving to nucleic acid-based detection of genital Chlamydia trachomatis". Expert Review of Molecular Diagnostics. 2 (3): 257–66. doi:10.1586/14737159.2.3.257. PMID 12050864.
- Deyde VM, Sampath R, Gubareva LV (January 2011). "RT-PCR/electrospray ionization mass spectrometry approach in detection and characterization of influenza viruses". Expert Review of Molecular Diagnostics. 11 (1): 41–52. doi:10.1586/erm.10.107. PMID 21171920.
- Pai M, Kalantri S, Dheda K (May 2006). "New tools and emerging technologies for the diagnosis of tuberculosis: part I. Latent tuberculosis". Expert Review of Molecular Diagnostics. 6 (3): 413–22. doi:10.1586/14737159.6.3.413. PMID 16706743.
- Burkardt HJ (January 2011). "Pandemic H1N1 2009 ('swine flu'): diagnostic and other challenges". Expert Review of Molecular Diagnostics. 11 (1): 35–40. doi:10.1586/erm.10.102. PMID 21171919.
- Han ET (March 2013). "Loop-mediated isothermal amplification test for the molecular diagnosis of malaria". Expert Review of Molecular Diagnostics. 13 (2): 205–18. doi:10.1586/erm.12.144. PMID 23477559.
- Tang YW, Procop GW, Persing DH (November 1997). "Molecular diagnostics of infectious diseases". Clinical Chemistry. American Association for Clinical Chemistry. 43 (11): 2021–38. PMID 9365385.
- van Lier MG, Wagner A, van Leerdam ME, Biermann K, Kuipers EJ, Steyerberg EW, Dubbink HJ, Dinjens WN (January 2010). "A review on the molecular diagnostics of Lynch syndrome: a central role for the pathology laboratory". Journal of Cellular and Molecular Medicine. 14 (1-2): 181–97. doi:10.1111/j.1582-4934.2009.00977.x. PMC 3837620 Freely accessible. PMID 19929944.
- Shrimpton AE (May 2002). "Molecular diagnosis of cystic fibrosis". Expert Review of Molecular Diagnostics. 2 (3): 240–56. doi:10.1586/14737159.2.3.240. PMID 12050863.
- Andorno R (October 2004). "The right not to know: an autonomy based approach". Journal of Medical Ethics. 30 (5): 435–9; discussion 439–40. doi:10.1136/jme.2002.001578. PMC 1733927 Freely accessible. PMID 15467071.
- Harmon, Amy (2008-02-24) Insurance Fears Lead Many to Shun DNA Tests. New York Times
- Minamoto T, Ougolkov AV, Mai M (November 2002). "Detection of oncogenes in the diagnosis of cancers with active oncogenic signaling". Expert Review of Molecular Diagnostics. 2 (6): 565–75. doi:10.1586/14737159.2.6.565. PMID 12465453.
- Brennan DJ, O'Connor DP, Rexhepaj E, Ponten F, Gallagher WM (September 2010). "Antibody-based proteomics: fast-tracking molecular diagnostics in oncology". Nature Reviews. Cancer. 10 (9): 605–17. doi:10.1038/nrc2902. PMID 20720569.
- Ferracin M, Veronese A, Negrini M (April 2010). "Micromarkers: miRNAs in cancer diagnosis and prognosis". Expert Review of Molecular Diagnostics. 10 (3): 297–308. doi:10.1586/erm.10.11. PMID 20370587.
- Misale S, Yaeger R, Hobor S, Scala E, Janakiraman M, Liska D, Valtorta E, Schiavo R, Buscarino M, Siravegna G, Bencardino K, Cercek A, Chen CT, Veronese S, Zanon C, Sartore-Bianchi A, Gambacorta M, Gallicchio M, Vakiani E, Boscaro V, Medico E, Weiser M, Siena S, Di Nicolantonio F, Solit D, Bardelli A (June 2012). "Emergence of KRAS mutations and acquired resistance to anti-EGFR therapy in colorectal cancer". Nature. 486 (7404): 532–6. doi:10.1038/nature11156. PMID 22722830.
- Emmadi R, Boonyaratanakornkit JB, Selvarangan R, Shyamala V, Zimmer BL, Williams L, Bryant B, Schutzbank T, Schoonmaker MM, Amos Wilson JA, Hall L, Pancholi P, Bernard K (November 2011). "Molecular methods and platforms for infectious diseases testing a review of FDA-approved and cleared assays". The Journal of Molecular Diagnostics. 13 (6): 583–604. doi:10.1016/j.jmoldx.2011.05.011. PMID 21871973.
- FDA-approved next-generation sequencing system could expand clinical genomic testing: experts predict MiSeqDx platform will make genetic testing more affordable for smaller labs. AM. J. Med. Genet. A 164, X-XI.
- Angulo B, Conde E, Suárez-Gauthier A, Plaza C, Martínez R, Redondo P, Izquierdo E, Rubio-Viqueira B, Paz-Ares L, Hidalgo M, López-Ríos F (2012-08-27). "A comparison of EGFR mutation testing methods in lung carcinoma: direct sequencing, real-time PCR and immunohistochemistry". PLOS One. 7 (8): e43842. doi:10.1371/journal.pone.0043842. PMID 22952784.
- Gonzalez de Castro D, Angulo B, Gomez B, Mair D, Martinez R, Suarez-Gauthier A, Shieh F, Velez M, Brophy VH, Lawrence HJ, Lopez-Rios F (July 2012). "A comparison of three methods for detecting KRAS mutations in formalin-fixed colorectal cancer specimens". British Journal of Cancer. 107 (2): 345–51. doi:10.1038/bjc.2012.259. PMC 3394984 Freely accessible. PMID 22713664.
- Marchant J, Mange A, Larrieux M, Costes V, Solassol J (July 2014). "Comparative evaluation of the new FDA approved THxID™-BRAF test with High Resolution Melting and Sanger sequencing". BMC Cancer. 14: 519. doi:10.1186/1471-2407-14-519. PMID 25037456.
- Salazar R, Roepman P, Capella G, Moreno V, Simon I, Dreezen C, Lopez-Doriga A, Santos C, Marijnen C, Westerga J, Bruin S, Kerr D, Kuppen P, van de Velde C, Morreau H, Van Velthuysen L, Glas AM, Van't Veer LJ, Tollenaar R (January 2011). "Gene expression signature to improve prognosis prediction of stage II and III colorectal cancer". Journal of Clinical Oncology. 29 (1): 17–24. doi:10.1200/JCO.2010.30.1077. PMID 21098318.
- Wittner BS, Sgroi DC, Ryan PD, Bruinsma TJ, Glas AM, Male A, Dahiya S, Habin K, Bernards R, Haber DA, Van't Veer LJ, Ramaswamy S (May 2008). "Analysis of the MammaPrint breast cancer assay in a predominantly postmenopausal cohort". Clinical Cancer Research. 14 (10): 2988–93. doi:10.1158/1078-0432.CCR-07-4723. PMC 3089800 Freely accessible. PMID 18483364.
- Lee CC, McMillin GA, Babic N, Melis R, Yeo KT (May 2011). "Evaluation of a CYP2C19 genotype panel on the GenMark eSensor® platform and the comparison to the Autogenomics Infiniti™ and Luminex CYP2C19 panels". Clinica Chimica Acta; International Journal of Clinical Chemistry. 412 (11-12): 1133–7. doi:10.1016/j.cca.2011.03.001. PMID 21385571.
- Pfundt R, Kwiatkowski K, Roter A, Shukla A, Thorland E, Hockett R, DuPont B, Fung ET, Chaubey A (February 2016). "Clinical performance of the CytoScan Dx Assay in diagnosing developmental delay/intellectual disability". Genetics in Medicine. 18 (2): 168–73. doi:10.1038/gim.2015.51. PMID 25880438.
- Khodakov, Dmitriy; Wang, Chunyan; Yu Zhang, David (2016-04-01). Diagnostics based on nucleic acid sequence variant profiling: PCR, hybridization, and NGS approaches. 105.
- Hovelson DH, McDaniel AS, Cani AK, Johnson B, Rhodes K, Williams PD, et al. (April 2015). "Development and validation of a scalable next-generation sequencing system for assessing relevant somatic variants in solid tumors". Neoplasia. 17 (4): 385–99. doi:10.1016/j.neo.2015.03.004. PMC 4415141 Freely accessible. PMID 25925381.
- Imperiale TF, Ransohoff DF, Itzkowitz SH (July 2014). "Multitarget stool DNA testing for colorectal-cancer screening". The New England Journal of Medicine. 371 (2): 187–8. doi:10.1056/NEJMc1405215. PMID 25006736.
- Rakovitch E, Nofech-Mozes S, Hanna W, Baehner FL, Saskin R, Butler SM, Tuck A, Sengupta S, Elavathil L, Jani PA, Bonin M, Chang MC, Robertson SJ, Slodkowska E, Fong C, Anderson JM, Jamshidian F, Miller DP, Cherbavaz DB, Shak S, Paszat L (July 2015). "A population-based validation study of the DCIS Score predicting recurrence risk in individuals treated by breast-conserving surgery alone". Breast Cancer Research and Treatment. 152 (2): 389–98. doi:10.1007/s10549-015-3464-6. PMC 4491104 Freely accessible. PMID 26119102.
- ترجمة diagnostics حسب المعجم الطبي الموحد؛ مكتبة لبنان ناشرون. نسخة محفوظة 11 أغسطس 2018 على موقع واي باك مشين.
- "DIRECTIVE 98/44/EC OF THE EUROPEAN PARLIAMENT AND OF THE COUNCIL of 6 July 1998 on the legal protection of biotechnological inventions"، Official Journal of the European Communities، European Parliament & Council، : 46-52، يوليو 1998، مؤرشف من الأصل في 16 سبتمبر 2020.
- بوابة تقانة
- بوابة تقانة حيوية
- بوابة طب
- بوابة علم الأحياء