ARN polimerasa
Las ARN-polimerasas (ARNP o ARNp) (RNAP en inglés) son un conjunto de enzimas capaces de emplear los ribonucleótidos para sintetizar ARN a partir de una secuencia de ADN que sirve como patrón o molde. La ARN polimerasa más importante es la implicada en la síntesis del ARN mensajero o transcripción del ADN.
| ARN polimerasas | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 2.7.7 | |||
| Estructura/Función proteica | ||||
| Funciones | Enzimas | |||
| Ortólogos | ||||
| Especies |
| |||
| Ubicación (UCSC) |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
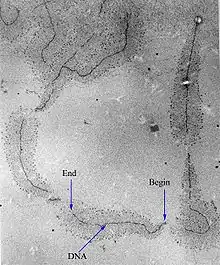
La ARN polimerasa es la enzima soluble conocida de mayor tamaño puesto que mide unos 100 Å de diámetro y es visible en micrografías electrónicas, donde se observa unida al promotor en la hebra del ADN.
Funciones
La reacción química que cataliza la ARN polimerasa (EC 2.7.7) consiste en la unión de ribonucleótidos trifosfato, adenosín trifosfato (ATP), uridín trifosfato (UTP), guanosin trifosfato (GTP) y citidín trifosfato (CTP), liberándose los grupos fosfato y convirtiéndose estos en nucleótidos.
Además de la polimerización de los ribonucleótidos trifosfato, la ARN polimerasa tiene otras funciones como:
- Reconocer y unirse a localizaciones específicas o promotores de la molécula de ADN.
- Desenrollar parcialmente la molécula del molde de ADN, gracias a su actividad helicasa intrínseca.
- Sintetizar un ARN cebador para la elongación posterior.
- Terminación de la cadena.
La ARN polimerasa cataliza consecutivamente la elongación de la cadena de ARN, al mismo tiempo que enrolla y desenrolla la doble cadena de ADN, y termina la transcripción después de copiar el gen.
Estructura
Esta complejidad de funciones se manifiesta en su estructura cuaternaria, ya que al igual que la ADN polimerasa, está formada por varias subunidades que conforman la holoenzima, que en unión con proteínas accesorias forman una máquina proteica o complejo de transcripción que llevan a cabo la síntesis del ARN.
Algunas subunidades aisladas de la ARN polimerasa son catalíticamente funcionales, mientras que otras sólo pueden detectarse cuando el complejo de transcripción se encuentra totalmente ensamblado.
Los complejos de transcripción de distintos organismos presentan una composición variable, pero esencialmente todos catalizan el mismo tipo de reacciones. Debido a esta coincidencia, en el estudio del proceso de transcripción se toma como modelo la reacciones catalizadas por el complejo de transcripción de la bacteria Escherichia coli, que aunque se diferencia en el ensamblamiento de las células eucarióticas, actúan de forma análoga.
La ARN polimerasa fue descubierta al mismo tiempo que el ARN mensajero en 1960 por los investigadores Samuel Weiss y Jerard Hurwits de laboratorios diferentes.
ARN polimerasa procariota
Los procariontes tienen un solo tipo de ARNP, responsable de la síntesis de todos los tipos de ARN. La misma enzima cataliza la síntesis de ARNm, ARNr y ARNt. Las ARN polimerasa procariota funciona de forma análoga, aunque alguna subunidad de la proteína difiera en su composición.
El ARNP procariota es una gran molécula que está especialmente relacionada con el ARNP II eucariota, tanto en Archaea[1] como en Bacteria,[2] debido a similitudes estructurales y mecánicas.
ARN polimerasa en bacterias
El ARNP bacteriano está formado por subunidades en número de 4 a 5. E. coli por ejemplo tiene el ARNP con 5 subunidades (β', β, α, ω y σ) y una masa de 400 UMA.[2]
Está formada por cinco subunidades de aproximadamente 410 kilodaltons α2ββ'ω, con dos unidades α idénticas, que se une al ADN de forma inespecífica para catalizar la síntesis de ARN. Para unirse a regiones promotoras específicas, la holoenzima requiere un factor σ con el que se reduce enormemente la afinidad con regiones de ADN inespecíficas, aumentando la especificidad por regiones promotoras para formar la holoenzima de cinco subunidades α2ββ'σω (~480 kDa). La estructura de la ARN polimerasa presenta una ranura de 55 Å de longitud y una anchura 25 Å. Esta ranura permite el paso de la doble hélice de ADN que mide 20 Å. La longitud de 55 Å puede aceptar la secuencia de 18 nucleótidos.
Todas las unidades que forman la enzima funcionan conjuntamente para llevar a cabo las reacciones de transcripción. La subunidad β' participa en la unión del ADN, la subunidad β contiene parte del centro activo y la subunidad σ está implicada principalmente en la iniciación de la transcripción, disociándose del resto de la enzima una vez iniciada la transcripción.
ARN polimerasa en arqueas
El ARNP arqueano está formado por subunidades en número de 8 a 14. Por ejemplo, el ARNP de Sulfolobus shibatae presenta 13 subunidades.[1]
ARN polimerasa eucariota
En las células eucariotas existen tres tipos de polimerasas nucleares de ARN (I, II y III), cada una especializada en la síntesis de un ARN determinado:
- ARN polimerasa I: síntesis, reparación y revisión. Sintetiza precursores de ARN ribosómico.
- ARN polimerasa II: reparación, Sintetiza precursores de ARN mensajero, microARNs y otros tipos de ácido ribonucleico.[3] Esta polimerasa es el tipo más estudiado, y se requieren factores de transcripción para que se una a los promotores del ADN. Su estructura tridimensional ha sido dilucidada por Roger Kornberg de la Universidad de Stanford, que recibió el Premio Nobel de Química en 2006 por sus trabajos. Se da la circunstancia que este investigador es hijo de otro Premio Nobel, Arthur Kornberg, que recibió el galardón en 1959 por el descubrimiento de una enzima análoga, la ADN polimerasa.[4] La enzima purificada tiene típicamente de 10 a 12 subunidades (12 en humanos y levaduras).
- ARN polimerasa III: sintetiza ARN de transferencia, ARN ribosómico de 5S y otros pequeños ARN (ARNpequeños) encontrados en el núcleo celular (ARNp nucleares) y en el citoplasma (ARNp citoplasmáticos).
- ARNP mitocondrial: El ARNPmt interviene en la transcripción y replicación del genoma mitocondrial.
ARN polimerasa en plantas
Adicionalmete a las ARNP eucariotas mencionadas, en plantas se encuentran la polimerasa IV y polimerasa V para reparación en condiciones únicas y síntesis de ARNip.
Además, dos tipos de ARNP se encuentran en el cloroplasto: uno de tipo fago y el otro de tipo procariota. Las subunidades proteicas de estas polimerasas están codificadas en los genomas nuclear y plastidial; por lo tanto, su activación y acción requieren una regulación altamente sofisticada que controla y coordina la expresión de los componentes.[5]
ARN polimerasa en virus
Los orthopoxvirus y algunos virus nucleocitoplasmáticos de ADN de gran tamaño presentan un ARNP vírico de múltiples subunidades. Son más similares a los ARNP eucariotas, con algunas subunidades minificadas o eliminadas.[6] Algunos virus de ADN usan una ARNP dependiente de ADN de una sola subunidad. Quizás el ARNP de subunidad única más ampliamente estudiado es la del bacteriófago T7.[cita requerida] Los virus de ARN usan exclusivamente una ARNP dependiente de ARN también llamada ARN replicasa, una polimerasa única de estos virus que cataliza la síntesis de la hebra de ARN complementaria a una plantilla de ARN determinada. Esto contrasta con las típicas ARN polimerasas dependientes de ADN, que todos los organismos celulares utilizan para catalizar la transcripción de ARN a partir de una plantilla de ADN.[7] Se ha sugerido que la ARNP dependiente de ARN o ARN replicasa fue la primera polimerasa en surgir y que las polimerasas del mundo de ARN + proteínas eran ARNP dependientes de ARN.[8][9]
Referencias
- Korkhin, Yakov; Unligil, Ulug M.; Littlefield, Otis; Nelson, Pamlea J.; Stuart, David I.; Sigler, Paul B.; Bell, Stephen D.; Abrescia, Nicola G. A. (5 de mayo de 2009). «Evolution of Complex RNA Polymerases: The Complete Archaeal RNA Polymerase Structure». PLOS Biology (en inglés) 7 (5): e1000102. ISSN 1545-7885. PMC 2675907. PMID 19419240. doi:10.1371/journal.pbio.1000102. Consultado el 24 de septiembre de 2022.
- Ebright, Richard H. (15 de diciembre de 2000). «RNA Polymerase: Structural Similarities Between Bacterial RNA Polymerase and Eukaryotic RNA Polymerase II». Journal of Molecular Biology (en inglés) 304 (5): 687-698. ISSN 0022-2836. doi:10.1006/jmbi.2000.4309. Consultado el 24 de septiembre de 2022.
- Kronberg, R.D. (1 de diciembre de 2009). «Eukaryotic transcriptional control». Trends in Cell Biology 9 (12): M46-49. PMID 10611681. doi:10.1016/S0962-8924(99)01679-7. Consultado el 24 de septiembre de 2022.
- Alentorn, Miquel Coll I. (10 de octubre de 2006). «El transcriptor de ADN». El País. ISSN 1134-6582. Archivado desde el original el 24 de septiembre de 2022. Consultado el 24 de septiembre de 2022.
- Pfannschmidt, Thomas; Blanvillain, Robert; Merendino, Livia; Courtois, Florence; Chevalier, Fabien; Liebers, Monique; Grübler, Björn; Hommel, Elisabeth et al. (9 de septiembre de 2015). «Plastid RNA polymerases: orchestration of enzymes with different evolutionary origins controls chloroplast biogenesis during the plant life cycle». Journal of Experimental Botany 66 (22): 6957–6973. doi:10.1093/jxb/erv415. Consultado el 24 de septiembre de 2022.
- Mirzakhanyan, Yeva; Gershon, Paul D. (Septiembre de 2017). «Multisubunit DNA-Dependent RNA Polymerases from Vaccinia Virus and Other Nucleocytoplasmic Large-DNA Viruses: Impressions from the Age of Structure». Microbiology and molecular biology reviews: MMBR 81 (3): e00010-17. ISSN 1098-5557. PMC 5584312. PMID 28701329. doi:10.1128/MMBR.00010-17. Consultado el 24 de septiembre de 2022.
- Koonin EV, Gorbalenya AE, Chumakov KM (July 1989). «Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases». FEBS Letters 252 (1–2): 42-6. PMID 2759231. doi:10.1016/0014-5793(89)80886-5.
- Koonin, Eugene V; Dolja, Valerian V (1 de octubre de 2013). «A virocentric perspective on the evolution of life». Current Opinion in Virology. Antivirals and resistance / Virus evolution (en inglés) 3 (5): 546-557. ISSN 1879-6257. doi:10.1016/j.coviro.2013.06.008. Consultado el 24 de septiembre de 2022.
- Robertson MP, Joyce GF (May 2012). «The origins of the RNA world». Cold Spring Harbor Perspectives in Biology 4 (5): a003608. PMC 3331698. PMID 20739415. doi:10.1101/cshperspect.a003608.