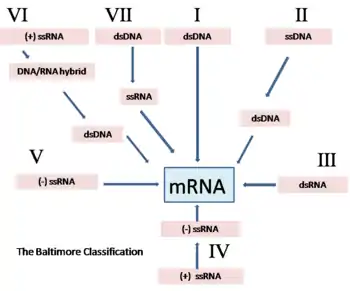
Classification Baltimore
La classification Baltimore est une classification des virus, proposée par le biologiste américain[1],[2] David Baltimore (lauréat du prix Nobel de physiologie ou médecine en 1975). Il s’agit d’un système de classification scientifique basé sur le génome des virus et leur type d'acide nucléique (à ADN ou à ARN, simple brin, double-brin) et son mode d'expression dans la synthèse de l’ARN messager viral, ainsi que le procédé de réplication de l'ADN.
Elle permet la classification des virus par leurs caractéristiques biologiques, qui peuvent être classées selon[3] :
- le type de cellule permettant la réplication du virus ;
- la durée du cycle de réplication virale ;
- la stratégie de réplication du virus (la classification de Baltimore).
Classification
La classification des virus en fonction de leur génome signifie que dans une catégorie donnée tous se comportent de la même façon, ce qui donne une certaine indication sur la façon de procéder à des recherches plus approfondies. La classification est :
Virus à ADN, groupes I et II :
- Groupe I - Virus à ADN à double brin (Adenovirus, Herpesvirus, Poxvirus)
- Groupe II - Virus à ADN à simple brin : ADN à polarité (+) (Parvovirus)
Virus à ARN, groupes III, IV et V :
- Groupe III - Virus à ARN à double brin (Reovirus)
- Groupe IV - Virus à ARN simple brin à polarité positive : ARN à polarité (+) (Picornavirus, Togavirus, Coronavirus)
- Groupe V - Virus à ARN simple brin à polarité négative : ARN à polarité (-) (Orthomyxovirus, Rhabdovirus)
Virus à transcription inverse, groupes VI et VII :
- Groupe VI - Rétrovirus à ARN simple brin : ARN à polarité (+) avec ADN intermédiaire dans le cycle de vie (Retrovirus)
- Groupe VII - Pararétrovirus à ADN double brin (Hepadnavirus)
Virus à ADN
Groupe I - Virus à ADN à double brin
Ce type de virus doit habituellement entrer dans le noyau de la cellule hôte avant de pouvoir se répliquer. En outre, ces virus ont besoin de l’ADN polymérase de la cellule hôte pour la réplication du génome viral et sont donc fortement tributaires du cycle cellulaire. La propagation de l'infection et la production de nouveaux virus nécessitent que la cellule soit dans une phase de réplication car c’est à ce moment que les polymérases de la cellule sont actives. Le virus peut induire une division cellulaire forcée, et lorsque cela survient de façon chronique, cette action peut conduire à une transformation de la cellule et, finalement, au cancer. Les exemples connus concernent les Herpesviridae, les Adenoviridae et les Papovaviridae.
Il n'existe qu'un seul exemple bien étudié dans lequel un virus de groupe I ne se réplique pas dans le noyau, c’est celui de la famille des poxvirus, un groupe de virus hautement pathogènes qui infectent les vertébrés et dont l’un des représentants est le virus de la variole.
Groupe II - Virus à ADN à simple brin
Les virus qui entrent dans cette catégorie comprennent certains agents infectieux qui n’ont pas été aussi bien étudiés, mais sont toujours très liés aux vertébrés. Citons deux exemples, les Circoviridae et les Parvoviridae. Ils se répliquent dans le noyau et forment un ADN double brin intermédiaire pendant la réplication. Un circovirus humain répandu mais asymptomatique, appelée Virus transfusionnel (en anglais Transfusion Transmitted Virus ou TTV) fait partie de ce groupe.
Virus à ARN
Groupe III - Virus à ARN à double brin
Comme avec la plupart des virus à ARN, les virus de ce groupe se répliquent dans le cytoplasme, sans avoir à utiliser les polymérases de la cellule hôte autant que les virus à ADN. Cette famille n'a pas été aussi bien étudiée que les autres et comprend deux grandes sous familles, les Reoviridae et les Birnaviridae. La réplication est monocistronique et individuelle, par segments de génomes, ce qui signifie que chacun des gènes code une seule protéine, contrairement à d'autres virus, dont la transcription se montre plus complexe.
Groupes IV & V: virus à ARN simple brin
Ces virus sont constitués de deux types, tous deux partageant cependant le fait que la réplication se déroule essentiellement dans le cytoplasme, et que la réplication n'est pas aussi dépendante du cycle cellulaire que pour d'autres virus à ADN. Cette catégorie de virus est l'une des mieux étudiées, avec celle des virus à ADN double brin.
Groupe IV - virus à ARN simple brin à polarité positive
(Virus (+)ssARN ou de type ARN messager)
La polarité positive des virus à ARN et en conséquence le fait que tous les gènes soient définis comme sens permet aux ribosomes de l'hôte de les décrypter directement pour synthétiser immédiatement des protéines. Ceux-ci peuvent être divisés en deux groupes, qui tous deux se répliquent dans le cytoplasme:
- Virus avec ARN messager polycistronique où l'ARN génomique forme l'ARNm et est traduit en poly-protéine qui est ensuite clivée pour former des protéines matures. Cela signifie que le gène peut utiliser plusieurs méthodes pour produire des protéines à partir du même brin d'ARN, le tout dans un souci de réduire la taille de son génome.
- Les virus à transcription complexe, pour lequel un ARNm subgénomique, l’intervention des ribosomes et un traitement protéolytique des polyprotéines peut être nécessaire. Tous ces mécanismes différents produisent des protéines à partir du même brin d'ARN.
Parmi les exemples de cette catégorie on trouve les familles des Astroviridae, des Caliciviridae, des Coronaviridae, des Flaviviridae, des Picornaviridae, des Arteriviridae et des Togaviridae.
Groupe V - virus à ARN simple brin à polarité négative
Les virus à ARN à polarité négative et même l'ensemble des gènes définis comme antisens ne peuvent pas être directement décrypté par les polymérases de l'hôte pour produire immédiatement des protéines. Au lieu de cela, ils doivent être transcrits par les polymérases virales dans une forme "lisible" à polarité positive. Ceux-ci peuvent également être divisés en deux groupes:
- Les virus contenant un génome non segmenté pour lesquels la première étape de la réplication est la transcription du brin de génome (-) par la polymérase virale ARN-dépendante en ARNm monocistronique codant les différentes protéines virales. Une copie (+) du génome est alors produite qui sert de matrice pour la production d’un brin (-) du génome. La réplication se déroule dans le cytoplasme.
- Les virus dont le génome est segmenté pour lesquels la réplication se produit dans le noyau cellulaire, et dont l'ARN polymérase virale ARN-dépendante monocistronique produit un ARNm à partir de chaque segment de génome. La différence la plus importante entre les deux est l'emplacement de la réplication.
Parmi les exemples dans cette catégorie figurent les familles des Arenaviridae, des Orthomyxoviridae, des Paramyxoviridae, des Bunyaviridae, des Filoviridae et des Rhabdoviridae (cette dernière famille inclut le virus de la rage).
Virus à ADN ou à ARN à transcription inverse
Ces virus utilisent la transcriptase inverse ou rétrotranscriptase (en anglais reverse transcriptase ou encore RT), une enzyme qui transcrit l'information génétique des virus de l'ARN en ADN, qui peut s'intégrer dans le génome de l'hôte.
Groupe VI – rétrovirus à ARN simple brin
Une famille bien étudiée dans cette classe de virus est celle des rétrovirus. Un élément déterminant est l'utilisation de la transcriptase inverse pour convertir l'ARN à polarité positive en ADN. Au lieu d'utiliser l'ARN pour les matrices de protéines, ils utilisent l'ADN pour créer les matrices qui sont greffées dans le génome de l'hôte en utilisant l’intégrase. La réplication peut alors commencer avec l'aide des polymérases de la cellule hôte. Un exemple bien étudié est celui du VIH.
Groupe VII - Pararétrovirus à ADN double brin
Ce petit groupe de virus, illustré par le virus de l’hépatite B (qui fait partie de la famille des Hepadnaviridae), comprend des virus à double brin, un génome sans chevauchement qui est ensuite complété afin de former un cercle fermé par liaison covalente (ADN ccc) qui sert de matrice pour la production d’ARNm viral et d’ARN subgénomique. L'ARN prégénomique sert de matrice pour la transcriptase inverse virale et pour la production d'ADN génomique.
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Baltimore classification » (voir la liste des auteurs).
- (en) Baltimore D, « Expression of animal virus genomes », Bacteriol Rev, vol. 35, no 3, , p. 235–41 (PMID 4329869, lire en ligne)
- Professeur Colimon, « Classification modifiée de Baltimore selon la stratégie de réplication des virus », Université de Rennes I - Département de virologie, (consulté le )
- Professeur Colimon, « Structure et classification des virus », Université de Rennes I - Département de virologie, (consulté le )
Liens externes
- "Virus Taxonomy Portal." (Website.) Viral Bioinformatics Resource Center & Viral Bioinformatics - Canada. Retrieved on 2007 -09-27.
- Family Groups - The Baltimore Method
- The Universal Virus Database of the International Committee on Taxonomy of Viruses
- The taxonomy portal of the Genbank database
- Portail de la virologie