Glutathion réductase
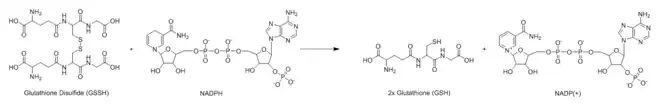
Une glutathion réductase est une oxydoréductase qui catalyse la réaction :
- 2 glutathion + NADP+ disulfure de glutathion + NADPH + H+.
| Glutathion réductase | ||
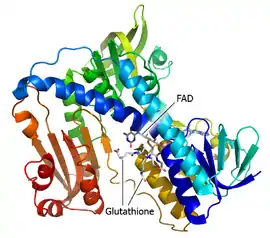 Structure d'une glutathion réductase cristallisée montrant le FAD et les deux molécules de glutathion (PDB 1GRE[1]) | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Glutathion disulfure réductase | |
| Symbole | GSR | |
| N° EC | 1.8.1.7 | |
| Homo sapiens | ||
| Locus | 8p12 | |
| Masse moléculaire | 56 257 Da[2] | |
| Nombre de résidus | 522 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
Cette enzyme permet de réduire le disulfure de glutathion en glutathion à l'aide de NADPH provenant notamment de la voie des pentoses phosphates chez les bactéries, les plantes et les animaux afin de régénérer le glutathion, molécule essentielle à la résistance contre le stress oxydant et à la préservation du pH intracellulaire[3],[4],[5].
La glutathion réductase est conservée à travers tous les règnes du vivant. On ne trouve qu'un seul gène codant la glutathion réductase chez les bactéries, les levures et les animaux. Le génome des plantes code en revanche deux isoenzymes. La drosophile et les trypanosomes sont dépourvus de glutathion réductase mais réalisent la réduction du glutathion à l'aide respectivement de thiorédoxine[6] et de trypanothion[7]. Chez l'homme, elle est codée par le gène GSR, sur le chromosome 8.
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | FAD |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Notes et références
- (en) P. Andrew Karplus et Georg E. Schulz, « Substrate binding and catalysis by glutathione reductase as derived from refined enzyme: Substrate crystal structures at 2Å resolution », Journal of Molecular Biology, vol. 210, no 1, , p. 163-180 (PMID 2585516, DOI 10.1016/0022-2836(89)90298-2, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Marcel Deponte, « Glutathione catalysis and the reaction mechanisms of glutathione-dependent enzymes », Biochimica et Biophysica Acta (BBA) - General Subjects, vol. 1830, no 5, , p. 3217-3266 (PMID 23036594, DOI 10.1016/j.bbagen.2012.09.018, lire en ligne)
- (en) Alton Meister, « Glutathione metabolism and its selective modification », Journal of Biological Chemistry, vol. 263, no 33, , p. 17205-17208 (PMID 3053703, lire en ligne)
- (en) B. Mannervik, « The enzymes of glutathione metabolism: an overview », Biochemical Society Transactions, vol. 15, no 4, , p. 717-718 (PMID 3315772, lire en ligne)
- (en) Stefan M. Kanzok, Anke Fechner, Holger Bauer, Julia K. Ulschmid, Hans-Michael Müller, José Botella-Munoz, Stephan Schneuwly, R. Heiner Schirmer et Katja Becker, « Substitution of the Thioredoxin System for Glutathione Reductase in Drosophila melanogaster », Science, vol. 291, no 5504, , p. 643-646 (PMID 11158675, DOI 10.1126/science.291.5504.643, lire en ligne)
- (en) R. Luise Krauth-Siegel et Marcelo A. Comini, « Redox control in trypanosomatids, parasitic protozoa with trypanothione-based thiol metabolism », Biochimica et Biophysica Acta (BBA) - General Subjects, vol. 1780, no 11, , p. 1236-1248 (PMID 18395526, DOI 10.1016/j.bbagen.2008.03.006, lire en ligne)
- Portail de la biochimie