Modèle cellulaire de Potts
Le modèle cellulaire de Potts (ou CPM, de l'anglais Cellular Potts model) est un modèle informatique de cellules et de tissus[1]. Il est aussi connu sous le nom de modèle Glazier-Graner-Hogeweg[2].
Le CPM est utilisé pour simuler des comportement cellulaires individuels et collectifs, la morphogenèse de tissus ou encore le développement d'un cancer. Dans ce modèle, une cellule est un objet déformable d'un certain volume, qui peut adhérer à d'autres cellules et au milieu dans lequel elle se trouve. Le modèle peut être étndu pour intégrer d'autres comportements cellulaires tels que la migration cellulaire, la croissance, la division et la signalisation.
Le premier CPM est une modification du modèle de Potts et a été proposé pour simuler un tri cellulaire par François Graner et James Glazier. Le CPM a ensuite été popularisé par Paulien Hogeweg pour l'étude de la morphogenèse[3]. Bien que le modèle ait été développé pour décrire des cellules biologiques, il peut être utilisé pour modéliser des sous-parties d'une cellule, ou même des zones ou des fluides.
Description du modèle
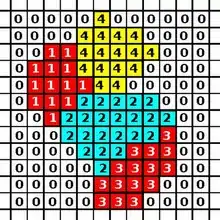
Le CPM est composé d'une grille rectangulaire où chaque pixel peut appartenir soit à une cellule, soit au milieu. Une cellule se compose donc d'un ensemble de pixels qui partagent le même identifiant (id).
La dynamique du modèle est régie par une fonction d'énergie hamiltonnienne qui décrit l'énergie d'une configuration particulière des cellules sur la grille. Dans le CPM classique, cette énergie dépend de l'adhésion entre les cellules et de la résistance des cellules au changement de volume. L'algorithme qui met à jour les identités cellulaires de chaque pixel minimise cette énergie en suivant un algorithme de type Monte-Carlo par chaîne de Markov (MCMC) :
- Choix d'un pixel aléatoire i par loi uniforme sur tous les pixels
- Choix aléatoire d'un pixel voisin de j , dont l'identité serait copié sur le pixel i
- Calcul de la différence d'énergie () entre l'ancienne et la potentielle nouvelle configuration sur la grille
- Acceptation ou rejet de la modification en fonction de :
- Si la nouvelle énergie est plus faible ( ), toujours accepter le changement
- Si la nouvelle énergie est plus élevée (), accepter le changement avec une probabilité où est la température de Boltzmann et détermine la probabilité de fluctuations énergétiquement défavorables.
L'hamiltonien
Le modèle original proposé par Graner et Glazier contient deux types de cellules, qui ont des paramètres différents pour l'adhésion cellulaire, avec une cellule du même type, de l'autre type ou avec le milieu. Le volume cellulaire est supposé proche d'une valeur cible et variant peu. L'hamiltonien est alors :
où et sont des pixels, la cellule au pixel , le type cellulaire de la cellule , le coefficient déterminant l'adhésion entre deux types cellulaires, le delta de Kronecker, le volume de la cellule , son volume cible et le multiplicateur de Lagrange déterminant la force de la contrainte de volume.
Les cellules avec un d'adhésion de leurs membranes plus faible vont avoir tendance à rester ensemble, ce qui permet de simuler différents motifs de tri cellulaires (mélange de types cellulaires, regroupement de cellules de même type, etc.).
Extensions
Au cours du temps, le CPM est passé d'un modèle spécifique du tri cellulaire à un modèle généraliste permettant l'intégration de nombreuses extensions[2] : chimiotaxie, élongation, ou motilité directionnelle. Ces extensions peuvent se faire en changeant soit l'hamiltonien soit l'énergie . D'autres sous-grilles peuvent être ajoutées pour inclure davantage d'information spatiale, comme la concentration en molécules.
Chimiotaxie
Dans le CPM, les cellules peuvent se déplacer dans la direction d'un gradient de concentration de chimiokine en augmentant la probabilité de copie d'id du pixel au pixel lorsque la concentration en chimiokine est plus grande au site . Pour cela, le calcul du changement d'énergie est modifié par un terme proportionnel à la différence de concentration entre et [3] :
où est la force du mouvement chimiotactique et et sont les concentrations en chimiokines aux sites et respectivement. Le gradient en chimiokine est généralement implémenté dans une grille secondaire de même dimension que la grille des cellules.
Modélisation multi-échelle et hybride
Des dynamiques de signalement internes aux cellules ainsi que des dynamiques à d'autres échelles temporelles (plus rapide ou plus lent que le mouvement des cellules) peuvent également être intégrés au CPM[4].
Code
Depuis 2020, une bibliothèque, intitulée Artistoo, est disponible en ligne[5] permettant de facilement télécharger une implémentation du CPM et d'y intégrer les extensions souhaitées. Le logiciel est sous licence libre et permet de simuler des cellules et des tissus directement dans un navigateur web[6].
Le logiciel open-source Bionetseolver peut également être utilisé pour intégrer une dynamique intracellulaire à l'algorithme CPM[7].
Notes et références
- (en) Marco Scianna et Luigi Preziosi, Cellular Potts Models: Multiscale Extensions and Biological Applications, CRC Press, (ISBN 978-1-4665-1479-9, lire en ligne)
- (en) Ariel Balter, Roeland M. H. Merks, Nikodem J. Popławski et Maciej Swat, « The Glazier-Graner-Hogeweg Model: Extensions, Future Directions, and Opportunities for Further Study », dans Single-Cell-Based Models in Biology and Medicine, Birkhäuser Basel, (ISBN 978-3-7643-8101-1, DOI 10.1007/978-3-7643-8123-3_7, lire en ligne), p. 151–167
- (en) Nicholas J. Savill et Paulien Hogeweg, « Modelling Morphogenesis: From Single Cells to Crawling Slugs », Journal of Theoretical Biology, vol. 184, no 3, , p. 229–235 (ISSN 0022-5193, DOI 10.1006/jtbi.1996.0237, lire en ligne, consulté le )
- András Szabó et Roeland M. Merks, « Cellular Potts Modeling of Tumor Growth, Tumor Invasion, and Tumor Evolution », Frontiers in Oncology, vol. 3, (ISSN 2234-943X, PMID 23596570, PMCID PMC3627127, DOI 10.3389/fonc.2013.00087, lire en ligne, consulté le )
- (en) « Home | Artistoo », sur artistoo.net (consulté le )
- (en) Inge MN Wortel et Johannes Textor, « Artistoo, a library to build, share, and explore simulations of cells and tissues in the web browser », eLife, vol. 10, , e61288 (ISSN 2050-084X, DOI 10.7554/eLife.61288, lire en ligne, consulté le )
- (en) Vivi Andasari, Ryan T. Roper, Maciej H. Swat et Mark A. J. Chaplain, « Integrating Intracellular Dynamics Using CompuCell3D and Bionetsolver: Applications to Multiscale Modelling of Cancer Cell Growth and Invasion », PLOS ONE, vol. 7, no 3, , e33726 (ISSN 1932-6203, PMID 22461894, PMCID PMC3312894, DOI 10.1371/journal.pone.0033726, lire en ligne, consulté le )
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Cellular Potts model » (voir la liste des auteurs).
- Portail de la biologie
- Portail de l’informatique