Réseau complexe
En théorie des graphes, un réseau complexe est un réseau possédant une architecture et une topologie complexe et irrégulière. Comme tous les réseaux, ils sont composés de nœuds (ou sommets ou points) représentant des objets, interconnectés par des liens (ou arêtes ou lignes). Ces réseaux sont des représentations abstraites des relations principalement présentes dans la vie réelle dans une grande diversité de systèmes biologiques et technologiques.
L’étude des réseaux complexes à fait l’objet d’une grande attention de la part de la communauté scientifique depuis le début des années 2000[1], et s’est montrée utile dans de nombreux domaines tels que la physique, la biologie, les télécommunications, l’informatique, la sociologie, l'épidémiologie entre autres.
Les réseaux complexe dans la vie réelle
Les réseaux complexes sont omniprésents autour de nous et ont de nombreuses applications dans la vie courante. Pour n’en nommer que quelques-uns, nous pouvons citer le World Wide Web, Internet, les réseaux trophiques (ou chaîne alimentaire) ou encore les réseaux métaboliques. Cette grande diversité de réseaux complexes rend leur classification selon leurs propriétés communes difficiles, mais nous pouvons retenir quatre groupes principaux[2] : les réseaux sociaux, les réseaux d’informations, les réseaux technologiques, et les réseaux biologiques.
Les réseaux sociaux
Un graphe de réseau social est un graphe permettant de représenter les interactions spécifiques entre différents groupes de personnes, représenté respectivement par les liens et les nœuds du graphe. Ces interactions peuvent être très variées, comme des liens d'amitié ou de parenté, des activités professionnelles ou personnelles communes, ou encore partager les mêmes opinions[3]. Les réseaux sociaux en ligne en sont un bon exemple, où Facebook peut être vu comme un graphe non orienté, puisque les “amitiés” sont bidirectionnelles, et Twitter quant à lui est un graphe orienté, puisque les "abonnements" sont à sens unique[4].
Les réseaux d’information
Les réseaux d’information sont une autre catégorie de réseaux. Un exemple typique de ce type de réseau est le World Wide Web[5], où les nœuds correspondent aux pages web contenant de l’information, et les liens sont les hyperliens permettant de naviguer d’une page à l'autre. Ce réseau de plusieurs milliards de nœuds est un graphe dirigé, mais qui ne contient malgré tout pas de boucles fermées, puisqu’il n’y a pas de contraintes dans le classement des sites internet.
Les réseaux de citations des articles académiques sont également un bon exemple de réseau d’information[6]. Ces réseaux sont acycliques, puisque des articles ne peuvent citer que des travaux déjà publiés.
Les réseaux technologiques
Nous pouvons également identifier les réseaux technologiques. Ce sont généralement des réseaux créés par l’Homme, comme les réseaux électriques[7], les réseaux de télécommunications, les réseaux aériens[7], les réseaux routiers[8] ou ferrés[9]. Mais, le réseau technologique le plus étudié est actuellement Internet[10], le réseau informatique mondial. Dans ce réseau, les ordinateurs et les routeurs sont les nœuds du réseau, et ces derniers sont connectés par des liens physiques comme la fibre optique modélisant les liens de ce réseau complexe.
Les réseaux biologiques
Les réseaux complexes permettent également de représenter la majorité des systèmes biologiques. Ils sont de ce fait très étudiés en biologie des réseaux et en bio-informatique.
Les organismes vivants étant très complexes, le nombre de réseaux biologiques présents dans une cellule vivante est énorme. Ces réseaux complexes possèdent des fonctions spécifiques souvent indispensables au bon fonctionnement cellulaire. De plus, ces réseaux sont fortement interconnectés et fonctionnent de façon coordonnée et synchronisée avec une grande précision, puisque le moindre dysfonctionnement peut entraîner une maladie.
Parmi ces nombreux réseaux, nous pouvons citer les réseaux d’interaction protéine-protéine[11], les réseaux de régulation des gènes[12], les réseaux de signalisation ou encore les réseaux métaboliques[13]. Les réseaux métaboliques (ou voies métaboliques) sont un exemple typique de réseau biologique. Ils représentent l’ensemble des réactions biochimiques permettant de convertir un composé en un autre dans les cellules. Dans un tel réseau, les nœuds seront les molécules biochimiques et les liens les réactions ayant permis de les obtenir.
En plus de permettre de mieux comprendre le fonctionnement cellulaire complexe, l'analyse de ces réseaux permet d’identifier plus précisément les causes de différentes maladies, et ainsi de développer de nouveaux traitements, ce qui a même mené à la création d’une nouvelle discipline : la médecine des réseaux[14].
Propriétés des réseaux complexes
Une des caractéristiques principales des réseaux complexes est qu'ils possèdent généralement un très grand nombre de nœuds reliés entre eux sans organisation évidente, si bien qu’ils peuvent faire penser à des réseaux aléatoires. Mais comme nous l’avons vu dans les exemples précédents, les réseaux complexes sont tout sauf aléatoire. Différentes mesures simplifiées ont donc été définies afin de caractériser les propriétés des réseaux complexes. Les trois principaux concepts sont la distribution des degrés, la longueur moyenne des chemins, et le coefficient de clustering.
Distribution des degrés
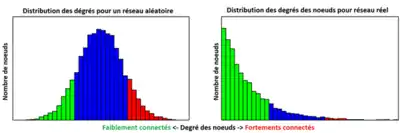
Le degré des nœuds d’un réseau est une des caractéristiques les plus importantes pour définir les réseaux complexes[16]. Le degré d'un nœud correspond au nombre de connexions qu’il possède avec les autres nœuds du réseau. Ainsi, plus le degré d'un nœud est élevé, plus le nœud d'un réseau est connecté et est important. Pour reprendre un exemple précédent, dans un réseau social, le nœud d'une personne avec 100 amitiés possède un degré de 100.
Nous pouvons ensuite étudier la distribution de ces degrés pour caractériser la structure d’un réseau. Cette fonction de distribution est définie comme la probabilité qu'un nœud sélectionné aléatoirement ait exactement k arêtes. Un réseau régulier à un degré simple, car chaque nœud est connecté à un nombre égal d'autres nœuds. On n'observe donc qu'un seul pic dans un graphique représentant la distribution des degrés. Pour un graphe aléatoire, les degré de distribution obéissent à un distribution de Poisson (Fig. 1, à gauche). Et, pour un réseau complexe, la distribution des degrés suit généralement une loi exponentielle et permet de caractériser de façon précise le type de réseau complexe d’un système (Fig. 1, à droite).
La longueur moyenne des chemins
La longueur moyenne des (plus courts) chemins est une autre mesure robuste de la topologie d’un réseau. En choisissant deux nœuds quelconques dans un graphe, la distance entre ces nœuds correspond au nombre d'arêtes du plus petit chemin reliant ces deux nœuds. La longueur moyenne des chemins d'un réseau est donc définie comme la distance moyenne entre deux nœuds, moyennée sur tous les chemins disponibles entre ces nœuds[16].
L’analyse de cette propriété est très utile. Dans un réseau comme Internet, avoir une courte longueur moyenne des chemins permettra un transfert rapide des informations et de ce fait réduire les coûts de calculs. Et, dans un réseau électrique, minimiser cette longueur moyenne des plus courts chemins permet de réduire les déperditions d’énergie.
La plupart des réseaux réels ont une longueur de chemin moyenne très courte conduisant au concept d'un réseau « petit monde » où beaucoup des nœuds sont interconnectés par des chemins très courts.
Le coefficient de clustering
Le coefficient de clustering, (aussi appelé coefficient d'agglomération, de connexion, de regroupement, d'agrégation ou de transitivité) est la probabilité que deux nœuds soient connectés sachant qu'ils ont un voisin commun[16].
Dans un réseau complexe, ce coefficient sera généralement élevé, puisqu’il est probable que des voisins d’un nœud soient également liés l’un de l’autre.
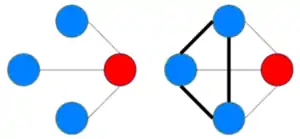
Dans l’image ci-contre (Fig. 2), nous pouvons voir sur le réseau de gauche que les 3 nœuds bleu sont reliés au nœud rouge, mais qu’ils ne forment pas de liaison entre eux. Dans le réseau à droite, tous les nœuds voisins du nœud rouge sont reliés entre eux. Ce dernier réseau à donc un coefficient de regroupement plus important.
Comme observé sur le graphique précédent, chaque voisin du nœud rouge () est connecté à chaque autre voisin du nœud rouge (), alors au maximum il peut exister arêtes.
Ainsi, le coefficient de clustering du nœud (rouge) est défini comme :
où est le rapport entre le nombre de ponts qui existent parmi ces nœuds et le nombre total de ponts . Pour le réseau le plus connecté à droite on obtient donc le calcul :
Méthodes d'analyse des réseaux complexes
Il existe nombre de façons pour analyser un réseau, mais toutes les méthodes ne sont pas forcément applicables aux réseaux complexes.
Une première manière de procéder est d’utiliser des mesures statistiques pour identifier de façon quantitative les caractéristiques du réseau, comme l’étude du degré des nœuds[16], ou la distribution des degrés.
Une autre approche est de représenter graphiquement le réseau afin de permettre à l'Homme de l'analyser et de l’interpréter. Cette visualisation permet aux experts du domaine d’extraire des motifs intéressants des données, et de découvrir des informations qui n’auraient pas été identifiées en utilisant simplement des mesures statistiques.
Mais, les réseaux complexes étant bien souvent difficiles à visualiser dans leur ensemble et à étudier de manière statistique, du fait de leur taille pouvant être très importante, il devient pertinent de décomposer le réseau en plusieurs composantes. En d’autres termes, il est possible de diviser les nœuds du réseau en plusieurs sous-groupes basés sur différents critères, comme la nature des données du réseau ou le type de relations que l’on souhaite identifier. Cette décomposition permettra par la suite d’appliquer les approches précédemment décrites[16].
Décomposition de réseau
Il existe différentes méthodes pour décomposer un réseau en sous-graphes de sommets.
Il est dans un premier temps possible de réaliser une décomposition basée sur les k-cores. Les k-cores sont des sous-graphes où chaque sommet possède au moins k voisins, avec k une valeur limite choisie par l’analyste et dépendante du graphe étudié. Cela a notamment permis d’étudier les clusters des réseaux sociaux ou décrire l'évolution des réseaux aléatoires et est fréquemment utilisé en bio-informatique pour visualiser les réseaux complexes[16], [18].
Une autre méthode est la décomposition par k-truss. Les k-truss sont des sous graphes où chaque sommet est relié à au moins k-2 autres sommets. En d’autre termes, chaque sommet du k-truss fait partie de k-2 triangles formés à partir de nœuds de ce k-truss[19].
Il existe également des méthodes hybrides combinant la décomposition par k-core et la décomposition par k-truss comme la décomposition par noyaux. Technique qui emploie un ordre hiérarchique plus important que les deux méthodes séparées et pouvant révéler des groupes non détectés avec les méthodes précédentes[20].
Enfin, la décomposition topologique, dans laquelle le réseau est décomposé selon la distribution des degrés et la densité des noyaux. Cette approche permet de révéler la présence de groupes de nœuds partageant des densités similaires.
Analyse
Ces groupes de fortes connectivité permettent d’identifier les nœuds centraux du réseau, qui possèdent potentiellement une fonction commune. Un bon exemple de ce procédé est l’étude des réseaux biologiques, en identifiant par exemple un groupe de protéines impliquées dans un symptôme important d’une maladie[21].
Une fois que le nombre de nœud du réseau à diminué, il est tout à fait possible d’appliquer les différentes métriques classique d’analyse de réseau, comme la connectivité du réseau (distribution des degrés), la distance du réseau et de ses composants (Exemple : Réseau « petit monde »), la centralité intermédiaire des nœuds, ou encore des métriques de similarité des nœuds. Il sera également possible d’utiliser des algorithmes de partitionnement des données, comme les méthodes de K-means ou de clustering hiérarchique[22].
Réseau "petit monde"
Le principe mathématique du réseau « petit monde », établi par Watts et Strongatz[23] sur l’hypothèse du phénomène de petit monde[24] décrit par Stanley Milgram dit qu’il est possible de créer une relation entre deux individus avec un maximum de 6 degrés de séparation. De nombreux systèmes sont formés sur le principe de réseau « petit monde », mais ceux qui les représentent le mieux sont Internet, les réseaux sociaux et les réseaux métaboliques[25].
Propriétés d'un réseau petit monde
La classification d’un graphe repose généralement sur plusieurs paramètres définit précisément, comme le coefficient de clustering et la longueur de chemin . Classiquement, un réseau est considéré comme étant de type « petit monde » si le plus court chemin entre deux nœuds choisis aléatoirement croît de façon logarithmique en fonction du nombre de nœuds du réseau. Mais, il existe également plusieurs métriques spécifiques ayant pour but de déterminer si un réseau est un réseau « petit monde » ou non. L'une d'entre elles est le ratio entre le réseau supposé « petit monde » et un réseau aléatoire. Cette métrique ()[26] indiquera donc dans quelle mesure un réseau est de type « petit monde ». Si > 1 alors celui peut être considéré comme un réseau « petit monde ».
Ce ratio σ est grandement influencé par la taille du réseau, ce qui rend son appréciation difficile. Pour pouvoir mesurer la capacité d’un réseau à être de type « petit monde », il est également possible d’utiliser la différence du ratio entre un réseau aléatoire et un réseau voulu.
et correspondent respectivement aux valeurs de longueur de chemin et du coefficient de clustering d’un réseau aléatoire, tandis que correspond à un réseau équivalent. [26] va varier entre -1 et 1 quelle que soit la taille du réseau. Plus la valeur de ω est proche de zéro plus le réseau est considéré comme « petit monde ». Des valeurs positives indiquent un caractère aléatoire dans le réseau. Des valeurs négatives indiquent un caractère plus régulier dans la composition du réseau. Le modèle de réseau « petit monde », Watts et Strogatz définissent les paramètres d’un réseau avec une petite valeur de qui est représentatif des graphes dit "aléatoires" et une valeur de clustering très élevé représentatif des graphes plus réguliers.
Applications
Les domaines d’application d’un réseau « petit monde » sont variés. On les retrouve en sociologie, avec les réseaux sociaux. Dans ces réseaux, il est question de la capacité d’une information à se transmettre à travers l’ensemble d’un réseau par les individus et comment l’affinité des membres peuvent influer sur cette même capacité de transmission.
Dans une application plus concrète comme la gestion des fermetures des aéroports. Grâce à l’application du réseau « petit monde », il est possible d’expliquer pourquoi la fermeture de certains aéroports ou lieu de transits obligerait les voyageurs à faire escale plus souvent que la normale.
On les retrouve également en informatique[27], ou l’étude des réseaux « petit monde », ont permis de mieux comprendre comment stocker et gérer les informations dans un base de données pour rendre accessible le plus rapidement possible dans le futur une information pour l’utilisateur.
Enfin, ces réseaux sont présents en sciences biologiques et plus particulièrement dans les neurosciences[28]. Ici il est question de pouvoir adapter et comparer un réseau « petit monde » à un réseau cérébral. La plupart des études se sont intéressées à la comparaison sur l’anatomie et la structure des réseaux, l’aspect fonctionnel mais aussi les interactions entre nœuds et arrêtes. Dans certains cas, il a été question d’utiliser ce genre de réseau pour mimer les AVC et les crises d'épilepsie[29]. Dans le cadre de la microbiologie par exemple, on étudiera la capacité de transmission des plasmides ou de l’information génétiques liés à la résistance bactérienne ou virale.
Réseau spatial
Les systèmes complexes sont très souvent des réseaux où les nœuds et les arêtes sont compris dans l'espace. Les réseaux de transport, Internet, les réseaux de téléphonie mobile, les réseaux électriques, les réseaux sociaux et les réseaux de neurones sont tous des exemples où la prise en compte de l’espace est pertinente puisque leur topologie seule ne contient pas toutes les informations disponible pour leur analyse. La caractérisation et la compréhension de la structure et de l'évolution des réseaux spatiaux sont donc cruciaux dans de nombreux domaines, comme l'urbanisme ou l'épidémiologie.
La prise en compte de l'espace sur les réseaux est possible avec l’ajout d’un coût associé à la longueur des nœuds. Ces contraintes spatiales affectent la structure topologique et les propriétés de ces réseaux.
Les réseaux spatiaux ont été dans un premier temps développés en géographie quantitative et font l'objet de nombreuses recherches comme l’étude des lieux, des activités, des flux d'individus et de biens, qui sont des réseaux évoluant dans le temps et dans l'espace. Ces premières recherches ont permis de développer des méthodes et outils pour caractériser les réseaux spatiaux. Cela a entre autres permis de résoudre de nombreux problèmes importants tels que la localisation des nœuds d'un réseau, l'évolution des réseaux de transport et leur interaction avec la population et la densité d'activité. Mais de nombreux points importants restent encore flous et bénéficieront certainement des connaissances actuelles sur les réseaux et les systèmes complexes.
Applications
Pour la plupart des applications pratiques, l'espace de ces réseaux est un espace bidimensionnel, et la valeur associée aux nœuds est la distance euclidienne classique[30]. Cela implique en général que la probabilité de trouver un lien entre deux nœuds diminue avec la distance. Cependant, cela n'implique pas forcément qu'un réseau spatial soit planaire. Par exemple, le réseau aérien reliant les aéroports du monde entier n'est pas un réseau planaire, soit un réseau qui peut être représenté dans un plan de façon ou ses liens ne se croisent pas. Avec cette définition d'un réseau spatial, les liens ne sont pas nécessairement dans l'espace : les réseaux sociaux par exemple relient les individus à travers des relations d'amitié. Dans ce cas, l'espace intervient dans le fait que la probabilité de connexion entre deux individus diminue généralement avec la distance qui les sépare. Cependant, de nombreux réseaux d'infrastructure seront inévitablement planaires. En plus d’être des réseaux spatiaux, les routes, le rail et les autres réseaux de transport sont principalement des réseaux planaires.
Références
- Réka Albert et Albert-László Barabási, « Statistical mechanics of complex networks », Reviews of Modern Physics, vol. 74, no 1, , p. 47–97 (DOI 10.1103/RevModPhys.74.47, lire en ligne, consulté le )
- (en) M. E. J. Newman, « The Structure and Function of Complex Networks », SIAM Review, vol. 45, no 2, , p. 167–256 (ISSN 0036-1445 et 1095-7200, DOI 10.1137/S003614450342480, lire en ligne, consulté le )
- John Scott et Peter Carrington, « The SAGE Handbook of Social Network Analysis », Manuel, (DOI 10.4135/9781446294413, lire en ligne, consulté le )
- Johan Ugander, Brian Karrer, Lars Backstrom et Cameron Marlow, « The Anatomy of the Facebook Social Graph », arXiv:1111.4503 [physics], (lire en ligne, consulté le )
- B. A. Huberman, The laws of the Web : patterns in the ecology of information, MIT Press, (ISBN 978-0-262-27583-5, 0-262-27583-X et 0-585-44840-X, OCLC 52289489, lire en ligne)
- « Introduction to Informetrics: Quantitative Methods in Library, Documentation and Information Science. Leo Egghe, Ronald Rousseau », The Library Quarterly, vol. 61, no 2, , p. 220–221 (ISSN 0024-2519, DOI 10.1086/602337, lire en ligne, consulté le )
- (en) L. A. N. Amaral, A. Scala, M. Barthelemy et H. E. Stanley, « Classes of small-world networks », Proceedings of the National Academy of Sciences, vol. 97, no 21, , p. 11149–11152 (ISSN 0027-8424 et 1091-6490, PMID 11005838, PMCID PMC17168, DOI 10.1073/pnas.200327197, lire en ligne, consulté le )
- (en) Vamsi Kalapala, Vishal Sanwalani, Aaron Clauset et Cristopher Moore, « Scale invariance in road networks », Physical Review E, vol. 73, no 2, , p. 026130 (ISSN 1539-3755 et 1550-2376, DOI 10.1103/PhysRevE.73.026130, lire en ligne, consulté le )
- (en) Vito Latora et Massimo Marchiori, « Is the Boston subway a small-world network? », Physica A: Statistical Mechanics and its Applications, horizons in Complex Systems, vol. 314, no 1, , p. 109–113 (ISSN 0378-4371, DOI 10.1016/S0378-4371(02)01089-0, lire en ligne, consulté le )
- Qian Chen, Hyunseok Chang, R. Govindan et S. Jamin, « The origin of power laws in Internet topologies revisited », Proceedings.Twenty-First Annual Joint Conference of the IEEE Computer and Communications Societies, IEEE, vol. 2, , p. 608–617 (ISBN 978-0-7803-7476-8, DOI 10.1109/INFCOM.2002.1019306, lire en ligne, consulté le )
- (en) A. R. Mashaghi, A. Ramezanpour et V. Karimipour, « Investigation of a protein complex network », The European Physical Journal B - Condensed Matter and Complex Systems, vol. 41, no 1, , p. 113–121 (ISSN 1434-6036, DOI 10.1140/epjb/e2004-00301-0, lire en ligne, consulté le )
- (en) Shai S. Shen-Orr, Ron Milo, Shmoolik Mangan et Uri Alon, « Network motifs in the transcriptional regulation network of Escherichia coli », Nature Genetics, vol. 31, no 1, , p. 64–68 (ISSN 1546-1718, DOI 10.1038/ng881, lire en ligne, consulté le )
- (en) Jörg Stelling, Steffen Klamt, Katja Bettenbrock et Stefan Schuster, « Metabolic network structure determines key aspects of functionality and regulation », Nature, vol. 420, no 6912, , p. 190–193 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/nature01166, lire en ligne, consulté le )
- (en) Albert-László Barabási, Natali Gulbahce et Joseph Loscalzo, « Network medicine: a network-based approach to human disease », Nature Reviews Genetics, vol. 12, no 1, , p. 56–68 (ISSN 1471-0064, PMID 21164525, PMCID PMC3140052, DOI 10.1038/nrg2918, lire en ligne, consulté le )
- « Network science: node degree distribution », sur www.network-science.org (consulté le )
- (en) « Complex Networks: A Review », International Journal of Computer Applications,
- (en) Atanu Chatterjee, « Studies on the Structure and Dynamics of Urban Bus Networks in Indian Cities », Thesis,
- Luca Dall’Asta, Ignacio Alvarez-Hamelin, Alain Barrat et Alexei Vázquez, « k-core decomposition: a tool for the analysis of largescale Internet graphs », Physical Review E, vol. 71, no 3, (ISSN 1539-3755 et 1550-2376, DOI 10.1103/physreve.71.036135, lire en ligne, consulté le )
- (en) « Finding Trusses »
- Finding the Hierarchy of Dense Subgraphsusing Nucleus Decompositions, Ahmet Erdem Sarıyüce, https://arxiv.org/pdf/1411.3312.pdf
- (en) Faraz Zaidi, « Analysis, Structure and Organization of Complex Networks », Networking and Internet Architecture, , Chap 2 p21-22 (lire en ligne)
- (en) Miloš Savić, Mirjana Ivanović et Lakhmi C. Jain, « Fundamentals of Complex Network Analysis », dans Complex Networks in Software, Knowledge, and Social Systems, Springer International Publishing, coll. « Intelligent Systems Reference Library », (ISBN 978-3-319-91196-0, DOI 10.1007/978-3-319-91196-0_2, lire en ligne), p. 17–56
- (en) Duncan J. Watts et Steven H. Strogatz, « Collective dynamics of ‘small-world’ networks », Nature, vol. 393, no 6684, , p. 440–442 (ISSN 0028-0836 et 1476-4687, DOI 10.1038/30918, lire en ligne, consulté le )
- Stanley Milgram, « The small-world problem », sur PsycEXTRA Dataset, (consulté le )
- (en) Andreas Wagner et David A. Fell, « The small world inside large metabolic networks », Proceedings of the Royal Society of London. Series B: Biological Sciences, vol. 268, no 1478, , p. 1803–1810 (ISSN 0962-8452 et 1471-2954, PMID 11522199, PMCID PMC1088812, DOI 10.1098/rspb.2001.1711, lire en ligne, consulté le )
- (en) Qawi K. Telesford, Karen E. Joyce, Satoru Hayasaka et Jonathan H. Burdette, « The Ubiquity of Small-World Networks », Brain Connectivity, vol. 1, no 5, , p. 367–375 (ISSN 2158-0014 et 2158-0022, PMID 22432451, PMCID PMC3604768, DOI 10.1089/brain.2011.0038, lire en ligne, consulté le )
- « Small Worlds Business Measure of Data », dans Information-Driven Business, John Wiley & Sons, Inc., (ISBN 978-1-119-20033-8, lire en ligne), p. 55–65
- (en) Shan Yu, Debin Huang, Wolf Singer et Danko Nikolić, « A Small World of Neuronal Synchrony », Cerebral Cortex, vol. 18, no 12, , p. 2891–2901 (ISSN 1460-2199 et 1047-3211, PMID 18400792, PMCID PMC2583154, DOI 10.1093/cercor/bhn047, lire en ligne, consulté le )
- (en) O Sporns, D Chialvo, M Kaiser et C Hilgetag, « Organization, development and function of complex brain networks », Trends in Cognitive Sciences, vol. 8, no 9, , p. 418–425 (DOI 10.1016/j.tics.2004.07.008, lire en ligne, consulté le )
- (en) Marc Barthélemy, « Spatial networks », Physics Reports, vol. 499, no 1, , p. 1–101 (ISSN 0370-1573, DOI 10.1016/j.physrep.2010.11.002, lire en ligne, consulté le )
- Portail de l'informatique théorique