Virome
Le terme virome fait référence à l'ensemble des génomes des virus[1],[2] souvent étudié et décrit par séquençage métagénomique d'acides nucléiques viraux[3] associés à un écosystème, un organisme ou un holobionte particulier. Le mot est fréquemment utilisé pour décrire les métagénomes viraux obtenus par séquençage shotgun. Les virus, y compris les bactériophages, se trouvent dans tous les environnements, et les études du virome ont fourni des informations sur le cycle des nutriments[4],[5], développement de l'immunité,[6] et une source majeure de gènes par conversion lysogène[7].
Histoire
Les premières études complètes sur les viromes ont été effectuées par séquençage communautaire[8] souvent appelée métagénomique. Dans les années2000, le laboratoire Rohwer a séquencé des viromes à partir de l'eau de mer[9], de sédiments marins[10], de selles humaines adultes[11] et infantiles[12], de terre[13] et de sang[14]. Ce groupe a également réalisé le premier virome ARN avec des collaborateurs de l'Institut génomique de Singapour[15]. De ces premiers travaux, il a été conclu que la majeure partie de la diversité génomique est contenue dans le virome global et que la majeure partie de cette diversité reste non caractérisée[16]. Ce point de vue a été soutenu par un projet de séquençage génomique individuel, en particulier le phage mycobacterium[17].
Hôtes de virus
Les virus sont les entités biologiques les plus abondantes sur Terre, mais les défis de détection, d'isolement et de classification des virus inconnus ont empêché des études exhaustives du virome mondial[18]. Plus de 5 Tb de données de séquence métagénomique ont été utilisés à partir de 3 042 échantillons géographiquement diversifiés pour évaluer la distribution mondiale, la diversité phylogénétique et la spécificité de l'hôte des virus.
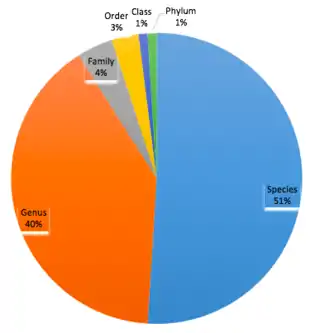
En août 2016, plus de 125 000 génomes viraux à ADN partiel, y compris le plus gros phage encore identifié, ont multiplié par seize le nombre de gènes viraux connus[18]. Une suite de méthodes de calcul a été utilisée pour identifier les connexions de virus hôtes putatifs. Les informations sur l'hôte viral de l'isolat ont été projetées sur un groupe, ce qui a donné lieu à des attributions d'hôte pour 2,4% des groupes viraux.
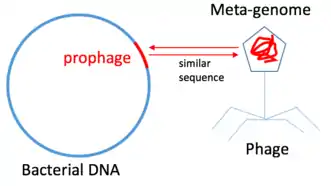
Ensuite, le système immunitaire procaryote CRISPR–Cas qui contient une "bibliothèque" de fragments de génome provenant de phages (proto-espaceurs) ayant précédemment infecté l'hôte[18]. Des espaceurs de génomes microbiens isolés avec des correspondances avec des contigs viraux métagénomiques (mVC) ont été identifiés pour 4,4% des groupes viraux et 1,7% des singletons. L'hypothèse a été explorée que les gènes d'ARN de transfert viral (ARNt) proviennent de leur hôte.
Les ARNt viraux identifiés dans 7,6% des mVC ont été appariés pour isoler les génomes d'une seule espèce ou d'un seul genre[18]. La spécificité de l'attribution virale de l'hôte basée sur l'ARNt a été confirmée par des correspondances d'espacement CRISPR – Cas montrant une concordance de 94% au niveau du genre. Ces approches ont identifié 9 992 associations hôtes-virus putatives permettant l'attribution d'hôte à 7,7% des mVC. La majorité de ces connexions étaient auparavant inconnues et incluent des hôtes de seize phylums procaryotes pour lesquels aucun virus n'a été identifié auparavant.
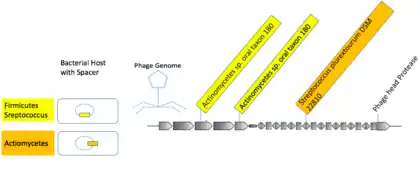
De nombreux virus se spécialisent dans l'infection d'hôtes associés[18]. Il peut exister des généralistes viraux qui infectent les hôtes de tous les ordres taxonomiques. La plupart des correspondances d'espaceurs CRISPR provenaient de séquences virales vers des hôtes d'une espèce ou d'un genre. Certains mVC étaient liés à plusieurs hôtes de taxons supérieurs. Un groupe viral composé de macs (en) d'échantillons oraux humains contenait trois photo-espaceurs distincts avec des correspondances presque exactes aux espaceurs dans Actinobacteria et Firmicutes.
En janvier 2017, le système IMG/VR[19] — la plus grande base de données virale publique interactive — contenait 265 000 séquences virales métagénomiques et isolait les virus. Ce nombre est passé à plus de 760 000 en novembre 2018 (IMG/VR v.2.0)[20]. Les systèmes IMG / VR servent de point de départ pour l'analyse de séquence de fragments viraux dérivés d'échantillons métagénomiques.
Notes et références
- Anderson, Gerin et Anderson, « Global Screening for Human Viral Pathogens. », Emerging Infectious Diseases, vol. 9, no 7, , p. 768-773 (PMID 12890315, PMCID 3023425, DOI 10.3201/eid0907.030004)
- Zárate, Taboada, Yocupicio-Monroy et Arias, « Human Virome. », Archives of Medical Research, vol. 48, no 8, , p. 701-716 (DOI 10.1016/j.arcmed.2018.01.005)
- McDaniel, Breitbart, Mobberley et Long, « Metagenomic analysis of lysogeny in Tampa Bay: implications for prophage gene expression. », PLOS ONE, vol. 3, no 9, , e3263 (PMID 18810270, PMCID 2533394, DOI 10.1371/journal.pone.0003263)
- Wilhelm et Suttle, « Viruses and Nutrient Cycles in the Sea », BioScience, vol. 49, no 10, , p. 781–788 (ISSN 1525-3244, DOI 10.2307/1313569, JSTOR 1313569)
- Wegley, Edwards, Rodriguez-Brito et Liu, « Metagenomic analysis of the microbial community associated with the coral Porites astreoides. », Environmental Microbiology, vol. 9, no 11, , p. 2707–19 (PMID 17922755, DOI 10.1111/j.1462-2920.2007.01383.x)
- Barr, Auro, Furlan et Whiteson, « Bacteriophage adhering to mucus provide a non-host-derived immunity. », Proceedings of the National Academy of Sciences of the United States of America, vol. 110, no 26, , p. 10771–6 (PMID 23690590, PMCID 3696810, DOI 10.1073/pnas.1305923110)
- Sharon, Battchikova, Aro et Giglione, « Comparative metagenomics of microbial traits within oceanic viral communities. », The ISME Journal, vol. 5, no 7, , p. 1178–90 (PMID 21307954, PMCID 3146289, DOI 10.1038/ismej.2011.2)
- Breitbart, Salamon, Andresen et Mahaffy, « Genomic analysis of uncultured marine viral communities. », Proceedings of the National Academy of Sciences of the United States of America, vol. 99, no 22, , p. 14250–5 (PMID 12384570, PMCID 137870, DOI 10.1073/pnas.202488399)
- Angly, Felts, Breitbart et Salamon, « The marine viromes of four oceanic regions. », PLOS Biology, vol. 4, no 11, , e368 (PMID 17090214, PMCID 1634881, DOI 10.1371/journal.pbio.0040368)
- Breitbart, Felts, Kelley et Mahaffy, « Diversity and population structure of a near-shore marine-sediment viral community. », Proceedings of the Royal Society B: Biological Sciences, vol. 271, no 1539, , p. 565–74 (PMID 15156913, PMCID 1691639, DOI 10.1098/rspb.2003.2628)
- Breitbart, Hewson, Felts et Mahaffy, « Metagenomic analyses of an uncultured viral community from human feces. », Journal of Bacteriology, vol. 185, no 20, , p. 6220–3 (PMID 14526037, PMCID 225035, DOI 10.1128/jb.185.20.6220-6223.2003)
- Breitbart, Haynes, Kelley et Angly, « Viral diversity and dynamics in an infant gut. », Research in Microbiology, vol. 159, no 5, , p. 367–73 (PMID 18541415, DOI 10.1016/j.resmic.2008.04.006)
- Fierer, Breitbart, Nulton et Salamon, « Metagenomic and small-subunit rRNA analyses reveal the genetic diversity of bacteria, archaea, fungi, and viruses in soil. », Applied and Environmental Microbiology, vol. 73, no 21, , p. 7059–66 (PMID 17827313, PMCID 2074941, DOI 10.1128/aem.00358-07)
- Breitbart et Rohwer, « Method for discovering novel DNA viruses in blood using viral particle selection and shotgun sequencing. », BioTechniques, vol. 39, no 5, , p. 729–36 (PMID 16312220, DOI 10.2144/000112019)
- Zhang, Breitbart, Lee et Run, « RNA viral community in human feces: prevalence of plant pathogenic viruses. », PLOS Biology, vol. 4, no 1, , e3 (PMID 16336043, PMCID 1310650, DOI 10.1371/journal.pbio.0040003)
- Edwards et Rohwer, « Viral metagenomics. », Nature Reviews. Microbiology, vol. 3, no 6, , p. 504–10 (PMID 15886693, DOI 10.1038/nrmicro1163)
- Rohwer, « Global phage diversity. », Cell, vol. 113, no 2, , p. 141 (PMID 12705861, DOI 10.1016/s0092-8674(03)00276-9)
- « Uncovering Earth's virome », Nature, vol. 536, no 7617, , p. 425–30 (PMID 27533034, DOI 10.1038/nature19094, lire en ligne)
- « IMG/VR: a database of cultured and uncultured DNA Viruses and retroviruses », Nucleic Acids Res., vol. 45, no D1, , D457–D465 (PMID 27799466, PMCID 5210529, DOI 10.1093/nar/gkw1030)
- « IMG/VR v.2.0: an integrated data management and analysis system for cultivated and environmental viral genomes », Nucleic Acids Res., vol. 47, no Database issue, , D678–D686 (PMID 30407573, PMCID 6323928, DOI 10.1093/nar/gky1127)