Caudoviricetes
Caudoviricetes es una extensa clase de virus que infectan procariotas (bacterias y arqueas). Son llamados "caudovirus" o "bacteriófagos de cola".
| Caudoviricetes | ||
|---|---|---|
 | ||
| Taxonomía | ||
| Dominio: | Duplodnaviria | |
| Clase: | Caudoviricetes | |
| Clasificación de Baltimore | ||
| Grupo: | I (Virus ADN bicatenario) | |
Comprenden la mayor parte de los bacteriófagos. En el marco del esquema de la Clasificación de Baltimore se integran en el Grupo I, ya que tienen un genoma ADN bicatenario. El tamaño del genoma está comprendido entre 18 y 500 kbp. Las partículas virales tienen una forma distintiva con una cabeza icosaedral que contiene el genoma y conectada a una cola. La clase abarca una amplia gama de virus, muchos de los cuales tienen los mismos o similares genes, mientras que la secuencia de nucleótidos puede variar considerablemente, incluso entre el mismo género. Debido a su característica estructural, se cree que comparten un origen común y que fueron los ancestros de los herpesvirus según análisis filogenéticos y estructurales.
Los caudovirus infectan bacterias y arqueas de casi todos los filos según el gran incremento por metagenómica y la abundancia de elementos virales endógenos. Aunque la mayoría de los filos bacterianos y arqueanos permanecen sin cultivarse, se ha descrito como un elemento viral endógeno en los genomas de muchos de estos filos candidatos o los huéspedes se determinaron por técnicas de metagenómica. También infectaron al último antepasado común universal según investigaciones recientes.[1] Los caudovirus desde su origen aparentemente fueron los virus que le ganaron la competencia a los restantes tipos de virus procariotas y por ello son muy abundantes, se hallan con más frecuencia en los cultivos, muestras metagenómicas, endogenizados y su forma de virion es la popularmente concebida para todos los bacteriófagos a pesar de que los bacteriófagos pueden tener diferentes formas de virion al igual que los virus eucariotas.
Infección
Al encuentro con una bacteria o arquea huésped, la cola del virus se une a los receptores de la superficie celular procariota y el virus introduce su ADN en la célula mediante el uso de un mecanismo de inyección. La cola hace un agujero a través de la pared celular y de la membrana plasmática de la bacteria o arquea y el genoma pasa por la cola a la célula. Una vez dentro, los genes se expresan mediante la transcripción hecha por la maquinaria huésped, haciendo uso de los ribosomas. Típicamente, el genoma se replica haciendo uso de concatémeros.
Ensamblado y maduración
Las proteínas de la cápside se reúnen para formar una protocabeza, en el que se introduce el genoma. Una vez que esto ha ocurrido, la maduración de la protocabeza se realiza por la división de las unidades de la cápside para formar una cabeza icosaedral con simetría 5. Después de la maduración, la cola se añade por una de dos maneras: o bien la cola se construye por separado y se une con la cabeza, o bien se construye directamente sobre la cabeza del fago. La cola consiste en una hélice de proteínas con simetría 6. Después de la maduración de las partículas virales, la célula es lisada usando lisinas, holinas o una combinación de ambas.
Características por morfotipos
Se pueden hallar tres morfotipos dentro los caudovirus: podovirus, sifovirus y miovirus antiguamente clasificados como familias:
Podovirus
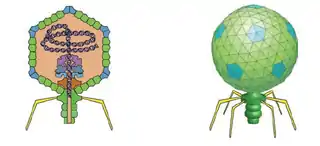
Los viriones de los podovirus tienen cápsides con geometrías icosaédricas y cabeza-cola. No poseen envoltura vírica. El diámetro es de alrededor de 60 nm, y consta de 72 capsómeros. Las proteínas de la cabeza tiene una masa molecular de ~ 38 kiloDaltons y está presente en 460 copias por virión. Hay 9 proteínas estructurales. La cola no es contráctil y tiene 6 fibras subterminales cortas. Es grueso, tiene forma de varilla y está construido con discos apilados. La longitud máxima es de ~ 17 nm.
El genoma es lineal y de ADN bicatenario, de alrededor de 40-42 kb de longitud, y codifica ~ 55 genes. El contenido de guanina + citosina es ~ 50%. Tienen secuencias terminalmente redundantes y no están permutadas. En peso, el genoma constituye ~ 50% de los virus. El genoma codifica 9 proteínas estructurales, una ADN polimerasa de tipo transferasa B adenilada y una ARN polimerasa. Tres proteínas internas constituyen el complejo polimerasa. Se reconocen dos clases de genes (temprano y tardío). Esta clasificación se basa en el momento de la transcripción que está regulado temporalmente. Los genes con funciones relacionadas se agrupan. La replicación del genoma es bidireccional.
La replicación viral se produce en el citoplasma. La entrada en la célula huésped se logra mediante adsorción en la célula huésped. La replicación sigue el modelo de desplazamiento de la cadena de ADN. La transcripción con plantilla de ADN es el método de transcripción. El virus sale de la célula huésped por lisis y proteínas holina/endolisina/spanina. Las bacterias y arqueas sirven como huéspedes naturales. Las rutas de transmisión son por difusión pasiva.
Se considera que HTVC010P, bacteriófago de Pelagibacter ubique, es el organismo más abundante de la Tierra.[2]
Sifovirus
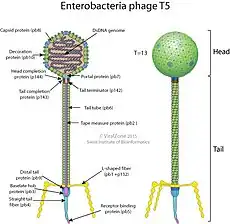
Los viriones tienen cápsides con geometrías icosaédricas y cabeza-cola (morfotipo B1) o una cápside alargada (morfotipo B2), y simetría T = 7. No poseen envoltura vírica. El diámetro es de unos 60 nm. Los virus de este tipo se caracterizan por sus colas filamentosas, con bandas cruzadas y no contráctiles, generalmente con fibras terminales y subterminales cortas. Los genomas lineales y de ADN bicatenario con alrededor de 50 kb de longitud y contienen alrededor de 70 genes. El contenido de guanina/citosina suele rondar el 52%.
La replicación viral se produce en el citoplasma. La entrada en la célula huésped se logra mediante adsorción en la célula huésped. La replicación sigue el modelo de transposición replicativa. La transcripción con plantilla de ADN es el método de transcripción. La traducción se realiza mediante un desplazamiento del marco ribosómico -1 y un desplazamiento del marco ribosómico +1. El virus sale de la célula huésped por lisis y proteínas holina/endolisina/spanina. Las bacterias y las arqueas sirven como hospedadores naturales. Las rutas de transmisión son por difusión pasiva.
Miovirus

Los viriones de los miovirus tienen geometrías icosaedricas con cabeza-cola (con cuello). No poseen envoltura vírica. Los genomas son lineales de ADN bicatenario, de alrededor de 33-244 kb de longitud. El genoma codifica de 40 a 415 proteínas. Tiene secuencias terminalmente redundantes. El contenido de GC es ~ 35%. El genoma codifica 200-300 proteínas que se transcriben en operones. La 5-hidroximetilcitosina puede estar presente en el genoma (en lugar de timidina).
La cola tubular tiene simetría helicoidal y tiene un diámetro de 16-20 nm. Consiste en un tubo central, una vaina contráctil, un collar, una placa base, seis pines de cola y seis fibras largas. Es similar a los siphovirus, pero difiere en el hecho de que la cola de un miovirus es permanente. Las contracciones de la cola requieren ATP. Al contraerse la vaina, las subunidades de la vaina se deslizan una sobre la otra y la cola se acorta a 10-15 nm de longitud.
Al adherirse a una célula huésped, el virus usa su vaina contráctil como una jeringa, perforando la pared celular con su tubo central e inyectando el material genético en el huésped. El ADN inyectado se hace cargo de los mecanismos de transcripción y traducción de la célula huésped y comienza a fabricar nuevos viriones. La replicación sigue el modelo de transposición replicativa. La transcripción con plantilla de ADN es el método de transcripción. La traducción se realiza mediante un desplazamiento del marco ribosómico -1. El virus sale de la célula huésped por lisis y proteínas holina/endolisina/spanina. Las bacterias y arqueas sirven como hospedadores naturales. Las rutas de transmisión son por difusión pasiva.
Aunque los miovirus son en general líticos y carecen de los genes necesarios para volverse lisogénicos, se conocen varias especies de zonas templadas. Debido a que la mayoría de los miovirus son fagos líticos, en lugar de templados, algunos investigadores han investigado su uso como terapia para enfermedades bacterianas en humanos y otros animales.
Taxonomía
Los caudovirus son un grupo que está creciendo fuertemente en número de especies y géneros descubiertos por metagenómica, pero también en la identificación en cultivos de nuevos procariotas, por lo que se ha propuesto muchas familias adicionales. Se ha sugerido abolir las familias tradicionales Podoviridae, Siphoviridae y Myoviridae elevando sus subfamilias a rango de familia y abolir el orden Caudovirales para crear nuevos órdenes con nuevas familias descubiertas o elevadas de rango debido al gran incremento para acomodar taxones, la gran divergencia evolutiva y porque dichas familias son polifiléticas.[3][4]
Los caudovirus se clasifican bajo los siguientes órdenes y familias según el ICTV:[5]
- Orden Crassvirales
- Familia Crevaviridae
- Familia Intestiviridae
- Familia Steigviridae
- Familia Suoliviridae
- Orden Kirjokansivirales
- Familia Flexireviridae
- Familia Haloferuviridae
- Familia Pyrstoviridae
- Familia Shortaselviridae
- Orden Juravirales
- Familia Yangangviridae
- Familia Yanlukaviridae
- Orden Methanobavirales
- Familia Anaeroviridae
- Familia Leisingerviridae
- Orden Nakonvirales
- Familia Ahpuchviridae
- Familia Ekchuahviridae
- Orden Thumleimavirales
- Familia Hafunaviridae
- Familia Halomagnusviridae
- Familia Queuoviridae
- Familia Soleiviridae
- Familias de colocación incierta:
- Familia Aoguangviridae
- Familia Aliceevansviridae
- Familia Arenbergviridae
- Familia Ackermannviridae
- Familia Aggregaviridae
- Familia Assiduviridae
- Familia Autographiviridae
- Familia Casjensviridae
- Familia Chaseviridae
- Familia Demerecviridae
- Familia Drexlerviridae
- Familia Duneiviridae
- Familia Fervensviridae
- Familia Fredfastierviridae
- Familia Forsetiviridae
- Familia Herelleviridae
- Familia Helgolandviridae
- Familia Guelinviridae
- Familia Grimontviridae
- Familia Rountreeviridae
- Familia Salasmaviridae
- Familia Schitoviridae
- Familia Zobellviridae
- Familia Kleczkowskaviridae
- Familia Kyanoviridae
- Familia Madisaviridaeviridae
- Familia Molycoviridae
- Familia Pachyviridae
- Familia Peduoviridae
- Familia Pootjesviridae
- Familia Pungoviridae
- Familia Naomiviridae
- Familia Vilmaviridae
- Familia Zierdtviridae
- Familia Pervagoviridae
- Familia Orlajensenviridae
- Familia Mesyanzhinoviridae
- Familia Saparoviridae
- Familia Speroviridae
- Familia Straboviridae
- Familia Stanwilliamsviridae
- Familia Suolaviridae
- Familia Verdandiviridae
- Familia Vertoviridae
- Familia Winoviridae
- Agentes de transferencia de genes (elementos genéticos procariotas derivados):[6]
- Familia Bartogtaviriformidae
- Familia Brachygtaviriformidae
- Familia Rhodogtaviriformidae
Relación con los organismos celulares
Los caudovirus junto con los virus gigantes son los únicos virus que codifican genes necesarios y tienen algunos elementos para los análisis filogenéticos del árbol de la vida, lo que les permite poderlos incluir en el árbol de la vida. En los análisis filogenéticos basados en genes de polimerasas y traducción los caudovirus aparecen como un linaje basal del árbol filogenético, mientras que los virus gigantes suelen aparecer dentro los eucariotas, por la transferencia horizontal de genes.[7] Por otra parte, en los análisis del proteoma incluido todos los virus emergen como un grupo parafilético, siendo los virus de ARN parafiléticos de los virus de ADN y los virus gigantes los más cercanos a los organismos celulares. También los análisis filogenéticos y estructurales han revelado que los herpesvirus que infectan animales junto con los mirusvirus que infectan eucariotas unicelulares son descendientes de los caudovirus estando estrechamente emparentados con la subfamilia Tevenvirinae de la familia Straboviridae, además ambos comparten una proteína única denominada HK97-MCP, por ello se clasifican en el dominio Duplodnaviria.[8] Esto es consistente con que los caudovirus y otros virus se originaron antes que el último antepasado común universal (LUCA).
Véase también
Referencias
- Mart Krupovic, Valerian V. Dolja, Eugene V. Koonin (2020). The LUCA and its complex virome. Nature.
- Zhao, Y.; Temperton, B.; Thrash, J. C.; Schwalbach, M. S.; Vergin, K. L.; Landry, Z. C.; Ellisman, M.; Deerinck, T.; Sullivan, M. B.; Giovannoni, S. J. (2013). "Abundant SAR11 viruses in the ocean". Nature. 494 (7437): 357–360. Bibcode:2013Natur.494..357Z. doi:10.1038/nature11921. PMID 23407494.
- Bacterial and archaeal viruses proposals. ICTV.
- A Roadmap for Genome-Based Phage Taxonomy - MDPI.
- «Virus Taxonomy: 2020 Release». talk.ictvonline.org. International Committee on Taxonomy of Viruses. Consultado el 25 de abril de 2020.
- Jens Kuhn, Eugene Koonin (2022). Viriforms—A New Category of Classifiable Virus-Derived Genetic Elements. NCBI.
- A Mystery clade in the Tree of Life.
- Andrade-Martínez JS, Moreno-Gallego JL, Reyes A (August 2019). «Defining a Core Genome for the Herpesvirales and Exploring their Evolutionary Relationship with the Caudovirales». Sci Rep 9 (1): 11342. Bibcode:2019NatSR...911342A. PMC 6683198. PMID 31383901. doi:10.1038/s41598-019-47742-z. Consultado el 19 de mayo de 2020.
- Maniloffi, J., and Ackermann, H.W., 1998, “Taxonomy of bacterial viruses: establishment of tailed virus genera and the order Caudovirales”, Achievements in Virology, 143, 10
- Xu, J., et al, 2004, “Conserved Translational Frameshift in dsDNA Bacteriophage Tail Assembly Genes”, Molecular Cell, 16, 1
- Casjens, S.R., 2005, “Comparative genomics and evolution of the tailed-bacteriophages”, Current Opinion in Microbiology, 8, 4, 451-458