Piridoxal fosfato
El piridoxal fosfato (PLP, piridoxal-5'-fosfato, P5P) es un grupo prostético caracterizado por presentar un aldehído como sustituyente del carbono 4 del núcleo de piridina, ligado a ciertas enzimas. Es la forma activa de la vitamina B6, la cual comprende tres compuestos naturales, piridoxal, piridoxamina y piridoxina. Esta coenzima actúa como un sumidero de electrones para estabilizar los intermediarios carbaniónicos tanto en las reacciones de eliminación como de sustitución que involucran a compuestos aminados.
| Piridoxal fosfato | ||
|---|---|---|
 Diagrama de esqueleto del piridoxal fosfato. | ||
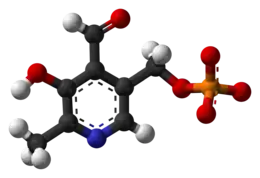 | ||
| Nombre IUPAC | ||
| Ácido [(4-formil-5-hidroxi-6-metilpiridin-3-il)metoxi] fosfónico | ||
| General | ||
| Otros nombres | Piridoxal 5-fosfato, PAL-P, PLP, Vitamina B6 fosfato | |
| Fórmula molecular |
C 8H 10NO 6P | |
| Identificadores | ||
| Número CAS | 54-47-7[1] | |
| ChEMBL | 82202 | |
| PubChem | 1051 | |
| KEGG | C00018 | |
|
CC1=NC=C(C(=C1O)
C=O)COP(=O)(O)O | ||
| Propiedades físicas | ||
| Densidad | 1638 kg/m³; 1,638 g/cm³ | |
| Masa molar | 247,142 g/mol | |
| Punto de fusión | 139/−142 °C (412/131 K) | |
| Propiedades químicas | ||
| Acidez | 1.56[2] pKa | |
| Peligrosidad | ||
| Punto de inflamabilidad | 569 K (296 °C) | |
| Valores en el SI y en condiciones estándar (25 ℃ y 1 atm), salvo que se indique lo contrario. | ||
Papel como coenzima
El PLP actúa como coenzima en todas las reacciones de transaminación, y en ciertas reacciones de descarboxilación, desaminación, y racemización de aminoácidos. El grupo aldehído del PLP forma una estructura tipo base de Schiff (una aldimina interna) con el grupo ε-amino de una lisina ubicado en el sitio activo de la aminotransferasa. El grupo α-amino del aminoácido sustrato desplaza al grupo ε-amino de la lisina ubicada en el sitio activo en un proceso conocido como "transaldiminación". La aldimina externa resultante puede perder un protón, dióxido de carbono, o la cadena lateral del aminoácido para formar un intermediario quinoide, el cual puede además actuar como nucléofilo en numerosas vías de reacción.
En las transaminaciones, luego de la desprotonación, el intermediario quinoide acepta un protón en una posición diferente para convertirse en una cetimina. La cetimina resultante se hidroliza de forma que el grupo amino permanece en el complejo.[3] Adicionalmente, el PLP es utilizado por las aminotransferasas (conocidas también como transaminasas) que actúan sobre algunos azúcares inusuales, tales como la perosamina y la desosamina.[4] En estas reacciones, el PLP se combina con glutamato, el cual transfiere su grupo alfa amino al PLP para formar piridoxamina fosfato (PMP). Luego este PMP transfiere su nitrógeno al azúcar, formando un amino azúcar.
El PLP también se encuentra involucrado en varias reacciones de eliminación tales como las que llevan a cabo la serina deshidratasa y la GDP-4-ceto-6-desoximanosa-3-deshidratasa (Co1D).[4]
También participa en la reacción de condensación que tiene lugar en la síntesis del heme.
El PLP, también desempeña una función en la conversión de la levodopa en dopamina, facilitando la conversión del neurotransmisor excitatorio glutamato en el neurotransmisor inhibitorio GABA, y permite que la SAM se pueda descarboxilar para convertirse en propilamina, la cual es una precursora de las poliaminas.
Ejemplos no clásicos de PLP
El PLP también se puede encontrar en la glucógeno fosforilasa en el hígado, donde se utiliza para degradar el glucógeno en el proceso de glucogenólisis cuando el glucagón o la adrenalina señalizan la necesidad de llevar a cabo la reacción. Sin embargo, esta enzima no explota la reactividad del grupo aldehído, en cambio hace uso del grupo fosfato del PLP para llevar a cabo esta reacción.
A pesar de que la gran mayoría de las enzimas dependientes de PLP forman una aldimina interna con el PLP por medio de un residuo lisina en el sitio activo, algunas enzimas dependientes del PLP no poseen este residuo lisina, pero en cambio, poseen una histidina en el sitio activo. En estos casos, la histidina no puede formar la aldimina interna, y, por lo tanto, el cofactor no deviene covalentemente unido a la enzima. Un ejemplo de este tipo de enzimas es la GDP-4-ceto-6-desoximanosa-3-deshidratasa (ColD).[5]
Mecanismo catalítico
Las enzimas dependientes de piridoxal-5'-fosfato (enzimas PLP) catalizan un millar de reacciones bioquímicas. Aunque en un primer vistazo las reacciones catalizadas por PLP parecen ser inmensamente diversas, poseen un principio unificador bastante simple: En el estado inactivo, el cofactor (PLP) se encuentra unido covalentemente al grupo amino de una lisina presente en el sitio activo, formando una aldimina interna. Cuando el sustrato amino interactúa con el sitio activo, se forma una nueva base de Schiff, que por lo común se suele nombrar como aldimina externa. Luego de este paso, la vía mecanística para cada una de las reacciones catalizadas por PLP divergen. Se han aplicado métodos de densidad funcional para investigar la reacción de transiminación, el resultado ha demostrado que la reacción involucra tres pasos secuenciales:
- Formación de un intermediario tetraédrico con la lisina del sitio activo y el sustrato amino unidos al cofactor PLP.
- Transferencia indirecta de un protón entre el sustrato amino y el residuo lisina.
- Formación de una aldimina externa luego de la disociación del residuo lisina.
La reacción total es exotérmica (-12 Kcal/mol), siendo el segundo paso el limitante de la velocidad, con una energía de activación de 12,6 Kcal/mol.[6]

Especificidad
La especificidad se encuentra conferida por el hecho de que, de los cuatro enlaces del carbono alfa en el estado aldimina aminoácido, el enlace perpendicular al anillo piridina es el que resulta roto (hipótesis estereoelectrónica de Dunathan).[7] En consecuencia, la especificidad se encuentra dictada por la manera en como la enzima liga a sus sustratos. La facilidad con la que el nitrógeno del anillo piridina se protona juega un papel adicional en la especificidad.[7]
Enzimas PLP
El PLP se encuentra fijado en el sitio activo no sólo gracias a la lisina, sino también gracias a la interacción del grupo fosfato con el bolsillo fijador de fosfato, y en forma menos intensa, gracias al apilamiento de bases que se produce entre el anillo piridina y un residuo aromático colgante, generalmente una tirosina (el cual también toma parte en el mecanismo de catálisis ácido-base). A pesar de los escasos requerimientos necesarios para que se produzca el bolsillo fijador de PLP, las enzimas PLP pertenecen sólo a cinco familias diferentes. Estas familias no guardan una buena correlación con un tipo particular de reacción. Las cinco familias se clasifican con números romanos de acuerdo a su tipo de plegamiento.[7]
- Plegamiento Tipo I — familia de la aspartato aminotransferasa
- Plegamiento Tipo II — familia de la triptófano sintasa
- Plegamiento Tipo III — familia de la alanina racemasa (barril-TIM)
- Plegamiento Tipo IV — familia de la D-aminoácido aminotransferasa
- Plegamiento Tipo V — familia de la glucógeno fosforilasa
Biosíntesis
A partir de vitámeros
Los animales son auxótrofos para este cofactor enzimático, y necesitan de él o de un intermediario como suplemento, de ahí su clasificación como vitamina (B
6); a diferencia de lo que ocurre, por ejemplo, con el cofactor de molibdeno o la coenzima CoQ10. El PLP se sintetiza a partir de piridoxal por medio de una enzima llamada piridoxal quinasa, requiriendo de una molécula de ATP. El metabolismo del PLP ocurre en el hígado.
Prototrofía
Se conocen actualmente dos vías metabólicas naturales para la síntesis de PLP: una requiere de deoxixilulosa 5-fosfato (DXP), mientras que la otra no; por lo tanto se conocen como DXP dependiente y DXP independiente. Estas vías metabólicas han sido estudiadas extensivamente en Escherichia coli y Bacillus subtilis, respectivamente. A pesar de su disparidad en cuanto a los compuestos iniciales y al diferente número de pasos que requieren, las dos vías metabólicas poseen muchas similitudes.[8]
Biosíntesis dependiente de DXP
La síntesis dependiente de DXP requiere de varios pasos y de la convergencia de dos ramas metabólicas subsidiarias, una que produce 3-hidroxi-1-aminoacetona fosfato a partir de eritrosa 4-fosfato, mientras que la otra produce deoxixilulosa 5-fosfato (DXP) a partir de gliceraldehido 3-fosfato (GAP) y piruvato. La condensación del 3-hidroxi-1-aminoacetona fosfato y deoxixilulosa 5-fosfato produce piridoxina 5-fosfato. La condensación es catalizada por la PNP sintasa, enzima codificada por el gen pdxJ.[9] El paso final es catalizado por una PNP oxidasa (pdxH), la cual cataliza la oxidación del grupo OH 4', para formar un aldehído utilizando una molécula de oxígeno diatómico, y produciendo peróxido de hidrógeno como subproducto.
En E. coli la primera rama de la vía se encuentra catalizada por las enzimas codificadas por los genes epd, pdxB, serC y pdxA. Esta vía comparte similitudes mecanísticas y homología con las tres enzimas en la síntesis de la serina (serA, homóloga de pdxB; serC, serB; epd es un homólogo de gap), lo cual apunta a un origen evolutivo compartido entre ambas vías metabólicas.[10] En varias especies hay dos homólogos del gen serC de E. coli, por lo general uno en el operon ser, y otro en el operón pdx, en cuyo caso es llamado pdxF.

Se ha encontrado también una vía serendípica en E. coli que puede suprimir la auxotrofía causada por la delección de pdxB (el gen que codifica la enzima eritronato 4 fosfato deshidrogenasa). Esta vía accidental es muy ineficiente, pero resulta posible gracias a la promiscuidad enzimática de varias enzimas. Esta vía comienza con 3-fosfohidroxipiruvato (3PHP) -el producto del gen serA que participa en la biosíntesis de la serina)- y no requiere de eritronato 4-fosfato. El 3PHP se desfosforila, produciendo un compuesto inestable que se descarboxila espontáneamente (de allí la presencia de fosfato en la síntesis de serina), para formar glicaldehido. El glicaldehido se condensa con glicina y el producto fosforilado resulta ser 4-fosfohidroxitreonina (4PHT), el sustrato canónico de la 4-PHT deshidrogenasa (pdxA).[8]
Biosíntesis independiente de DXP
La ruta biosintética de PLP independiente de DXP consiste de un único paso catalizado por la PLP-sintasa, una enzima compuesta por dos subunidades: PdxS y PdxT. La subunidad PdxS cataliza la condensación de ribulosa 5-fosfato, gliceraldehído 3-fosfato y amonio para producir PLP. La subunidad PdxT produce el amonio necesario para la condensación a partir de glutamina. La PdxS es un barril (β/α)8 (conocido también como barril TIM) que forma un dodecámero.[11]
Síntesis prebiótica
La amplia utilización del PLP en el metabolismo central, especialmente en la biosíntesis de aminoácidos, y su actividad en ausencia de enzimas, sugieren que el PLP podría haber sido un compuesto prebiótico.[12] De hecho, calentando NH
3 y glicoaldehído, espontáneamente se forman varios tipos de piridinas, incluyendo piridoxal.[12] Bajo ciertas condiciones, el PLP puede formarse a partir de cianoacetileno, diacetileno, monóxido de carbono, hidrógeno, agua y ácido fosfórico.[13]
Inhibidores
Se conocen varios inhibidores de las enzimas PLP.
Un tipo de inhibidor es el que forma un electrófilo con el PLP, causando que reaccione irreversiblemente con la lisina del sitio activo. Ejemplos de este tipo de inhibidores son los compuestos acetilénicos (por ejemplo la propargiglicina) y vinílicos (por ejemplo vinilglicina). Otro tipo de inhibidores son sustratos análogos α-metil y amino-oxy, por ejemplo el α-metilglutamato). Finalmente otro grupo de inhibidores son aquellos que siendo buenos grupos salientes ejercen un ataque nucleofílico sobre el PLP, tales como la clororanilina, la cual inhibe un gran número de enzimas.[7]
Ejemplos de inhibidores:
- AlaP (alanina fosfonato) inihibe a las alanina racemasas, pero carece de especificidad, lo que ha llevado al diseño de nuevos inhibidores de la ALR.[14]
- Gabaculina y Vigabatrina inhiben a la GABA aminotransferasa
- Canalina y 5-fluorometilornitina inhiben a la ornitina aminotransferasa
- Amino-oxy SAM inhibe a la 1-aminociclopropano-1-carboxilato sintasa (ACC sintasa)
Etimología
Las siglas PLP provienen de pyridoxal 5' phosphate (piridoxal 5 fosfato en inglés), mientras que las siglas PMP son de pyridoxamine 5' phosphate (piridoxamina 5 fosfato). Por lo tanto la L en PLP no indica un centro quiral levorrotatorio.
Los nombres comunes piridoxamina (un grupo aminometilo en la posición 4), piridoxal (un grupo carbaldehído) y piridoxina (un alcaloide, posee hidroximetilo), deriva de piridina + -oxi- + un sufijo IUPAC. Piridina, por otro lado, proviene del griego πῦρ, πυρός (pyr, pyros, fuego) + -idi + -ina.
Debería notarse que el infijo -oxi- en el nombre de la piridoxina no se encuentra basado en la nomenclatura Hantzsch-Widman (el anillo heterocíclico simplemente es una piridina modificada), y que la raíz pyr- no indica que se trata de un anhídrido ácido dimérico, a diferencia de lo que ocurre con el pirofosfato.
Véase también
- L-aminoácido-aromático descarboxilasa
- Ornitina descarboxilasa
Referencias
- Número CAS
- Calculated using Advanced Chemistry Development (ACD/Labs) Software V11.02 (© 1994-2011 ACD/Labs)
- Toney, M. D. "Reaction specificity in pyridoxal enzymes." Archives of biochemistry and biophysics (2005) 433: 279-287.
- Samuel, G. and Reeves, P. "Biosynthesis of O-antigens: genes and pathways involved in nucleotide sugar precusor synthesis and O-antigen assembly." Carbohydrate research (2003) 338:2503-2519.
- Cook P. D., Thoden J.B. and Holden H. M. "The structure of GDP-4-keto-6-deoxymannose-3-dehydratase: a unique coenzyme B6-dependent enzyme." Protein Science (2006) 15:2093-2106.
- N. M. F. S. A. Cerqueira, P. A. Fernandes, M. J. Ramos (2011). «Computational Mechanistic Studies Addressed to the Transimination Reaction Present in All Pyridoxal 5′-Phosphate-Requiring Enzymes». Journal of Chemical Theory and Computation 7 (5): 1356-1368. doi:10.1021/ct1002219.
- Eliot, A. C.; Kirsch, J. F. (2004). «PYRIDOXALPHOSPHATE ENZYMES: Mechanistic, Structural, and Evolutionary Considerations». Annual Review of Biochemistry 73: 383-415. PMID 15189147. doi:10.1146/annurev.biochem.73.011303.074021.
- Fitzpatrick, T. B.; Amrhein, N.; Kappes, B.; Macheroux, P.; Tews, I.; Raschle, T. (2007). «Two independent routes of de novo vitamin B6 biosynthesis: Not that different after all». Biochemical Journal 407 (1): 1-13. PMID 17822383. doi:10.1042/BJ20070765.
- Sakai, A.; Kita, M.; Tani, Y. (2004). «Recent progress of vitamin B6 biosynthesis». Journal of nutritional science and vitaminology 50 (2): 69-77. PMID 15242009. doi:10.3177/jnsv.50.69.
- Lam, H. M.; Winkler, M. E. (1990). «Metabolic relationships between pyridoxine (vitamin B6) and serine biosynthesis in Escherichia coli K-12». Journal of bacteriology 172 (11): 6518-6528. PMC 526841. PMID 2121717.
- Zhu, J.; Burgner, J. W.; Harms, E.; Belitsky, B. R.; Smith, J. L. (2005). «A New Arrangement of (α/β)8 Barrels in the Synthase Subunit of PLP Synthase». Journal of Biological Chemistry 280 (30): 27914-27923. PMID 15911615. doi:10.1074/jbc.M503642200.
- Austin, S. M.; Waddell, T. G. (1999). «Prebiotic synthesis of vitamin B6-type compounds». Origins of life and evolution of the biosphere : the journal of the International Society for the Study of the Origin of Life 29 (3): 287-296. PMID 10389266.
- Aylward, N.; Bofinger, N. (2006). «A plausible prebiotic synthesis of pyridoxal phosphate: Vitamin B6 – A computational study». Biophysical Chemistry 123 (2–3): 113-121. PMID 16730878. doi:10.1016/j.bpc.2006.04.014.
- Anthony, K. G.; Strych, U.; Yeung, K. R.; Shoen, C. S.; Perez, O.; Krause, K. L.; Cynamon, M. H.; Aristoff, P. A.; Koski, R. A. (2011). «New Classes of Alanine Racemase Inhibitors Identified by High-Throughput Screening Show Antimicrobial Activity against Mycobacterium tuberculosis». En Ahmed, Niyaz, ed. PLoS ONE 6 (5): e20374. PMC 3102704. PMID 21637807. doi:10.1371/journal.pone.0020374.