Métodos de ácidos nucleicos
Métodos de ácidos nucleicos son las técnicas utilizadas para estudiar ácidos nucleicos: ADN y ARN.
Purificación
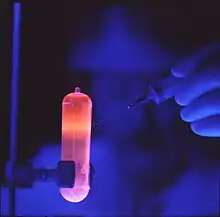
Un ADN purificado, de color naranja fluorescente bajo la luz UV, siendo extraído con una jeringa y utilizado para estudios de biología molecular. El ADN purificado, en un gradiente de cloruro de cesio, se une al colorante bromuro de etidio, que absorbe la luz ultravioleta y hace que el ADN presente fluorescencia naranja. Esta visualización de una única banda de ADN facilita el aislamiento y la extracción del ADN para futuros estudios de biología molecular.
- Extracción de ADN
- Extracción fenol–cloroformo
- Purificación con minicolumnas
- Extracción de ARN
- Método de boom
- Coeficiente síncrono de arrastrar alteración (SCODA) purificación de ADN
Cuantificación
- Abundancia en peso: espectroscopia para la cuantificación de ácidos nucleicos
- Abundancia absoluta en número: PCR en tiempo real (PCR cuantitativa)
- Alto rendimiento de abundancia relativa: Chip de ADN
- Alto rendimiento de abundancia absoluta: Análisis en serie de la expresión génica (SALVIA)
- Tamaño: electrophoresis en gel
Síntesis
- De novo: síntesis de oligonucleotidos
- Amplificación: reacción en cadena de la polimerasa (PCR)
Cinética
- Resonancia de plasmón de superficie multiparamétrica[1][2]
- Interferometría de polarización dual[3]
- Microbalanza de cristal de cuarzo con control de disipación (QCM-D)[4]
Función del gen
Otros
- Secuenciación por bisulfito
- Secuenciación del ADN
- Clonación de expresión
- Hibridación fluorescente in situ (FISH)
- Lab on a chip (LOC)
- Comparación de programas informáticos de simulación de ácidos nucleicos
- Northern blot
- Ensayo de corrimiento nuclear
- Radiactividad en las ciencias de vida
- Southern blot
- Centrifugación diferencial (gradiente de sacarosa)
- Ensayo de inhibición de la extensión del cebador
- Varios métodos bioinformaticos, vistos en lista de software de predicción de estructura de ARN
Véase también
- CSH Protocolos
- Protocolos actuales
Referencias
- Tang, Wei; Hu, Shichao; Wang, Huaming; Zhao, Yan; Li, Na; Liu, Feng (23 de septiembre de 2014). «A universal molecular translator for non-nucleic acid targets that enables dynamic DNA assemblies and logic operations». Chem. Commun. 50 (92): 14352-14355. PMID 25295484. doi:10.1039/C4CC07041K.
- Ihalainen, Petri; Pettersson, Fredrik; Pesonen, Markus; Viitala, Tapani; Määttänen, Anni; Österbacka, Ronald; Peltonen, Jouko (7 de marzo de 2014). «An impedimetric study of DNA hybridization on paper-supported inkjet-printed gold electrodes». Nanotechnology 25 (9): 094009. Bibcode:2014Nanot..25i4009I. PMID 24522116. doi:10.1088/0957-4484/25/9/094009.
- Berney, H.; Oliver, K. (2005). «Dual polarization interferometry size and density characterisation of DNA immobilisation and hybridisation». Biosensors and Bioelectronics 21 (4): 618-626. PMID 16202875. doi:10.1016/j.bios.2004.12.024.
- Dixon, Matthew C. (July 2008). «Quartz Crystal Microbalance with Dissipation Monitoring: Enabling Real-Time Characterization of Biological Materials and Their Interactions». Journal of Biomolecular Techniques 19 (3): 151-158. PMC 2563918. PMID 19137101.
- Hannon, Gregory J. (July 2002). «RNA interference». Nature (en inglés) 418 (6894): 244-251. ISSN 1476-4687. doi:10.1038/418244a.
Este artículo ha sido escrito por Wikipedia. El texto está disponible bajo la licencia Creative Commons - Atribución - CompartirIgual. Pueden aplicarse cláusulas adicionales a los archivos multimedia.