Variantes de SARS-CoV-2
El coronavirus de tipo 2 causante del síndrome respiratorio agudo severo (SARS-CoV-2), el virus que causa la enfermedad por coronavirus 2019 (COVID-19), posee múltiples variantes notables que corresponden a alteraciones puntuales a nivel de sus diferentes proteínas virales con aparente importancia particular. Las mutaciones o cambios en el ARN viral son detectables a través de la secuenciación genómica completa. Se cree que la secuencia WIV04/2019 fue la primera en infectar a los humanos, y es conocida como "secuencia cero".[2]
| SARS-CoV-2 | ||
|---|---|---|
.jpg.webp) Micrografía electrónica de transmisión de viriones de SARS-CoV-2, aislados desde un paciente. Imagen coloreada, para resaltar los virus. | ||
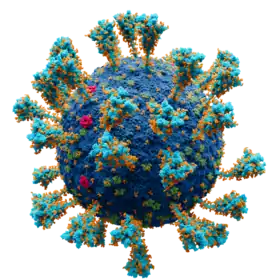 Imagen generada por ordenador de un virión del SARS-CoV-2
Envoltura vírica, compuesta por lípidos Proteína S o espícula viral Proteína E o de la envoltura Proteína M o de la membrana Glucosa | ||
| Taxonomía | ||
| Dominio: | Riboviria | |
| Reino: | Orthornavirae | |
| Filo: | Pisuviricota | |
| Clase: | Pisoniviricetes | |
| Orden: | Nidovirales | |
| Suborden: | Cornidovirineae | |
| Familia: | Coronaviridae | |
| Subfamilia: | Orthocoronavirinae | |
| Género: | Betacoronavirus | |
| Subgénero: | Sarbecovirus | |
| Especie: | Coronavirus relacionado con el síndrome respiratorio agudo grave | |
| Subespecie: | Coronavirus 2 del síndrome respiratorio agudo grave[1] | |
| Clasificación de Baltimore | ||
| Grupo: | IV (Virus ARN monocatenario positivo) | |
| Clasificación de Baltimore | ||
| ||
| Sinonimia | ||
| ||
Miles de genomas de SARS-CoV-2 muestreados a nivel mundial están disponibles de forma pública.[3]
Nomenclaturas
| Sublinajes de Rambaut et al. | Notas (Rambaut et al.)[4] | Clados de Nextstrain | Clados de GISAID | Variantes o mutaciones notables |
|---|---|---|---|---|
| A.1–A.6 | 19B | S | ||
| B.3–B.7, B.9, B.10, B.13–B.16 | 19A | L | ||
| O[nota 2] | ||||
| B.2 | V | |||
| B.1 | B.1.5–B.1.72 | 20A | G | El linaje B.1 incluye a las variantes con la mutación D614G |
| B.1.9, B.1.13, B.1.22, B.1.26, B.1.37 | GH | |||
| B.1.3–B.1.66 | 20C | Incluye 501.V2 alias 20C/501Y.V2 o linaje B.1.351 | ||
| B.1.1 | 20B | GR | Incluye VOC-202012/01 alias 20B/501Y.V1 o linaje B.1.1.7, B.1.1.207 y B.1.1.284 | |
| B.1.177 | 20A.EU1[5] | GV[nota 2] | ||
Si bien hay muchos miles de variantes de SARS-CoV-2,[7] también hay agrupaciones mucho más grandes llamados clados. Se han propuesto varias nomenclaturas para los diferentes clados del SARS-CoV-2, pero no se ha aceptado universalmente ninguna nomenclature para los linajes evolucionarios de SARS-CoV-2,[8] pero a enero de 2021, la OMS está trabajando en un "estándar de nomenclatura para las variantes de [SARS-CoV-2] que no haga referencia la localización geográfica".[9]
Si bien hay muchos miles de variantes del SARS-CoV-2,[10] los subtipos del virus pueden ser puestos en agrupaciones mucho más grandes como linajes o clados. Se han propuesto varias nomenclaturas diferentes para estos subtipos.
- Hasta diciembre de 2020, GISAID—refiriéndose al SARS-CoV-2 como hCoV-19[11]— identificó siete clados (O, S, L, V, G, GH y GR).[12][13]
- También hasta diciembre de 2020, Nextstrain identificó cinco (19A, 19B, 20A, 20B, and 20C).[14]
- En un artículo de la edición de noviembre de 2020 de la International Journal of Infectious Diseases, Guan et al. identificaron cinco clados globales (G614, S84, V251, I378 and D392).[15]
- Rambaut et al. propusieron que se empleen los linajes para los subtipos de SARS-CoV-2 en un artículo de 2020 en Nature Microbiology;[16] Hasta diciembre de 2020, se han identificado dos linajes raíz (A y B) de los que descienden otros con evidencia filogenética a los que se les asigna un valor numérico (ej. linajes A.1 o B.2), estableciéndose en conjunto cinco linajes principales (A, B, B.1, B.1.1 y B.1.177).[17] A principios de 2020, B.1 es el linajes gobal predominantemente conocido y ha sido subdividido en más de 70 sublinajes.
Un linaje activo es aquel que se ha documentado en el último mes y uno inactivo es el que no ha sido visto durante más de tres meses.
Nueva nomenclatura de la OMS
A partir del 31 de mayo del 2021 se decide utilizar el alfabeto griego para denominar a las variantes de COVID19, uno de los argumentos es desestigmatizar el vínculo de la variante con el país en donde se descubrió.[18]
La aparición de variantes que suponían un mayor riesgo para la salud pública mundial, a finales de 2020, hizo que se empezaran a utilizar las categorías específicas, con el fin de priorizar el seguimiento y la investigación a escala mundial y, en última instancia, orientar la respuesta a la pandemia de COVID-19.[19]
Nu y Xi
La Organización Mundial de la Salud decidió omitir el uso de la letra griega Nu para nombrar a alguna variante debido a que su pronunciación asemeja a la palabra en inglés new, (en español: nuevo), lo que podría causar que la variante sea confundida con un virus totalmente nuevo. Adicionalmente también omitió el uso de la letra griega Xi porque puede ser interpretada como el apellido del dirigente chino Xi Jinping.[20]
Variantes de gran consecuencia
(VHC, por sus siglas en inglés).
Variantes de preocupación
Las variantes preocupantes (VOC, por sus siglas en inglés) son aquellas que cumplen con los criterios para ser definidas como variantes de interés, pero que, tras una evaluación comparativa, se ha demostrado que está asociada a uno o más de los siguientes cambios en un grado que resulte significativo para la salud pública mundial:
- Aumento de la transmisibilidad o cambio perjudicial en la epidemiología de la COVID-19
- Aumento de la virulencia o cambio en la presentación clínica de la enfermedad
- Disminución de la eficacia de las medidas sociales y de salud pública o de los medios de diagnóstico, las vacunas y los tratamientos disponibles.
Alfa (linaje B.1.1.7)
La VUI-202012/01|Variante de Preocupación 202012/01 (VOC-202012/01),[21] previamente conocida como la primera Variante En Investigación en Diciembre de 2020 (VUI – 202012/01)[22] y también como linaje B.1.1.7 ó 20B/501Y.V1,[4][23][24] y comúnmente como Variante británica ,se detectó por primera vez en octubre de 2020 durante la pandemia de COVID-19 en el Reino Unido a partir de una muestra tomada el mes anterior.[25] Desde ese entonces, sus odds de prevalencia se han duplicado cada 6.5 días, el presunto intervalo generacional —tiempo mínimo para generar descendencia—.[26][27] Este se correlaciona con un aumento significativo en la tasa de infección por COVID-19 en el Reino Unido, asociado en parte con la mutación N501Y.
Beta (linaje B.1.351)
La variante 501.V2, también conocida como linaje 501Y.V2,[23] 20C/501Y.V2 ó B.1.351, fue detectada por primera vez en Sudáfrica y notificada por el Departamento de Salud de Sudáfrica el 18 de diciembre de 2020.[28] Los investigadores y funcionarios informaron que la prevalencia de la variante era mayor entre los jóvenes sin condiciones de salud subyacentes y, en comparación con otras variantes, conlleva de manera más frecuente a enfermedad grave en esos casos.[29][30] El Departamento de Salud de dicho país también puntualizó que la variante puede estar impulsando la segunda ola de la pandemia de COVID-19 en el país debido a que se propaga a un ritmo más rápido que otras variantes anteriores del virus.[28][29]
Los científicos observaron que la variante posee varias mutaciones que le permiten unirse más fácilmente a las células humanas debido a las siguientes tres mutaciones en el dominio de unión al receptor (RBD) en la glicoproteína de la espícula viral (spike): N501Y,[28][31] K417N y E484K.[32][33] También se ha detectado la mutación N501Y en el Reino Unido.[28][34]
Gamma (linaje P.1)
El linaje B.1.1.248, o variante P1[35] o brasileña,[36] fue detectado en Tokio el 6 de enero de 2021 por el Instituto Nacional de Enfermedades Infecciosas (NIID) y se encontró en cuatro personas que llegaron a Tokio tras viajar desde el estado de Amazonas el 2 de enero de 2021.[37] La Fundación Oswaldo Cruz —una institución estatal brasileña—, ha confirmado la suposición de que la variante estaba circulando en la selva amazónica.[38]
Esta variante de SARS-CoV-2 tiene 12 mutaciones en su proteína spike, incluidas N501Y y E484K.[39]
Delta (linaje B.1.617.1 y subvariantes)
El linaje B.1.617, también conocida como VUI (Variant Under Investigation)-21APR-01,[40] fue inicialmente identificada en la India el 5 de octubre del 2020 (de acuerdo a Professor Sharon Peacock, PhD, CBE, FMedSci, Director of the COVID-19 Genomics UK Consortium),[41] y es una variante con doble mutación.[42][43][44]
En 2021 se descubrieron las subvariantes AY.1, AY.4 y AY.4.2.
Ómicron (linaje B.1.1.529 y subvariantes)
La variante Ómicron, también conocida como linaje B.1.1.529, surgió muy probablemente en Botsuana y se detectó por primera vez en Sudáfrica en noviembre de 2021 y la OMS la designó como variante de preocupación el 26 de noviembre de 2021.
En Australia fue detectada en diciembre de 2021 la subvariante BA.2 , se cree que pudo haber surgido en China o la India, ya que se encontraron muestras que no fueron detectadas debido a las mutaciones que complicarían su detección.
Durante el año 2022 y 2023, se dio el surgimiento de múltiples subvariantes híbridas de ómicron, una de las de mayor importancia es XE.
Variantes de interés
Son consideradas variantes de interés (VOI, por sus siglas en inglés) aquellas que presentan cambios en el genoma que, según se ha demostrado o se prevé, afectan a características del virus como su transmisibilidad, la gravedad de la enfermedad que causa y su capacidad para escapar a la acción del sistema inmunitario, ser detectado por medios diagnósticos o ser atacado por medicamentos; y según se ha comprobado, dan lugar a una transmisión significativa en medio extrahospitalario o causan varios conglomerados de COVID-19 en distintos países, con una prevalencia relativa creciente y ocasionando números cada vez mayores de casos con el tiempo, o bien que presentan, aparentemente, otras características que indiquen que pueden entrañar un nuevo riesgo para la salud pública mundial.
Cluster 5
El Cluster 5, también denominado ΔFVI-spike por el Instituto Serológico Estatal Danés (SSI),[45] fue descubierto en el norte de Jutlandia en Dinamarca y se cree que se transmitió de visones a humanos a través de granjas de visones. El 4 de noviembre de 2020, se anunció que la población de visones en Dinamarca sería sacrificada para prevenir la posible propagación de esta mutación y reducir el riesgo de que ocurran nuevas mutaciones. Se implementaron restricciones de viaje y cuarentena en siete municipios del norte de Jutlandia para prevenir la posible propagación de esta mutación, lo que pudo haber comprometido las respuestas nacionales o internacionales a la pandemia de COVID-19. Para el 5 de noviembre de 2020, se habían detectado unos 214 casos humanos relacionados con el visón.[46]
La Organización Mundial de la Salud (OMS) ha indicado que el clúster 5 posee una "sensibilidad moderadamente disminuida a los anticuerpos neutralizantes".[47] El SSI advirtió que la mutación podría reducir el efecto de las vacunas COVID-19 en desarrollo, aunque era muy poco probable que las vuelva ineficaces. En consecuencia a la cuarentena y la aplicación de pruebas masivas, el SSI anunció el 19 de noviembre de 2020 que era muy probable que el Cluster 5 se hubiera extinguido por completo, por lo que ya no es de interés.[48]
Épsilon (linaje B.1.427 / B.1.429)
Denominada como Épsilon.
Zeta (linaje P.2 )
Denominada como Zeta.
Eta (linaje B.1.525 )
Denominada como Eta.
Theta (linaje P.3 )
Denominada como Theta.
Iota (linaje B.1.526 )
Denominada como Iota.
Kappa (linaje B.1.617.1 )
Denominada como Kappa.
Lambda (linaje C.37)
La variante Lambda, también conocida como linaje C.37, se detectó por primera vez en Perú en agosto de 2020 y fue designada por la OMS como una variante de interés el 14 de junio de 2021. Se extendió a al menos 30 países alrededor el mundo y, a partir de julio de 2021, se desconoce si es más infecciosa y resistente a las vacunas que otras cepas.
Mu (linaje B.1.621)
La variante Mu, también conocida como linaje B.1.621, se detectó por primera vez en Colombia en enero de 2021 y la OMS la designó como variante de interés el 30 de agosto de 2021. Ha habido brotes en América y Europa.
Variantes bajo seguimiento
Por sus siglas en inglés (VUM), son variantes a las que se les monitoreo de cerca, pero que por el poco peligro que representaban no se les asignó una letra griega.
Linaje B.1.1.207
La secuenciación realizada por el Centro Africano de Excelencia para la Genómica de Enfermedades Infecciosas en Nigeria descubrió una variante con la mutación P681H, compartida en común con VOC-202012/01. Secuenciada por primera vez en agosto de 2020,[49] las implicaciones para la transmisión y la virulencia no están claras, pero ha sido catalogada como una variante emergente por los Centros para el Control de Enfermedades de EE. UU.[50] No comparte otras mutaciones con VOC-202012/01 y para finales de diciembre de 2020, esta variante representó alrededor del 1% de los genomas virales secuenciados en Nigeria.[49]
Sumario

Variante preocupante (o VOC, por sus siglas en inglés) para el SARS-CoV-2 (que causa el COVID-19) es una categoría utilizada para las variantes del virus donde las mutaciones en su receptor RBD (Receptor Binding Domain) de la proteína de la espícula aumentan sustancialmente la afinidad de unión en el complejo RBD-hACE2, y que se relacionan con una rápida propagación de los contagios entre humanos[52]. Actualmente no hay variantes consideradas de preocupación por la OMS[53].
| Descubrimiento | Categoría máxima alcanzada | Denominaciones | Linaje | Cambios | Notas y referencias | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Posible origen | Primera detección | Muestra más temprana documentada | Denominación de la OMS | Denominaciones científicas | Denominaciones populares | Mutaciones notables | Transmisibilidad | Severidad | Inmunogenicidad | Antigenicidad | |||
| Octubre de 2020 | VOC | Alfa α | B.1.1.7 / 20B / 501Y.V1 / GR | Británica | B.1 | N501Y 69–70del P681H | [54] | ||||||
| Diciembre de 2020 | VOC | Beta ϐ | B.1.351 / 501.V2 / 20C / 501Y.V2 / GH | Sudafricana | B.1 | N501Y K417N E484K |
Sin evidencia contundente | Evidencia de ser menos detectable | [54] | ||||
| Enero 2021 | VOC | Gamma γ | B.1.1.248 | Brasileña / Amazónica | B.1 | N501Y E484K |
Evidencia de una mayor transmisibilidad | Evidencia de ser menos detectable | [54] | ||||
| Octubre de 2020 | VOC | Delta δ | B.1.617.2 | India | B.1.617 | E484Q L425R |
[54] | ||||||
| Mayo de 2021 | VOC | Delta plus δ+ | AY | Plus | B.1.617.2 | E484Q L425R |
Sin evidencia contundente | [55] | |||||
| Noviembre de 2021 | VOC | Ómicron ο | B.1.1.529 | Botsuanesa | B.1 | R346K, L452X, F486V | Sin evidencia contudente | [56] | |||||
Linajes ómicron
| Descubrimiento | Categoría máxima alcanzada | Denominaciones | Linaje | Cambios | Notas y referencias | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Posible origen | Primera detección | Muestra más temprana | Denominaciones científicas | Denominaciones populares | Mutaciones notables | Transmisibilidad | Severidad | Inmunogenicidad | Antigenicidad | |||
| Diciembre de 2021 | VOC | BA.2 / B.1.1.529.2 | Sigilosa / Furtiva | B.1.1.529 | T19I, L24del, P25del, P26del, A27S, V2113G, T376A, R3408S | [57] | ||||||
| Diciembre de 2021 | VOC | BA.3 / B.1.1.529.3 | B.1.1.529 | R3408S | Sin evidencia contudente de la reducción de la transmisibilidad | [57] | ||||||
| Enero de 2022 | VOC | BA.4 / B.1.1.529.4 | Variantes bestia | B.1.1.529 | L452R, F486V | Sin evidencia contudente | [57] | |||||
| Febrero de 2022 | VOC | BA.5 / B.1.1.529.5 | B.1.1.529 | L452R, F486V | Sin evidencia contudente | [57] | ||||||
Una variante emergente puede haber sido etiquetada como "variante de interés".[58] Durante o después de una evaluación más completa como una "variante de interés", la variante generalmente se asigna a un linaje en el sistema de nomenclatura PANGOLIN[59] y a clados en los sistemas Nextstrain[60] y GISAID.[61] Tras una actualización de criterios de clasificación de la OMS, ya no se asignan letras griegas para las variantes de interés.
| Descubrimiento | Categoría máxima alcanzada | Denominaciones | Linaje | Cambios | Notas y referencias | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Posible origen | Primera detección | Muestra más temprana documentada | Denominación de la OMS | Denominaciones científicas | Denominaciones populares | Mutaciones notables | Transmisibilidad | Severidad | Inmunogenicidad | Antigenicidad | |||
| Marzo de 2020 | VOI | Épsilon ε | B.1.427 / B.1.429 | Californiana | B.1 | L452R, S13I,
W152C¨ |
Sin evidencia de cambio | Sin evidencia de ser menos detectable | [54] | ||||
| Abril de 2020 | VOI | Zeta ζ | P.2 | Carioca | P | E484K,
D614G, V1176F |
[54] | ||||||
| Diciembre de 2020 | VOI | Eta η | B.1.525 | Nigeriana | B.1 | E484K
F888L |
Sin evidencia contundente | Sin evidencia contundente | Sin evidencia contundente | Sin evidencia contundente | [54] | ||
| Enero 2021 | VOI | Theta θ | P.3 | Filipina | P | E484K, N501Y, D614G, P681H,
E1092K, H1101Y, V1176F, K2Q |
[54] | ||||||
| Noviembre de 2020 | VOI | Iota ι | B.1.526 | Neoyorkina | B.1 | E484K,
S477N |
Sin evidencia contundente | [54] | |||||
| Octubre de 2020 | VOI | Kappa κ | B.1.617.1 | Pakistaní | B.1.617 | E484Q L425R |
Sin evidencia de una mayor mortalidad | Sin evidencia contudente | [54] | ||||
| Diciembre de 2020 | VOI | Lambda λ | C.37 | Peruana / Andina | C | G75V, T76I, Δ246-252, L452Q, F490S, D614G, T859N | Sin evidencia contudente | Sin evidencia contudente | [54] | ||||
| Enero 2021 | VOI | Mu μ | B.1.621 | Colombiana | B.1 | T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N, Y144S, Y145N, 256I, N257Q, P258, T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N | Sin evidencia contudente | Sin evidencia contudente | [54] | ||||
Mutaciones notables
D614G
D614G es una mutación con cambio de sentido que afecta la proteína de la espícula del SARS-CoV-2, detectada en Malasia.[62][63] La frecuencia de esta mutación en la población viral ha aumentado durante la pandemia. La G (glicina) ha reemplazado al D (ácido aspártico) en muchos países, especialmente en Europa, aunque más lentamente en China y el resto de Asia oriental, lo que respalda la hipótesis de que G aumenta la tasa de transmisión, lo que es consistente con títulos virales e infectividad más altos in vitro.[2] En julio de 2020, se informó que la variante SARS-CoV-2 DG14G se había convertido en la forma dominante (y por ende, más infecciosa) en ese momento de la pandemia.[64][65][66][67] La agencia de Salud Pública de Inglaterra confirmó que la mutación D614G tenía un "efecto moderado sobre la transmisibilidad" y se estaba rastreando internacionalmente.[68]
La prevalencia global de D614G se correlaciona con la prevalencia de pérdida del olfato (anosmia) como síntoma de COVID-19, posiblemente mediada por una mayor unión de la variante G al receptor ACE2 o una mayor estabilidad proteica y, por tanto, una mayor infectividad del epitelio olfativo.[69]
Las variantes que contienen la mutación G se consideran parte del clado G por GISAID[2] y parte del clado B.1 por la herramienta Phylogenetic Assignment of Named Global Outbreak LINeages (PANGOLIN).[70]
E484K
Se ha informado que E484K es una "mutación de escape" (es decir, una mutación que mejora la capacidad de un virus para evadir el sistema inmunológico del huésped ) de al menos una forma de anticuerpo monoclonal contra SARS-CoV-2, lo que indica que puede haber un "posible cambio en la antigenicidad".[36] B.1.1.248 (Brasil/Japón) y 501.V2 (Sudáfrica) exhiben esta mutación.[36] El nombre de la mutación, E484K, se refiere a un intercambio mediante el cual el ácido glutámico (E) se reemplaza por lisina (K) en la posición 484.[71]
N501Y
N501Y denota un cambio de asparagina (N) a tirosina (Y) en la posición del aminoácido 501,[68] la agencia de Salud Pública de Inglaterra asume que ello aumenta la afinidad de unión debido a su posición en el dominio de unión al receptor de la glicoproteína espicular, que se une a la ACE2 en las células humanas;[72] los datos también apoyan la hipótesis de una mayor afinidad de unión a partir de este cambio.[72] Las variantes con N501Y incluyen a B.1.1.248 (Brasil/Japón),[36] Variante de Preocupación 202012/01 (Reino Unido), 501.V2 (Sudáfrica) y una variante de Columbus, Ohio, que se convirtió en la forma dominante del virus en Columbus a finales de diciembre de 2020 y en enero de 2021. Esta última se denominó COH.20G/501Y y parece haber evolucionado independientemente de otras variantes.[73][74]
S477G/N
Una región altamente flexible en el dominio de unión al receptor (RBD) del SARS-CoV-2, comenzando desde el residuo 475 y continuando hasta el residuo 485, se identificó utilizando métodos bioinformáticos y estadísticos en varios estudios. La Universidad de Graz[75] y la empresa de biotecnología Innophore[76] han demostrado en una publicación reciente que, estructuralmente, la posición S477 muestra la mayor flexibilidad entre ellas. [ Al mismo tiempo, S477 es hasta ahora el residuo de aminoácido intercambiado con mayor frecuencia en los RBD de mutantes de SARS-CoV-2. Mediante el uso de simulaciones de dinámica molecular de RBD durante el proceso de unión a ACE2, se ha demostrado que tanto S477G como S477N fortalecen la unión del pico de SARS-CoV-2 con el receptor ACE2. El desarrollador de vacunas BioNTech hizo referencia a este intercambio de aminoácidos como relevante con respecto al diseño futuro de la vacuna en una preimpresión publicada en febrero de 2021.
P681H
En enero de 2021, los científicos informaron en una preimpresión que la mutación 'P681H', un rasgo característico de las nuevas variantes significativas del SARS-CoV-2 detectadas en el Reino Unido (B.1.1.7) y Nigeria (B.1.1.207), muestra un aumento exponencial significativo en la frecuencia mundial, similar al 'D614G' que ahora prevalece en todo el mundo.
Estadísticas
Por variante o subvariante (Actualizado a abril de 2022)
| # | Denominación | Categoría máxima alcanzada | Muestra más temprana | Casos confirmados | Casos diarios |
|---|---|---|---|---|---|
| 1 | VOC | Noviembre 2021 | 5 230 564 | 100 secuencias | |
| 2 | VOC | Octubre 2020 | 2 235 532 | 33,078,890 secuencias | |
| 3 | VOC | Octubre 2020 | 1 125 169 | 1,405 secuencias | |
| 4 | VOC | Diciembre 2021 | 274 069 | 0 secuencias | |
| 5 | VOC | Enero 2021 | 110 285 | 2,846 secuencias | |
| 6 | VUM | Marzo 2020 | 60 789 | 34 secuencias | |
| 7 | VUM | Noviembre 2020 | 42 298 | 1 secuencia | |
| 8 | VOC | Diciembre 2020 | 40 866 | 17 secuencias | |
| 9 | VOI | Enero 2021 | 29 848 | 306 secuencias | |
| 10 | VOI | Enero 2021 | 13 101 | 39 secuencias | |
| 11 | VOI | Diciembre 2020 | 8 796 | 0 secuencias | |
| 12 | VUM | Diciembre 2020 | 8 607 | 21 secuencias | |
| 13 | VUM | Octubre 2020 | 7 686 | 13 secuencias | |
| 14 | VUM | Abril 2020 | 5 250 | 56 secuencias | |
| 15 | VUM | Enero 2021 | 619 | 0 secuencias | |
| 16 | † [48] | Noviembre 2020 | 214 | 0 secuencias | |
Véase también
- RaTG13.
- Comité Internacional de Taxonomía de Virus
- Nucleótido
- Secuenciación genómica
- Variante del 2009 del AH1N1.
- Variante 2.3.4.4b del AH5N8.
Notas
- Esta tabla es una adaptación y expansión de Alm et al., Figura 1.
- En otra fuente, GISAID nombra un conjunto de 7 clados sin el clado 'O' pero incluye un clado 'GV'.[6]
Referencias
- «Taxonomy browser (Betacoronavirus)». Centro Nacional para la Información Biotecnológica, Biblioteca Nacional de Medicina de Estados Unidos. Archivado desde el original el 20 de marzo de 2020. Consultado el 14 de marzo de 2020.
- Zhukova, A; Blassel, L; Lemoine, F; Morel, M; Voznica, J; Gascuel, O (24 de noviembre de 2020). «Origin, evolution and global spread of SARS-CoV-2». Comptes Rendus Biologie: 1-20. PMID 33274614. doi:10.5802/crbiol.29. Archivado desde el original el 21 de febrero de 2021. Consultado el 15 de enero de 2021.
- «Genomic epidemiology of novel coronavirus - Global subsampling» (en inglés). 20 de abril de 2020. Archivado desde el original el 20 de abril de 2020. Consultado el 7 de mayo de 2020.
- Rambaut, Andrew; Loman, Nick; Pybus, Oliver; Barclay, Wendy; Barrett, Jeff; Carabelli, Alesandro; Connor, Tom; Peacock, Tom; L. Robertson, David; Vol, Erik (2020), Preliminary genomic characterisation of an emergent SARS-CoV-2 lineage in the UK defined by a novel set of spike mutations, Escrito en nombre del COVID-19 Genomics Consortium UK, consultado el 20 de diciembre de 2020.
- COG-UK update on SARS-CoV-2 Spike mutations of special interest: Report 1, COVID-19 Genomics UK Consortium (COG-UK), 20 de diciembre de 2020, p. 2, archivado desde el original el 25 de diciembre de 2020, consultado el 15 de enero de 2021.
- «clade tree (from 'Clade and lineage nomenclature')». www.gisaid.org (en inglés). 4 de julio de 2020. Consultado el 7 de enero de 2021.
- Koyama, Takahiko; Platt, Daniel; Parida, Laxmi (Junio 2020). «Variant analysis of SARS-CoV-2 genomes». Bulletin of the World Health Organization 98 (7): 495-504. PMC 7375210. PMID 32742035. doi:10.2471/BLT.20.253591. «Detectamos en total 65776 variantes con 5775 variantes distintas. »
- "6.8.7 Lineage classification". Genomic sequencing of SARS-CoV-2: a guide to implementation for maximum impact on public health. p. 56. Geneva: World Health Organization. 2021. Licence: CC BY-NC-SA 3.0 IGO. ISBN 978-92-4-001844-0.
- World Health Organization (15 de enero de 2021). «Statement on the sixth meeting of the International Health Regulations (2005) Emergency Committee regarding the coronavirus disease (COVID-19) pandemic». Consultado el 18 de enero de 2021.
- {Koyama, Takahiko; Platt, Daniel; Parida, Laxmi (Junio 2020). «Análisis de variantes de los genomas del SARS-CoV-2». Boletín de la Organización Mundial de la Salud 98: 495-504. PMC 7375210. PMID 32742035. doi:10.2471/BLT.20.253591. «Detectamos un total de 65776 variantes con 5775 variantes distintas. » Parámetro desconocido
|doi-acceso=ignorado (ayuda); Parámetro desconocido|emisión=ignorado (ayuda) - Alm, E.; Broberg, E. K.; Connor, T.; Hodcroft, E. B.; Komissarov, A.B.; Maurer-Stroh, S.; Melidou, A.; Neher, R. A.; O’Toole, Áine; Pereyaslov, D.; The WHO European Region sequencing laboratories and GISAID EpiCoV group et al. (2020). «Geographical and temporal distribution of SARS-CoV-2 clades in the WHO European Region, January to June 2020». Euro Surveillance: Bulletin Europeen Sur les Maladies Transmissibles = European Communicable Disease Bulletin 25 (32). PMC 7427299. PMID 32794443. doi:10.2807/1560-7917.ES.2020.25.32.2001410.
- «hCoV-19 clades» (JPG). www.gisaid.org. Consultado el 24 de diciembre de 2020.
- Dellicour, Simon; Hong, Samuel L.; Vrancken, Bram; Chaillon, Antoine; Gill, Mandev S.; Maurano, Matthew T.; Ramaswami, Sitharam; Zappile, Paul et al. (20 de mayo de 2021). «Dispersal dynamics of SARS-CoV-2 lineages during the first epidemic wave in New York City». PLOS Pathogens (en inglés) 17 (5): e1009571. ISSN 1553-7374. doi:10.1371/journal.ppat.1009571. Consultado el 31 de mayo de 2021.
- «Nextclade». clades.nextstrain.org. Consultado el 24 de diciembre de 2020. Citado en Alm et al.
- Guan, Qingtian (2020). «A genetic barcode of SARS-CoV-2 for monitoring global distribution of different clades during the COVID-19 pandemic». International Journal of Infectious Diseases 100: 216-223. PMC 7443060. PMID 32841689. doi:10.1016/j.ijid.2020.08.052.
- Rambaut, A.; Holmes, E.C.; O’Toole, Á. (2020). «A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology». Nature Microbiology (en inglés) 5 (11): 1403-1407. PMID 32669681. doi:10.1038/s41564-020-0770-5. S2CID 220544096. Citado en Alm et al.
- «Lineages». cov-lineages.org. Consultado el 24 de diciembre de 2020.
- https://www.the-scientist.com/news-opinion/who-updates-the-nomenclature-of-sars-cov-2-variants-68837
- World Health Organization. «Seguimiento de las variantes del SARS-CoV-2». Consultado el 21 de julio de 2021.
- Faith Karimi (29 de noviembre de 2021). «Why WHO skipped two letters of the Greek alphabet in naming Omicron». CNN. Consultado el 9 de enero de 2022.
- Chand, Meera; Hopkins, Susan; Dabrera, Gavin; Achison, Christina; Barclay, Wendy; Ferguson, Neil; Volz, Erik; Loman, Nick; Rambaut, Andrew; Barrett, Jeff (21 de diciembre de 2020), Investigation of novel SARS-COV-2 variant: Variant of Concern 202012/01, Public Health England, consultado el 23 de diciembre de 2020.
- «PHE investigating a novel strain of COVID-19». Public Health England. 14 de diciembre de 2020.
- «Emerging SARS-CoV-2 Variants» (en inglés estadounidense). Centers for Disease Control and Prevention. Consultado el 4 de enero de 2021.
- Kupferschmidt, Kai (20 de diciembre de 2020). «Mutant coronavirus in the United Kingdom sets off alarms but its importance remains unclear». Science Mag (en inglés). Consultado el 21 de diciembre de 2020.
- «Covid: Ireland, Italy, Belgium and Netherlands ban flights from UK». BBC News. 20 de diciembre de 2020.
- «New evidence on VUI-202012/01 and review of the public health risk assessment». khub.net. 15 de diciembre de 2020.
- «COG-UK Showcase Event - YouTube». YouTube. Consultado el 25 de diciembre de 2020.
- «South Africa announces a new coronavirus variant». The New York Times. 18 de diciembre de 2020. Consultado el 20 de diciembre de 2020.
- Wroughton, Lesley; Bearak, Max (18 de diciembre de 2020). «South Africa coronavirus: Second wave fueled by new strain, teen 'rage festivals'». The Washington Post. Consultado el 20 de diciembre de 2020.
- Mkhize, Dr Zwelini (18 de diciembre de 2020). «Update on Covid-19 (18th December 2020)». Sudáfrica. COVID-19 South African Online Portal. Consultado el 23 de diciembre de 2020. «Nuestros médicos también nos han advertido que las cosas han cambiado y que las personas más jóvenes, previamente sanas, ahora se están enfermando gravemente. »
- Abdool Karim, Salim S. (19 de diciembre de 2020). «The 2nd Covid-19 wave in South Africa:Transmissibility & a 501.V2 variant, 11th slide». www.scribd.com.
- Statement of the WHO Working Group on COVID-19 Animal Models (WHO-COM) about the UK and South African SARS-CoV-2 new variants, World Health Organization, 22 de diciembre de 2020, consultado el 23 de diciembre de 2020.
- Lowe, Derek (22 de diciembre de 2020). «The New Mutations». In the Pipeline. American Association for the Advancement of Science. Consultado el 23 de diciembre de 2020. «Debo puntualizar que hay otra cepa en Sudáfrica que está generando preocupaciones similares. Esta tiene ocho mutaciones en la proteína Spike, con tres de ellas (K417N, E484K y N501Y) que pueden tener algún papel funcional. »
- Novel mutation combination in spike receptor binding site, GISAID, 21 de diciembre de 2020, archivado desde el original el 22 de febrero de 2021, consultado el 23 de diciembre de 2020.
- Emerging SARS-CoV-2 Variants.
- «Brief report: New Variant Strain of SARS-CoV-2 Identified in Travelers from Brazil». Japón. NIID (National Institute of Infectious Diseases). 12 de enero de 2021. Consultado el 14 de enero de 2020.
- «Japan finds new coronavirus variant in travelers from Brazil». Japón. Japan Today. 11 de enero de 2021. Consultado el 14 de enero de 2021.
- Tony Winterburn (13 de enero de 2021). «Brazil Confirms The Circulation Of A New ‘Amazon Rain Forest’ Variant Of The Coronavirus». euroweeklynews.com. Consultado el 14 de enero de 2021.
- Lovett, Samuel (14 de enero de 2021). «What we know about the new Brazilian coronavirus variant». Londres: The Independent. Consultado el 14 de enero de 2021.
- «Variants: distribution of cases data». GOV.UK (en inglés). Consultado el 24 de abril de 2021.
- «expert reaction to cases of variant B.1.617 (the ‘Indian variant’) being investigated in the UK | Science Media Centre» (en inglés británico). Consultado el 24 de abril de 2021.
- «PANGO lineages». cov-lineages.org. Consultado el 24 de abril de 2021.
- Koshy, Jacob (8 de abril de 2021). «Coronavirus | Indian ‘double mutant’ strain named B.1.617». The Hindu (en Indian English). ISSN 0971-751X. Consultado el 24 de abril de 2021.
- «Cases of new 'double mutation' Indian Covid strain thought to be spreading in UK». inews.co.uk (en inglés). 19 de abril de 2021. Consultado el 24 de abril de 2021.
- Lassaunière, Ria (11 de noviembre de 2020). «SARS-CoV-2 spike mutations arising in Danish mink and their spread to humans». Dinamarca: Statens Serum Institut. Archivado desde el original el 10 de noviembre de 2020. Consultado el 11 de noviembre de 2020.
- «Detection of new SARS-CoV-2 variants related to mink». Centro Europeo para la Prevención y Control de Enfermedades. 12 de noviembre de 2020. Consultado el 12 de noviembre de 2020.
- «SARS-CoV-2 mink-associated variant strain – Denmark». World Health Organization. 6 de noviembre de 2020. Consultado el 16 de enero de 2021.
- «De fleste restriktioner læmpes i Nordjylland» [La mayoría de las restricciones disminuyeron en el norte de Jutlandia]. Sundheds- og Ældreministeriet. 19 de noviembre de 2020.
- «Detection of SARS-CoV-2 P681H Spike Protein Variant in Nigeria». Virological (en inglés estadounidense). 23 de diciembre de 2020. Consultado el 1 de enero de 2021.
- «Coronavirus Disease 2019 (COVID-19)» (en inglés estadounidense). CDC. 2 de noviembre de 2020. Consultado el 1 de enero de 2021.
- «Seguimiento de las variantes del SARS-CoV-2». WHO. Consultado el 19 de enero de 2023.
- Shahhosseini, Nariman; Babuadze, George (Giorgi); Wong, Gary; Kobinger, Gary P. (May 2021). «Mutation Signatures and In Silico Docking of Novel SARS-CoV-2 Variants of Concern». Microorganisms (en inglés) 9 (5): 926. PMC 8146828. PMID 33925854. doi:10.3390/microorganisms9050926.
- «Declaración sobre la actualización de las definiciones de trabajo y del sistema de seguimiento de las variantes preocupantes y las variantes de interés del SARS-CoV-2». OMS. 16 de marzo de 2023. Consultado el 26 de mayo de 2023.
- «Estas son todas las variantes de la covid hasta ahora: de la delta a la lambda». Heraldo de Aragón. 1 de julio de 2021. Consultado el 19 de enero de 2023.
- «Delta plus: la nueva mutación del coronavirus que está causando un número creciente de infecciones en Reino Unido». BBC. 21 de octubre de 2021. Consultado el 19 de enero de 2023.
- «"Ómicron, la inquietante variante multimutante del coronavirus"». DW. Noviembre de 2021. Consultado el 19 de enero de 2023.
- «Omicron variant (B.1.1.529) and its sublineages: What do we know so far amid the emergence of recombinant variants of SARS-CoV-2?». National Library of Medicine PubMed Central (en inglés). Octubre 2022. Consultado el 5 de enero de 2023.
- «Variants: distribution of cases data». GOV.UK. 28 de enero de 2021. At "Differences between a Variant of Concern and Variant Under Investigation". Consultado el 19 de febrero de 2021. «Las variantes del SARS-CoV-2, si se considera que tienen propiedades epidemiológicas, inmunológicas o patogénicas preocupantes, se plantean para una investigación formal. En este punto, se designan Variante Bajo Investigación (en inglés, VUI) con un año, mes y número. Después de una evaluación de riesgos con el comité de expertos correspondiente, pueden ser designados Variante de Preocupación (VOC) ».
- Rambaut, A.; Holmes, E.C.; O’Toole, Á. (2020). «A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology». Nature Microbiology 5 (11): 1403-1407. PMC 7610519. PMID 32669681. doi:10.1038/s41564-020-0770-5.
- Bedford, Trevor (6 de enero de 2021). «Updated Nextstrain SARS-CoV-2 clade naming strategy». nextstrain.org/blog. Consultado el 19 de enero de 2021.
- «clade tree (from 'Clade and lineage nomenclature')». www.gisaid.org. 4 de julio de 2020. Consultado el 7 de enero de 2021.
- «Making Sense of Mutation: What D614G Means for the COVID-19 Pandemic Remains Unclear». CellPress. Consultado el 28 de agosto de 2020.
- «En Malasia, detectan cepa de Covid-19, 10 veces más infecciosa». Forbes. 17 de agosto de 2020. Consultado el 28 de agosto de 2020.
- Schraer, Rachel (18 de julio de 2020). «Coronavirus: Are mutations making it more infectious?». BBC News. Consultado el 3 de enero de 2021.
- «New, more infectious strain of COVID-19 now dominates global cases of virus: study» (en inglés). medicalxpress.com. Archivado desde el original el 17 de noviembre de 2020. Consultado el 16 de agosto de 2021.
- Korber, Bette; Fischer, Will M.; Gnanakaran, Sandrasegaram; Yoon, Hyejin; Theiler, James; Abfalterer, Werner et al. (2 de julio de 2020). «Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus». Cell (en inglés) 182 (4): 812-827.e19. ISSN 0092-8674. PMC 7332439. PMID 32697968. doi:10.1016/j.cell.2020.06.043.
- «SARS-CoV-2 D614G variant exhibits efficient replication ex vivo and transmission in vivo». Science 370 (6523): 1464-1468. 18 de diciembre de 2020. doi:10.1126/science.abe8499. Consultado el 14 de enero de 2021. «Una sustitución emergente de Asp614 → Gly (D614G) en la glicoproteína espículo (spike) de las cepas de SARS-CoV-2, es ahora la forma más prevalente a nivel mundial. »
- COG-UK update on SARS-CoV-2 Spike mutations of special interest: Report 1, COVID-19 Genomics UK Consortium (COG-UK), 20 de diciembre de 2020, p. 7, archivado desde el original el 25 de diciembre de 2020, consultado el 15 de enero de 2021.
- Butowt, R; Bilinska, K; Von Bartheld, CS (21 de octubre de 2020). «Chemosensory Dysfunction in COVID-19: Integration of Genetic and Epidemiological Data Points to D614G Spike Protein Variant as a Contributing Factor». ACS Chem Neurosci. 11 (20): 3180-3184. PMC 7581292. PMID 32997488. doi:10.1021/acschemneuro.0c00596.
- «cov-lineages/pangolin: Software package for assigning SARS-CoV-2 genome sequences to global lineages» (en inglés). github.com. Consultado el 2 de enero de 2021.
- Greenwood, M (15 de enero de 2020). «What Mutations of SARS-CoV-2 are Causing Concern?». News Medical. Consultado el 16 de enero de 2020.
- Chand et al. "Potential impact of spike variant N501Y" (p. 6).
- «Researchers Discover New Variant of COVID-19 Virus in Columbus, Ohio». wexnermedical.osu.edu. 13 de enero de 2021. Consultado el 16 de enero de 2021.
- Huolin Tu, et al. (15 de enero de 2021). «Distinct Patterns of Emergence of SARS-CoV-2 Spike Variants including N501Y in Clinical Samples in Columbus Ohio». www.biorxiv.org. doi:10.1101/2021.01.12.426407. Consultado el 16 de enero de 2021.
- «University of Graz». www.uni-graz.at. Consultado el 22 de febrero de 2021.
- «Coronavirus SARS-CoV-2 - what we can find out on a structural bioinformatics level». Innophore (en inglés estadounidense). 23 de enero de 2020. Consultado el 22 de febrero de 2021.