SARS-CoV-2
El coronavirus de tipo 2 causante del síndrome respiratorio agudo severo,[2] abreviado SARS-CoV-2 (del inglés severe acute respiratory syndrome coronavirus 2)[3] o SRAS-CoV-2,[2] es un tipo de coronavirus causante de la enfermedad por coronavirus de 2019,[4][5][6] cuya expansión mundial provocó la pandemia de COVID-19. Inicialmente fue llamado 2019-nCoV (en inglés: 2019-novel coronavirus, ‘nuevo coronavirus de 2019’) y también, ocasionalmente, HCoV-19 (en inglés: human coronavirus 2019).[7][8] Se descubrió y se aisló por primera vez en Wuhan, China. Tiene un origen zoonótico, es decir, que se transmitió de un huésped animal a uno humano.[9]
| SARS-CoV-2 | ||
|---|---|---|
.jpg.webp) Micrografía electrónica de transmisión de viriones de SARS-CoV-2, aislados desde un paciente. Imagen coloreada, para resaltar los virus. | ||
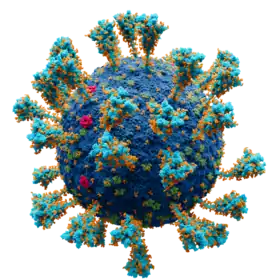 Imagen generada por ordenador de un virión del SARS-CoV-2.
Envoltura vírica, compuesta por lípidos Proteína S o espícula viral Proteína E o de la envoltura Proteína M o de la membrana Glucosa | ||
| Taxonomía | ||
| Dominio: | Riboviria | |
| Reino: | Orthornavirae | |
| Filo: | Pisuviricota | |
| Clase: | Pisoniviricetes | |
| Orden: | Nidovirales | |
| Suborden: | Cornidovirineae | |
| Familia: | Coronaviridae | |
| Subfamilia: | Orthocoronavirinae | |
| Género: | Betacoronavirus | |
| Subgénero: | Sarbecovirus | |
| Especie: | Coronavirus relacionado con el síndrome respiratorio agudo grave | |
| Subespecie: | Coronavirus 2 del síndrome respiratorio agudo grave[1] | |
| Clasificación de Baltimore | ||
| Grupo: | IV (Virus ARN monocatenario positivo) | |
| Sinonimia | ||
| ||
Es un clado dentro de la familia de los Coronaviridae, género Betacoronavirus, subgénero Sarbecovirus, especie virus SARS[10] (virus relacionado con el síndrome respiratorio agudo severo o grave).[11]
El genoma del virus está formado por una sola cadena de ARN, y se clasifica como un virus ARN monocatenario positivo. Su secuencia genética se ha aislado a partir de una muestra obtenida de un paciente afectado por neumonía en la ciudad china de Wuhan, aunque la falta de experimento de control doble ciego en la técnica de secuenciación publicada puede poner en cuestionamiento la validez científica de la técnica.[12][13][14][15][16][17] Se detectó por primera vez el 12 de noviembre de 2019. Puede producir el contagio de una persona a otra mediante las gotas de saliva expulsadas a través de la tos y el estornudo o al espirar (véase gotitas de Flügge).[18][19] Puede provocar enfermedad respiratoria aguda y neumonía grave en los seres humanos.[20]
Aunque previamente no había ningún tratamiento específico aprobado oficialmente, ya se habían desarrollado algunos antivirales existentes, así como el tratamiento con plasma convaleciente y la dexametasona, que parecen tener una mayor eficacia en el manejo de los síntomas o que parecen acortar el periodo de recuperación en poblaciones especiales.[21][22] En diciembre de 2020 comenzó una campaña de vacunación con las primeras vacunas experimentales autorizadas en emergencia, que se prolongó y mantuvo durante 2021 y 2022.[cita requerida] Fue Pfizer - BioNTech, con la vacuna Comirnaty, los laboratorios pioneros en patentar una vacuna[23] y posteriormente, los laboratorios Moderna y AstraZeneca se unieron a esta carrera por la vacuna.[24]
Historia
Existen diferentes teorías acerca de su origen, la comunidad científica internacional aún no ha podido ponerse de acuerdo; una de ellas sugiere un origen zoonótico, presumiendo ese origen, el 17 de noviembre de 2019, se pudo haber efectuado el primer contacto entre el SARS-CoV-2 y un individuo humano por infección zoonótica. La fecha ha sido estimada asumiendo un período máximo de incubación de 24 días. Esto supone que el virus se transmitió de manera silente hasta la detección oficial del primer caso confirmado.[25]
El 30 de diciembre de 2019, las autoridades sanitarias de la ciudad de Wuhan informaron sobre la aparición de veintisiete personas diagnosticadas de síndrome respiratorio agudo grave de origen desconocido; la mayor parte de los casos estaban relacionados con el Mercado Mayorista de Mariscos del Sur de China ubicado en la ciudad. El 7 de enero de 2020 las autoridades chinas declararon que habían descubierto que la causa de la enfermedad era un nuevo virus de la familia de los coronavirus que fue nombrado provisionalmente como 2019-nCoV (coronavirus de Wuhan).
El 10 de enero se anunció que se había aislado y se publicaría el primer genoma secuenciado del nuevo coronavirus.[17][26]
El 13 de enero se detectó un caso en Tailandia confirmado por pruebas de laboratorio. El 14 de enero se detectó un caso en Japón de una persona que había viajado recientemente a Wuhan. El 21 de enero se informó de la existencia de casos en Estados Unidos también en personas que habían viajado a Wuhan.[27]
Al 29 de enero de 2020 se habían descrito casos en: Bangkok (Tailandia), Tokio (Japón), Seúl (Corea del Sur), Pekín[28] (China), Shanghái[28] (China), Guangdong (China), Hong Kong[29] (China), Macao[29] (China), Estados Unidos,[30] Reino Unido, Vietnam,[31] Singapur,[32] Francia y Alemania. Hasta ese día había provocado 169 muertes, principalmente en Wuhan y alrededores.

El 30 de enero de 2020, el Comité de Emergencias del Reglamento Sanitario Internacional (2005) de la Organización Mundial de la Salud (OMS) declaró la situación como emergencia de salud internacional por el brote de SARS-CoV-2.[33] Hasta ese día se habían producido 7711 casos confirmados en la República Popular China, con 170 víctimas mortales. En el resto del mundo se habían confirmado 83 casos en 18 países, casi todos los pacientes procedían de China. Solamente 7 no tenían antecedentes de haber viajado recientemente a este país.[cita requerida]
El 13 de febrero se habían notificado 46 997 casos a nivel mundial, de los que 46 550 correspondían a la China continental y 447 en otros países. El número de fallecidos ascendía a 1339.[34]
El 11 de marzo, la Organización Mundial de la Salud caracterizó como pandemia a la infección por SARS-CoV-2 y la enfermedad denominada COVID-19,[35][36] mientras los casos confirmados a nivel mundial superaban los 118 000 en 114 países y el número de fallecidos ascendía a 4291.
Al 7 de abril, el GenBank contaba ya con más de quinientas secuencias genómicas del virus a partir de muestras colectadas en diferentes partes del mundo, como parte de un impulso colaborativo para facilitar su investigación y encontrar soluciones contra la infección.[37]
Virología

En la taxonomía de los virus, los coronavirus se corresponden con la subfamilia Orthocoronavirinae, que está incluida dentro de la familia Coronaviridae. Esta subfamilia se compone de cuatro géneros, según su estructura genética: Alphacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus. El SARS-CoV-2 se clasifica dentro del género Betacoronavirus.[38]
Los coronavirus forman una gran familia de virus. Tanto los alfacoronavirus como los betacoronavirus provocan distintas enfermedades en diferentes especies de mamíferos: infecciones respiratorias en humanos y procesos de gastroenteritis en algunos animales.[39] Existen CoVs que circulan globalmente en la población humana, y en raras ocasiones, los coronavirus procedentes de otros mamíferos pueden mutar e infectar al ser humano para después propagarse de una persona a otra, causando desde un simple resfriado común hasta enfermedades más graves como el síndrome respiratorio agudo grave (SARS) que apareció por primera vez en noviembre de 2002 en la provincia de Cantón (China) y el síndrome respiratorio de Oriente Medio (MERS) que fue identificado por primera vez en el año 2012 en Arabia Saudita. Solo se habían descubierto seis CoVs relacionados con enfermedades en humanos.[40] El coronavirus de Wuhan (SARS-CoV-2) sería el séptimo:
- HCoV-229E. Se descubrió en 1966. Provoca en humanos una enfermedad respiratoria similar a una gripe.
- HCoV-0C43. Se descubrió en 1967. También provoca en humanos una enfermedad respiratoria similar a una gripe.
- SARS-CoV. Originó la epidemia del síndrome respiratorio agudo grave. Se descubrió en noviembre de 2002, en la provincia de Cantón, China.
- HCoV-NL63. Se identificó en los Países Bajos en 2003, en un niño con bronquiolitis.
- HCoV-HKU1. Se descubrió en 2005 en dos pacientes de la ciudad china de Hong-Kong.
- MERS-CoV. Provoca el síndrome respiratorio de Oriente Medio, enfermedad infecciosa que se identificó por primera vez en 2012 en Arabia Saudita.
Filogenia

La secuenciación de genomas de SARS-CoV-2 en personas infectadas por todo el mundo ha permitido el desarrollo de un árbol filogenético de sus distintas cepas. La comparación de más de 4700 genomas virales disponibles en repositorios públicos como GISAID o Nextstrain, permitió la caracterización viral basada es sus mutaciones diferenciales.
Identidad genética
Los virus analizados por todo el globo presentaban una alta identidad de secuencias, con un 99.98 % de similitud.[25] Se compararon con los coronavirus precursores más cercanos en otras especies como RaTG13 de murciélago (similitud del 98 %, 1100 pb diferentes), o en pangolín (similitud 92 %, 1600 pb diferentes).
Tras las primeras infecciones el virus se dividió en dos macro-haplogrupos, el A (afectación internacional) y el B (afectación principalmente Asiática). Aunque ambas cepas aparecieron prácticamente a la vez, se ha demostrado que lo más probable es que la cepa original sea la A. Esto se explica porque la cepa B1 tendría que haber existido dos meses antes que la A, lo que es improbable, ya que esta no se detectó hasta el 19 de enero de 2020.[25]
Cada haplogrupo y los sub-haplogrupos correspondientes poseen mutaciones puntuales en posiciones características que los diferencian del resto. El virus cambia a un ritmo aproximado de 1-2 mutaciones por mes, y cuando acumula más de dos mutaciones no existentes en la cepa original, las cepas pasan a considerarse como novedosas. De esta manera, el haplogrupo B, por ejemplo, tiene mutaciones en los aminoácidos [C8782T, C18060T, T28144C] que lo diferencian del A,[25] y así para todos los distintos sub-haplogrupos.
Se ha especulado sobre ciertas mutaciones como la transversión A23403T, que provoca el cambio de un aminoácido de aspártico (Asp, D) a una glicina (Gly, G) en la posición 614 de la proteína Spike viral.[42] Parece que esta mutación le aporta una ventaja de transmisibilidad, lo que explicaría su rápida propagación por Europa a principio de febrero de 2020 y la selección natural de sub-haplogrupos más virulentos sobre otros. Esta mutación parece afectar a la habilidad de transmisión del virus al alterar la interacción entre los dominios de la proteína Spike. Esto puede suponer un problema para el desarrollo de vacunas por los posibles cambios en la antigenicidad de la misma.
Supercontagiadores y efecto fundador
El patrón de propagación de SARS-CoV-2 parece estar basado en la transmisión por supercontagiadores.[cita requerida] Estos son individuos con una capacidad superior de transmitir el virus, los cuales suponen una ventaja para el mismo. Cuando uno de estos individuos llega a una zona nueva comienza a transmitir el virus rápidamente con un patrón de propagación característico.
Los focos por supercontagiadores alcanzan incidencias altas en la población afectada, aparecen en un período breve de tiempo (normalmente días), y son específicos de cada región infectada. Como los aparecidos en Europa (A2a2), Estados Unidos (B1a1), Reino Unido - Australia (A2a4), o en Asia (B).[25]
En los focos por supercontagiadores existe una variedad alélica escasa, puesto que todos los individuos se contagian desde una única cepa de virus. Esto se conoce como efecto fundador, en el que ese virus en concreto sirve como único origen para la formación de nuevos sub-haplogrupos. Por ejemplo, el haplotipo A2a2a que se encuentra únicamente en Islandia.[25]
Así pues, estos factores contribuyeron a la rápida propagación del virus y al establecimiento de la pandemia del COVID-19.
Estructura

El tamaño de los viriones de SARS-CoV-2 es de aproximadamente 50 a 200 nm de diámetro, y su genoma es de ARN monocatenario de sentido positivo.[43] La secuencia del betacoronavirus de Wuhan muestra semejanzas con los betacoronavirus encontrados en murciélagos, pero son genéticamente distintos de otros coronavirus como el SARS-CoV y el MERS-CoV.[17][44]

Su secuencia de ARN es de aproximadamente treinta mil nucleótidos de longitud. Consta de cuatro genes para las proteínas estructurales características de los coronavirus que se designan con las letras S (homotrímero de glicoproteína cuyo ensanchamiento distal de sus pliegues forma las puntas de la superficie), E (pequeña proteína de la envoltura), M (proteína de la matriz que une la envoltura con el núcleo vírico) y N (fosfoproteína de la nucleocápside), además de los ORFs que codifican proteínas no estructurales incluyendo las enzimas que aparecen durante su ciclo reproductivo intrahospedero.[14][26][45]
Fuente del virus
Se cree que la fuente del virus es animal. Es probable que el brote se haya originado por contacto directo con animales en el mercado de la ciudad de Wuhan.[39] Una vez que el virus se encuentra en una persona puede transmitirse a otra.[39][46] Aunque no se ha logrado averiguar el reservorio específico, se han propuesto diversas posibilidades, entre ellas murciélagos, serpientes o pangolines.[47] Por ejemplo, el 22 de enero de 2020, el Journal of Medical Virology publicó un informe con el análisis genómico del virus, que refleja que las serpientes de la zona de Wuhan, infectadas con el virus por murciélagos, son el reservorio más probable del virus; sin embargo, se requieren más investigaciones para dar por segura esta posibilidad.[48]
El 26 de enero, se inició una investigación para estudiar la posibilidad de que la fuente sea una sopa de murciélago que se consume habitualmente en la zona, ya que estos animales podrían actuar como reservorio del virus. Un informe prematuro sobre un estudio de investigadores chinos, postulaba a los pangolines salvajes como posible intermediario del SARS-CoV-2, cuya captura y venta es ilegal en China, aunque se trafica con ellos de forma clandestina, pero los resultados finales concluyeron que los metagenomas del virus encontrado en estos y los del ser humano eran similares solo en un 90 por ciento y no tenía el motivo RRAR, una inserción peptídica única en el virus SARS-CoV-2 humano posiblemente involucrado en la escisión proteolítica de la spike y el rango de hospedadores y la transmisibilidad, lo que sugiere que el virus SARS-CoV-2 humano no provino directamente de los pangolines.[49][50][51]
Mecanismo de transmisión
La transmisión del virus entre humanos es posible a través de las secreciones respiratorias de las personas infectadas, sobre todo mediante la expulsión, a través de la tos o el estornudo, de pequeñas gotas y aerosoles que pueden cruzar el aire,[39][52][53][54] también mediante contacto directo con estas secreciones o por objetos contaminados por las mismas o fómites.[55]
Patogenia en el ser humano
Ciclo infectivo
El ciclo infectivo del virus SARS-CoV-2 comienza con la interacción entre la proteína "Spike1" (S1) del patógeno con la proteína de la membrana celular ACE2 o enzima convertidora de angiotensina 2.[56] Antes de continuar cabe destacar que esta proteína también es la puerta de entrada para el virus SARS-CoV, aunque su afinidad por esta enzima es menor que la del SARS-CoV-2.[57]
Posteriormente, la proteasa TMPRSS2 realiza el corte de la proteína S1, lo cual desencadena la fusión del virus con la membrana celular o su endocitosis. Una vez dentro, el virus es capaz de secretar su material genético o ARN en el interior de la célula. La propia maquinaria de la célula permitirá la traducción del genoma viral utilizando los ribosomas. La célula sintetizará proteínas virales y la nucleocápside del virus, para poder formar y ensamblar nuevos virus. Estos virus podrán salir de la célula mediante exocitosis e infectar a un mayor número de células.[58]
Periodo de incubación

El período de incubación, es decir el tiempo que transcurre desde que una persona se infecta por el virus hasta que presenta síntomas, oscila en general entre los 4 y los 7 días, en el 95 % de las ocasiones es menor a 12.5 días. Los límites extremos se han establecido entre 2 y 14 días después del contagio, aunque se han reportado casos inusuales de hasta 24 días.[20][39]
Clínica

Los síntomas iniciales de la infección pueden consistir en fiebre, tos, estornudos, dolor de garganta y manifestaciones generales como dolor articular, por lo que el cuadro sería similar al de la gripe. En algunas ocasiones se producen complicaciones como neumonía[59] y dificultad respiratoria que puede conducir a la muerte. A veces la única manifestación de la infección es el delirium (alteración aguda/fluctuante de la conciencia con fallos de la atención, la comprensión y el ciclo sueño-vigilia),[60] que si es grave puede indicar riesgo de muerte en pacientes críticamente enfermos.[61] Son más propensos a presentar complicaciones graves los varones de más de 60 años, sobre todo los que presentan enfermedades previas.[20] La fiebre no aparece en todos los pacientes; en ocasiones no existe fiebre en pacientes muy jóvenes, ancianos, personas con la respuesta inmune disminuida o personas que toman ciertos medicamentos.[62] En los pacientes con COVID-19, la circunferencia del cuello se ha objetivado como un factor predictivo de necesidad de ventilación mecánica invasiva.[63]
Inmunidad

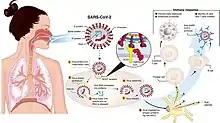
La respuesta inmunitaria natural de los seres humanos al virus SARS-CoV-2 se produce como una combinación de la inmunidad mediada por células y la producción de anticuerpos,[64] igual que con casi todas las otras infecciones.[65] La presencia de anticuerpos neutralizantes en la sangre se considera una prueba de inmunidad contra la infección, pero el nivel de anticuerpos neutralizantes disminuye con el tiempo, llegando a desaparecer tras tres meses en el 1 % de los pacientes y tras seis meses en el 12 %.[66] Sin embargo, la ausencia de anticuerpos en la sangre no significa que el sistema inmunitario no pueda producir nuevos anticuerpos rápidamente en caso de reexposición al SARS-CoV-2. Las células B de memoria específicas para las proteínas de la nucleocápside y el pico del SARS-CoV-2 duran al menos 6 meses después de la aparición de los síntomas.
Dado que el SARS-CoV-2 ha estado en la población humana solo desde diciembre de 2019, al principio de la pandemia se desconocía si la inmunidad era duradera en las personas que se recuperan de la enfermedad. Se reportaron casos de reinfección en pacientes que ya habían pasado la enfermedad y se temía que el SARS-CoV-2 se comportase como algunos otros coronavirus, que son capaces de reinfectar después de aproximadamente un año.[67]
En un estudio de enero de 2021 se encontró que la mayoría de los pacientes infectados por el virus SARS-CoV-2 quedan inmunizados durante al menos cinco meses, teniendo una probabilidad mucho más baja (el 83 %) de infectarse de nuevo que aquellos que no habían estado expuestos previamente al virus. Además, en caso de reinfección los previamente infectados solían no presentar síntomas (78 % de los casos) mientras que la ausencia de síntomas solo se dio en un 34 % de los no infectados previamente.[68][69] En un estudio de marzo de 2021 se observó que solo un 0,65 % de los pacientes infectados por SARS-CoV-2 volvieron a dar positivo en PCR al menos tres meses más tarde y que ninguno se infectó una tercera vez. Se estima que haber sido infectado reduce, de media, un 80 % la probabilidad de infectarse una segunda vez y que la protección dura al menos seis meses.[70][71] En otro estudio publicado en mayo de 2021, se concluyó que la inmunidad natural contra el SARS-CoV-2 dura años, probablemente toda la vida, incluso en personas que tuvieron síntomas leves de covid o no tuvieron cuando fueron infectados.[72][73][74] Ello se debe a la acción tanto de las células B como de las células plasmáticas de médula ósea de larga vida.
En total, se estima que haber contraído el virus proporciona una inmunización natural contra los síntomas de COVID-19 del 94 %, la cual es comparable a la de las mejores vacunas. No obstante, al igual que las vacunas, haber pasado el virus no garantiza que una persona no pueda volver a contraerlo y a contagiarlo, por lo cual las autoridades sanitarias recomiendan que todos los previamente infectados sigan aplicando los métodos de prevención habituales.[75]

La inmunidad innata frente al SARS-CoV-2 se utilizan principalmente por macrófagos y Células NK,en la inmunidad celular se utiliza por células dendriticas,linfocitos T CD4+ y linfocito T CD8+, y también en la inmunidad humoral la primera línea de defensa se utiliza la IgM y después la IgG, las citocinas implicadas en la inmunidad es el balance entre proinflamatorio y antiinflamatorio (IL-1β, IL-1RA, IL-2RA, IL-6, IL-7, IL-8, IL-9, IL-10, FGF básico, G-CSF, GM-CSF, HGF, Interferón gamma, IP-10, MCP-1, MIP-1a, MIP-1b, PDGF, TNF-α, VEGF y TGF-beta), la duración es alrededor de 2 semanas.[76][77][74][78]
Predisposición genética
Recientes estudios indican que puede existir una predisposición genética a ser infectados por el virus SARS-CoV-2. Concretamente, variantes genéticas en el gen de ACE2 en el cromosoma X han sido identificadas como posibles protectores de la infección. Estos polimorfismos darían lugar a niveles reducidos de la expresión de ACE2, reduciendo a su vez los puntos de entrada del virus en la célula. Se ha demostrado que estas variantes pueden suponer hasta un 37 % en la reducción de expresión de la proteína, evitando a su vez un gran número de infectados y muertes.[79]
Por otra parte, se han identificado polimorfismos genéticos en genes como los que codifican para IFNAR2, CXCR6 o el complejo mayor de histocompatibilidad (MHC) estar relacionados con la infección y síntomas de los pacientes de la enfermedad COVID-19. Estos genes están relacionados con el sistema inmune, lo cual nos podría dar una idea de la importancia de los mismos.[80][79] Otros genes como el del transportador de solutos SLC6A20 han demostrado no solo una relación con el gen ACE2 (importante para la entrada del virus en las células), sino que también una estrecha concordancia con los peores síntomas provocados por el SARS-CoV-2.[81]
Contención del virus
El 22 de enero de 2020, el gobierno chino declaró la cuarentena en las ciudades de Wuhan, Huanggang, Ezhou y otras, en un intento de contener la diseminación del brote viral.[82][83][84] Las autoridades chinas suspendieron viajes en avión, tren, autobuses y transbordadores dentro y fuera de Wuhan; para ayudar a limitar la propagación del virus, las autoridades sanitarias de Wuhan han hecho obligatorio el uso de mascarillas en lugares públicos.[82]
Medidas preventivas
Como medidas preventivas, se ha recomendado:[85]
- Lavarse frecuentemente las manos con agua y jabón, o si no con un desinfectante de manos a base de alcohol. La Organización Mundial de la Salud sugirió soluciones antisépticas[86] para manos en las que contienen el etanol o isopropanol, peróxido de hidrógeno y glicerol.
- Al toser o estornudar, cubrirse la boca y la nariz (dejando salir el aire adecuadamente, para así no aumentar la presión al interior de las vías respiratorias):
- con un pañuelo; desechar o lavar inmediatamente para evitar contaminar más superficies.
- con la fosa del codo.
- Mantener «al menos dos metros (6 pies) de distancia» respecto a otras personas, «particularmente aquellas que tosan, estornuden y tengan fiebre». Usar cubrebocas y mascarillas, ya sea quirúrgicas o de tela, uno sobre el otro.
- Evitar tocarse los ojos, la nariz y la boca.
- Permanecer en casa si empieza a encontrarse mal, si se trata de síntomas leves como cefalea o rinorrea leve hasta que se recupere, si se encuentra en zonas donde se está propagando el virus, o si las ha visitado en los últimos 14 días.[87]
- Consultar con el médico en caso de fiebre, tos y dificultad para respirar, llamando con antelación si se encuentra en zonas donde se está propagando el virus, o si las ha visitado en los últimos 14 días para que se tomen medidas para evitar que otros pacientes se contagien.
- Evitar el contacto sin protección con animales de granja o salvajes.[88]
- Evitar el consumo de alimentos poco cocinados o crudos, provenientes de animales.[89]
El 7 de julio de 2020, la OMS dijo en una conferencia de prensa que emitirá nuevas directrices sobre la transmisión en entornos con contacto cercano y falta de ventilación.[52]
Vacunas y antivirales
Para febrero de 2021, diez vacunas han sido autorizadas para su uso público por al menos una autoridad reguladora competente. Además, hay unas 70 vacunas candidatas en investigación clínica, de las cuales 17 en ensayos de fase I, 23 en ensayos de fase I-II, 6 en ensayos de fase II y 20 en ensayos de fase III. Las vacunas contra la COVID-19, se pueden clasificar según el vector que utilizan para introducir el material del SARS-CoV-2. El vector puede ser una versión inactivada del propio coronavirus, otro virus (generalmente un adenovirus) al que se le ha insertado ARN del SARS-CoV-2, o bien ARN mensajero solo.
La vacunas que se encuentran en uso en la actualidad son las:
- Vacunas de ARN mensajero: el tozinamerán de Pfizer-BioNTech y mRNA-1273 de Moderna.
- Vacunas de coronavirus inactivado: BBIBP-CorV de Sinopharm, BBV152 de Bharat Biotech, CoronaVac de Sinovac y WIBP-CorV de Sinopharm.
- Vacunas de otros vectores virales: Sputnik V del Instituto Gamaleya, AZD1222 de Oxford-AstraZeneca, Ad5-nCoV de CanSino Biologics y Ad26.COV2.S de Janssen-J&J.
- Vacuna de antígenos peptídicos: EpiVacCorona del Instituto Vector.
La eficacia más alta contra los síntomas obtenida hasta ahora por una vacuna contra la COVID-19 es del 95 %, un valor similar a la inmunidad natural que se obtiene al infectarse con el SARS-CoV-2. Otras vacunas, sin embargo, presentan una eficacia menor, algunas de solo el 50 %. Otra diferencia importante entre las diferentes vacunas es su temperatura de conservación. Mientras que las vacunas de adenovirus o coronavirus inactivados se conservan en refrigeradores, las de ARN mensajero requieren congeladores a -20 °C (Moderna) o incluso a -80 °C (Pfizer), lo cual complica su distribución.Debido a la capacidad de producción limitada de los fabricantes de vacunas, los estados han tenido que implementar planes de distribución por etapas, que dan prioridad a la población de riesgo, como los ancianos, y a las personas con alto grado de exposición y transmisión, como los trabajadores sanitarios. Para el 1 de febrero de 2021, se habían administrado 101,3 millones de dosis de vacunas contra la COVID-19 en todo el mundo, según informes oficiales de las agencias nacionales de salud.
A fecha de diciembre de 2020, los estados habían comprado por adelantado más de 10 mil millones de dosis de vacunas; de ellas, aproximadamente la mitad habían sido adquiridas por países de ingresos altos que representaban el 14 % de la población mundial. En el consejo de los ADPIC, el acuerdo que regula la propiedad intelectual y patentes dentro de la OMC, la India y Sudáfrica presentaron en octubre de 2020 una propuesta para la suspensión temporal —mientras dure la pandemia— de los medicamentos, vacunas e instrumentación médica de uso en el tratamiento de la COVID-19. A este propuesta se opusieron principalmente los países ricos, entre ellos la Unión Europea, los Estados Unidos, el Reino Unido y Brasil. En el mes de mayo de 2021, otros 60 países han copatrocinado la propuesta, que alcanza el apoyo de más de 100 países, y el 5 de mayo los Estados Unidos dio la sorpresa al anunciar que apoyaba la propuesta de suspensión de patentes, si bien sólo en relación con la vacunas.[90]
Variantes
Las variantes del SARS-CoV-2 corresponden a la presencia de alteraciones puntuales a nivel de diferentes proteínas del virus. A la fecha de finales de abril de 2022 se han descrito 20 variantes con importancia epidemiológica:
Véase también
- Coronavirus del síndrome respiratorio agudo grave (SARS) (genéticamente similar)
- Coronavirus del síndrome respiratorio de Oriente Medio (MERS) (genéticamente similar)
- Crisis sanitaria
- Anexo:Cronología de las pandemias
Referencias
- «Taxonomy browser (Betacoronavirus)». Centro Nacional para la Información Biotecnológica, Biblioteca Nacional de Medicina de Estados Unidos. Archivado desde el original el 20 de marzo de 2020. Consultado el 14 de marzo de 2020.
- «Los nombres de la enfermedad por coronavirus (COVID-19) y del virus que la causa». who.int. Organización Mundial de la Salud. Consultado el 3 de noviembre de 2020.
- «Enfermedad del coronavirus 2019 (COVID-19). urlarchivo=https://web.archive.org/web/20200318025404/https://www.cdc.gov/coronavirus/2019-ncov/faq-sp.html».
- «Coronavirus, claves de escritura». Fundéu BBVA. 29 de enero de 2020. Archivado desde el original el 30 de enero de 2020. Consultado el 25 de febrero de 2020.
- Organización Mundial de la Salud, ed. (11 de febrero de 2020). «WHO Director-General's remarks at the media briefing on 2019-nCoV on 11 February 2020». www.who.int (en inglés). Archivado desde el original el 12 de febrero de 2020. Consultado el 11 de febrero de 2020.
- Gorbalenya, A.E.; Baker, S.C.; Baric, R.S.; de Groot, R.J.; Drosten, C.; Gulyaeva, A.A.; Haagmans, B.L.; Lauber, C.; Leontovich, A.M.; Neuman, B.M.; Penzar, D.; Poon, L. L. M.; Samborskiy, D.; Sidorov, I. A.; Sola, I.; Ziebuhr, J. «Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group». bioRxiv (en inglés). doi:10.1101/2020.02.07.937862. Archivado desde el original el 11 de febrero de 2020. Consultado el 11 de febrero de 2020.
- Jiang, Shibo; Shi, Zhengli; Shu, Yuelong; Song, Jingdong; Gao, George F; Tan, Wenjie; Guo, Deyin (de marzo de 2020). «A distinct name is needed for the new coronavirus». The Lancet (en inglés) 395 (10228): 949. PMC 7124603. PMID 32087125. doi:10.1016/S0140-6736(20)30419-0. Consultado el 19 de septiembre de 2020.
- Wong, Gary; Bi, Yu-Hai; Wang, Qi-Hui; Chen, Xin-Wen; Zhang, Zhi-Gang; Yao, Yong-Gang (18 de mayo de 2020). «Zoonotic origins of human coronavirus 2019 (HCoV-19 / SARS-CoV-2): why is this work important?». Zoological Research (en inglés) 41 (3): 213-219. PMC 7231470. PMID 32314559. doi:10.24272/j.issn.2095-8137.2020.031. Consultado el 19 de septiembre de 2020.
- Zhou, Peng; Yang, Xing-Lou; Wang, Xian-Guang; Hu, Ben; Zhang, Lei; Zhang, Wei; Si, Hao-Rui; Zhu, Yan; Li, Bei; Huang, Chao-Lin; Chen, Hui-Dong; Chen, Jing; Luo, Yun; Guo, Hua; Jiang, Ren-Di; Liu, Mei-Qin; Chen, Ying; Shen, Xu-Rui; Wang, Xi; Zheng, Xiao-Shuang; Zhao, Kai; Chen, Quan-Jiao; Deng, Fei; Liu, Lin-Lin; Yan, Bing; Zhan, Fa-Xian; Wang, Yan-Yi; Xiao, Gengfu; Shi, Zheng-Li (23 de enero de 2020). «Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin». bioRxiv (en inglés): 2020.01.22.914952. doi:10.1101/2020.01.22.914952. Archivado desde el original el 24 de enero de 2020. Consultado el 5 de febrero de 2020.
- Rehman, Saif Ur; Shafique, Laiba; Ihsan, Awais; Liu, Qingyou (23 de marzo de 2020). «Evolutionary Trajectory for the Emergence of Novel Coronavirus SARS-CoV-2». Pathogens (Basel, Switzerland) 9 (3). ISSN 2076-0817. PMC 7157669. PMID 32210130. doi:10.3390/pathogens9030240. Consultado el 25 de abril de 2021.
- «Coronavirus y síndrome respiratorio agudo severo».
- «A new coronavirus associated with human respiratory disease in China».
- «Novel coronavirus (2019-nCoV), Wuhan, China». Centers for Disease Control and Prevention. 10 de enero de 2020. Archivado desde el original el 11 de enero de 2020. Consultado el 16 de enero de 2020.
- Zhang, Y.-Z., et al. (12 de enero de 2020). Centro Nacional para la Información Biotecnológica, EE. UU., ed. Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome (en inglés). Bethesda MD. p. GenBank. Archivado desde el original el 13 de enero de 2020. Consultado el 13 de enero de 2020.
- «中国疾病预防控制中心». www.chinacdc.cn. Archivado desde el original el 6 de febrero de 2020. Consultado el 9 de enero de 2020.
- Xinhua / huaxia |, ed. (9 de enero de 2020). «New-type coronavirus causes pneumonia in Wuhan: expert». www.xinhuanet.com. Archivado desde el original el 9 de enero de 2020. Consultado el 9 de enero de 2020.
- «CoV2020». platform.gisaid.org. Consultado el 12 de enero de 2020 (sin acceso público).
- European Centre for Disease Prevention and Control (ed.). «Q & A on novel coronavirus» (en inglés). Archivado desde el original el 4 de febrero de 2020. Consultado el 11 de febrero de 2020.
- Parker-Pope, Tara (21 abr. 2020). «¿El virus está en mi ropa? ¿En mis zapatos? ¿En mi pelo? ¿En mi periódico?». New York Times. Consultado el Domingo, 24 de mayo del 20202 – via NYTimes.com.
- Prompetchara, Eakachai; Ketloy, Chutitorn; Palaga, Tanapat (2020). «Immune responses in COVID-19 and potential vaccines:Lessons learned from SARS and MERS epidemic». Asian Pacific Journal of Allergy and Immunology (en inglés) (38): 1-9. doi:10.12932/AP-200220-0772.
- Nebehay, Stephanie. «WHO says new China coronavirus could spread, warns hospitals worldwide» (en inglés). Archivado desde el original el 14 de enero de 2020. Consultado el 19 de enero de 2020.
- «COVID-19, SARS CoV-2». The Sanford Guide to Antimicrobial Therapy (en inglés). Editorial Board. Consultado el 18 de agosto de 2020.
- «La OMS publica su primera validación para uso en emergencias de una vacuna contra la COVID-19 y hace hincapié en la necesidad de un acceso mundial equitativo». www.who.int. Consultado el 18 de mayo de 2021.
- «Vacunas contra la COVID-19». www.who.int. Consultado el 18 de mayo de 2021.
- Gómez-Carballa, Alberto; Bello, Xabier; Pardo-Seco, Jacobo; Martinón-Torres, Federico; Salas, Antonio (10 2020). «Mapping genome variation of SARS-CoV-2 worldwide highlights the impact of COVID-19 super-spreaders». Genome Research 30 (10): 1434-1448. ISSN 1549-5469. PMC 7605265. PMID 32878977. doi:10.1101/gr.266221.120. Consultado el 16 de diciembre de 2020.
- «Initial genome release of novel coronavirus». Virological (en inglés estadounidense). 11 de enero de 2020. Archivado desde el original el 12 de enero de 2020. Consultado el 12 de enero de 2020.
- Comité Nacional de Vigilancia Epidemiológica (CONAVE), ed. (21 de enero de 2020). «Aviso Epidemiológico». Archivado desde el original el 23 de enero de 2020. Consultado el 20 de marzo de 2020.
- «Wuhan virus: China reports sixth death in pneumonia outbreak, Asian markets spooked». The Straits Times (en inglés). 21 de enero de 2020. Archivado desde el original el 19 de febrero de 2020. Consultado el 19 de marzo de 2020.
- Chris Lau; Phila Siu; Kanis Leung; Chan Ho-him (23 de enero de 2020). «China coronavirus: Hong Kong scraps major Lunar New Year celebrations and extends health declaration requirements as it tightens monitoring of deadly infection». South China Morning Post. Archivado desde el original el 23 de enero de 2020. Consultado el 23 de enero de 2020.
- Michelle Fay Cortez; John Lauerman (21 de enero de 2020). Bloomberg L.P., ed. «New Virus Spreads to U.S., Sparking Rush to Contain Outbreak» (en inglés). Consultado el 20 de marzo de 2020.
- «Wuhan virus: Vietnam says two Chinese citizens in Vietnam confirmed to have coronavirus». The Straits Times (en inglés). 23 de enero de 2020. Archivado desde el original el 24 de enero de 2020. Consultado el 19 de marzo de 2020.
- «Singapore confirms first case of Wuhan virus; second case likely». The Straits Times (en inglés). 23 de enero de 2020. Archivado desde el original el 19 de marzo de 2020. Consultado el 19 de marzo de 2020.
- Organización Mundial de la Salud (ed.). «Declaración sobre la segunda reunión del Comité de Emergencias del Reglamento Sanitario Internacional (2005) acerca del brote del nuevo coronavirus (2019-nCoV)». Archivado desde el original el 20 de febrero de 2020. Consultado el 1 de febrero de 2020.
- Gobierno de México. Subsecretaría de Prevención y Promoción de la Salud (13 de febrero de 2020). «Comunicado Técnico Diario Nuevo Coronavirus en el Mundo (COVID-19)». (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última). enlace irrecuperable
- «WHO characterizes COVID-19 as a pandemic» (en inglés). canal de Youtube de la World Health Organization. 11 de marzo de 2020. Consultado el 14 de marzo de 2020.
- «Coronavirus confirmed as pandemic by World Health Organization» (en inglés). BBC News. 11 de marzo de 2020. Archivado desde el original el 11 de marzo de 2020. Consultado el 14 de marzo de 2020.
- «SARS-CoV-2 (Severe acute respiratory syndrome coronavirus 2) Sequences» (en inglés). GenBank NCBI. 7 de abril de 2020. Consultado el 8 de abril de 2020.
- Centro Nacional para la Información Biotecnológica, EE. UU. (ed.). «Severe acute respiratory syndrome coronavirus 2». Archivado desde el original el 21 de marzo de 2020. Consultado el 21 de marzo de 2020.
- Ministerio de Sanidad, España, ed. (10 de febrero de 2020). «Informe técnico. Nuevo coronavirus 2019-n-CoV». Archivado desde el original el 3 de marzo de 2020. Consultado el 14 de febrero de 2020.
- Lim, Yvonne Xinyi; Ng, Yan Ling; Tam, James P.; Liu, Ding Xiang (25 de julio de 2016). «Human Coronaviruses: A Review of Virus–Host Interactions». Diseases 4 (3): 26. ISSN 2079-9721. PMC 5456285. PMID 28933406. doi:10.3390/diseases4030026.
- Gómez-Carballa, Alberto; Bello, Xabier; Pardo-Seco, Jacobo; Martinón-Torres, Federico; Salas, Antonio (10 2020). «Mapping genome variation of SARS-CoV-2 worldwide highlights the impact of COVID-19 super-spreaders». Genome Research 30 (10): 1434-1448. ISSN 1549-5469. PMC 7605265. PMID 32878977. doi:10.1101/gr.266221.120. Consultado el 17 de diciembre de 2020.
- Korber, Bette; Fischer, Will M.; Gnanakaran, Sandrasegaram; Yoon, Hyejin; Theiler, James; Abfalterer, Werner; Hengartner, Nick; Giorgi, Elena E. et al. (2020-08). «Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus». Cell (en inglés) 182 (4): 812-827.e19. doi:10.1016/j.cell.2020.06.043. Consultado el 17 de diciembre de 2020.
- Chen, Nanshan; Zhou, Min; Dong, Xuan; Qu, Jieming; Gong, Fengyun; Han, Yang; et al. (30 de enero de 2020). «Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study». The Lancet (15 de febrero de 2020) 395 (10223): 507-513. doi:10.1016/S0140-6736(20)30211-7.
- Bedford, Trevor; Hodcroft, Emma. «Phylogeny of SARS-like betacoronaviruses including novel coronavirus SARS-CoV-2». https://nextstrain.org. Consultado el 18 de enero de 2020 (no archivable en el Internet Archive).
- Srinivasan, Suhas; Cui, Hongzhu; Gao, Ziyang; Liu, Ming; Lu, Senbao; Mkandawire, Winnie; Narykov, Oleksandr; Sun, Mo et al. (25 de marzo de 2020). «Structural Genomics of SARS-CoV-2 Indicates Evolutionary Conserved Functional Regions of Viral Proteins». Viruses 12 (4): 360. doi:10.3390/v12040360.
- «China confirms human-to-human transmission of new coronavirus» (en inglés). Associated Press (AP). 20 de enero de 2020 (no archivable en el Internet Archive).
- El coronavirus más mediático. Investigación y Ciencia, abril 2020, p. 12.
- Haitao Guo; Guangxiang Luo; Shou-Jiang Gao (24 de enero de 2020). CNN, ed. «Snakes could be the source of the Wuhan coronavirus outbreak» (en inglés). Archivado desde el original el 25 de enero de 2020. Consultado el 22 de marzo de 2020.
- China daily / Xinhua, ed. (7 de febrero de 2020). «Chinese study: Pangolins likely intermediate host of novel virus» (en inglés). Archivado desde el original el 20 de febrero de 2020. Consultado el 22 de marzo de 2020.
- «El pangolín, absuelto: no fue el origen del coronavirus». La Vanguardia. 27 de febrero de 2020. Consultado el 9 de abril de 2020.
- Deng, Junhua; Jin, Yipeng; Liu, Yuxiu; Sun, Jie; Hao, Liying; Bai, Jingjing; Huang, Tian; Lin, Degui et al. (2020). «Serological survey of SARS-CoV-2 for experimental, domestic, companion and wild animals excludes intermediate hosts of 35 different species of animals». Transboundary and Emerging Diseases (en inglés) 67 (4): 1745-1749. ISSN 1865-1682. PMC 7264586. PMID 32303108. doi:10.1111/tbed.13577. Consultado el 18 de agosto de 2020.
- Lewis, Dyani (8 de julio de 2020). «Mounting evidence suggests coronavirus is airborne — but health advice has not caught up». Nature (en inglés). doi:10.1038/d41586-020-02058-1. Consultado el 16 de julio de 2020.
- Lednicky, John A.; Lauzardo, Michael; Fan, Z. Hugh; Jutla, Antarpreet S.; Tilly, Trevor B.; Gangwar, Mayank; Usmani, Moiz; Shankar, Sripriya N. et al. (4 de agosto de 2020). «Viable SARS-CoV-2 in the air of a hospital room with COVID-19 patients». medRxiv (en inglés): 2020.08.03.20167395. doi:10.1101/2020.08.03.20167395. Consultado el 19 de agosto de 2020.
- Mandavilli, Apoorva (13 de agosto de 2020). «Unos científicos recolectaron coronavirus del aire de un hospital y descubrieron que puede ser infeccioso». The New York Times. ISSN 0362-4331. Consultado el 19 de agosto de 2020.
- Organización Mundial de la Salud (OMS), ed. (27 de febrero de 2020). «Getting your workplace ready for COVID-19». Archivado desde el original el 28 de febrero de 2020. Consultado el 21 de marzo de 2020.
- Medina-Enríquez, Miriam Marlene (22 de octubre de 2020). «ACE2: THE DOORWAY TO SARS-CoV-2». dx.doi.org. Consultado el 22 de enero de 2023.
- Ali, Amanat (14 de junio de 2020). «Dynamics of the ACE2 - SARS-CoV/SARS-CoV-2 spike protein interface reveal unique mechanisms». dx.doi.org. Consultado el 22 de enero de 2023.
- Shang, Jian; Wan, Yushun; Luo, Chuming; Ye, Gang; Geng, Qibin; Auerbach, Ashley; Li, Fang (6 de mayo de 2020). «Cell entry mechanisms of SARS-CoV-2». Proceedings of the National Academy of Sciences 117 (21): 11727-11734. ISSN 0027-8424. doi:10.1073/pnas.2003138117. Consultado el 22 de enero de 2023.
- Grant, Rogan A.; Morales-Nebreda, Luisa; Markov, Nikolay S.; Swaminathan, Suchitra; Querrey, Melissa; Guzman, Estefany R.; Abbott, Darryl A.; Donnelly, Helen K. et al. (2021-02). «Circuits between infected macrophages and T cells in SARS-CoV-2 pneumonia». Nature (en inglés) 590 (7847): 635-641. ISSN 1476-4687. doi:10.1038/s41586-020-03148-w. Consultado el 15 de abril de 2021.
- «Consenso del Comité de Psiquiatría de Enlace de la Asociación Colombiana de Psiquiatría sobre el diagnóstico y tratamiento del delirium en el contexto de la pandemia por COVID-19». Revista Colombiana de Psiquiatría (en inglés). 2 de diciembre de 2020. ISSN 0034-7450. PMC 7709601. doi:10.1016/j.rcp.2020.11.008. Consultado el 23 de febrero de 2021.
- Velásquez-Tirado, Juan D.; Trzepacz, Paula T.; Franco, José G. (12 de abril de 2021). «Etiologies of Delirium in Consecutive COVID-19 Inpatients and the Relationship Between Severity of Delirium and COVID-19 in a Prospective Study With Follow-Up». The Journal of Neuropsychiatry and Clinical Neurosciences (en inglés): appi.neuropsych.20100251. ISSN 0895-0172. doi:10.1176/appi.neuropsych.20100251. Consultado el 13 de abril de 2021.
- Departamento de Salud y Servicios para las Personas Mayores del Estado de Nueva Jersey, ed. (30 de enero de 2020). «Update and Interim Guidance on Infection Prevention and Control for 2019 Novel Coronavirus (2019‐nCoV)» (en inglés). p. 10. Archivado desde el original el 4 de febrero de 2020. Consultado el 21 de marzo de 2020.
- Di Bella, Stefano; Cesareo, Roberto; De Cristofaro, Paolo; Palermo, Andrea; Sanson, Gianfranco; Roman‐Pognuz, Erik; Zerbato, Verena; Manfrini, Silvia et al. (14 de junio de 2020). «Neck circumference as reliable predictor of mechanical ventilation support in adult inpatients with COVID‐19: A multicentric prospective evaluation». Diabetes/Metabolism Research and Reviews (en inglés). ISSN 1520-7552. PMC 7300447. PMID 32484298. doi:10.1002/dmrr.3354. Consultado el 11 de agosto de 2020.
- Immune responses and immunity to SARS-CoV-2Uso incorrecto de la plantilla enlace roto (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última)., by European Centre for Disease Prevention and Control
- Vabret N, Britton GJ, Gruber C, Hegde S, Kim J, Kuksin M, Levantovsky R, Malle L, Moreira A, Park MD, Pia L, Risson E, Saffern M, Salomé B, Esai Selvan M, Spindler MP, Tan J, van der Heide V, Gregory JK, Alexandropoulos K, Bhardwaj N, Brown BD, Greenbaum B, Gümüş ZH, Homann D, Horowitz A, Kamphorst AO, Curotto de Lafaille MA, Mehandru S, Merad M, Samstein RM (junio 2020). «Immunology of COVID-19: Current State of the Science». Immunity 52 (6): 910-941. PMC 7200337. PMID 32505227. doi:10.1016/j.immuni.2020.05.002.
- Gross, Anna (3 de febrero de 2021). «Antibody protection lasts at least six months, study finds». Financial Times. Consultado el 4 de febrero de 2021.
- «What if immunity to covid-19 doesn't last?». MIT Technology Review. Consultado el 1 de mayo de 2020.
- Hall, V.; Foulkes, S.; Charlett, A.; Atti, A.; Monk, E. J. M.; Simmons, R.; Wellington, E.; Cole, M. J. et al. (15 de enero de 2021). «Do antibody positive healthcare workers have lower SARS-CoV-2 infection rates than antibody negative healthcare workers? Large multi-centre prospective cohort study (the SIREN study), England: June to November 2020». medRxiv (en inglés): 2021.01.13.21249642. doi:10.1101/2021.01.13.21249642. Consultado el 21 de enero de 2021.
- «expert reaction to a preprint from the SIREN study looking at SARS-CoV-2 infection rates in antibody positive healthcare workers | Science Media Centre» (en inglés británico). Consultado el 21 de enero de 2021.
- Criado, Miguel Ángel (17 de marzo de 2021). «Menos del 1% de los afectados vuelve a contagiarse con el coronavirus». EL PAÍS. Consultado el 18 de marzo de 2021.
- Hansen, Christian Holm; Michlmayr, Daniela; Gubbels, Sophie Madeleine; Mølbak, Kåre; Ethelberg, Steen (2021-03). «Assessment of protection against reinfection with SARS-CoV-2 among 4 million PCR-tested individuals in Denmark in 2020: a population-level observational study». The Lancet (en inglés): S0140673621005754. PMC 7969130. doi:10.1016/S0140-6736(21)00575-4. Consultado el 18 de marzo de 2021.
- Domínguez, Nuño (2 de junio de 2021). «Los infectados son capaces de generar anticuerpos contra el coronavirus durante el resto de su vida». EL PAÍS. Consultado el 13 de junio de 2021.
- Turner, Jackson S.; Kim, Wooseob; Kalaidina, Elizaveta; Goss, Charles W.; Rauseo, Adriana M.; Schmitz, Aaron J.; Hansen, Lena; Haile, Alem et al. (24 de mayo de 2021). «SARS-CoV-2 infection induces long-lived bone marrow plasma cells in humans». Nature (en inglés): 1-8. ISSN 1476-4687. doi:10.1038/s41586-021-03647-4. Consultado el 2 de junio de 2021.
- https://www.eleconomista.es/sanidad/noticias/11236171/05/21/La-inmunidad-natural-frente-al-Covid19-podria-durar-para-toda-la-vida-.html La inmunidad natural frente al Covid-19 podría durar para toda la vida.Publicado el 26 de mayo de 2021.
- «Covid infection shown to provide as much immunity as vaccines». Financial Times. 14 de enero de 2021. Consultado el 21 de enero de 2021.
- https://elpais.com/elpais/2020/04/20/ciencia/1587379836_984471.html Así es la lucha entre el sistema inmune y el coronavirus. Autor:África González.Publicado el 21 de abril de 2020.
- https://www.scientificamerican.com/article/the-immune-havoc-of-covid-19/ The Immune Havoc of COVID-19.
- «Actualización sobre larespuesta inmunitaria a las infecciones por SARS-CoV-2 y otros virus». Consultado el 10 de febrero de 2021.
- Johnson, Norman (26 de junio de 2022). «Faculty Opinions recommendation of Genome-wide analysis provides genetic evidence that ACE2 influences COVID-19 risk and yields risk scores associated with severe disease.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado el 22 de enero de 2023.
- Dieter, Cristine; de Almeida Brondani, Leticia; Lemos, Natália Emerim; Schaeffer, Ariell Freires; Zanotto, Caroline; Ramos, Denise Taurino; Girardi, Eliandra; Pellenz, Felipe Mateus et al. (22 de diciembre de 2022). «Polymorphisms in ACE1, TMPRSS2, IFIH1, IFNAR2, and TYK2 Genes Are Associated with Worse Clinical Outcomes in COVID-19». Genes 14 (1): 29. ISSN 2073-4425. doi:10.3390/genes14010029. Consultado el 22 de enero de 2023.
- Kasela, Silva; Daniloski, Zharko; Bollepalli, Sailalitha; Jordan, Tristan X.; tenOever, Benjamin R.; Sanjana, Neville E.; Lappalainen, Tuuli (2021-12). «Integrative approach identifies SLC6A20 and CXCR6 as putative causal genes for the COVID-19 GWAS signal in the 3p21.31 locus». Genome Biology (en inglés) 22 (1): 242. ISSN 1474-760X. doi:10.1186/s13059-021-02454-4. Consultado el 22 de enero de 2023.
- British Broadcasting Corporation (BBC), ed. (23 de enero de 2020). «China coronavirus: Lockdown measures rise across Hubei province» (en inglés). Archivado desde el original el 15 de marzo de 2020. Consultado el 21 de marzo de 2020.
- Regan, Helen; Griffiths, James; Culver, David; Guy, Jack (24 de enero de 2020). Cable News Network (CNN), ed. «Wuhan coronavirus virus spreads as China scraps New Year celebrations» (en inglés). Archivado desde el original el 24 de enero de 2020. Consultado el 21 de marzo de 2020.
- «Wuhan virus: China locks down Huanggang, imposes tough travel restrictions in 3 other cities». Straits Times (en inglés). 3 de febrero de 2020. Archivado desde el original el 19 de marzo de 2020. Consultado el 21 de marzo de 2020.
- Organización Mundial de la Salud (OMS) (ed.). «Nuevo coronavirus (2019-nCoV): orientaciones para el público». www.who.int. Archivado desde el original el 14 de marzo de 2020. Consultado el 5 de febrero de 2020.
- Information, National Center for Biotechnology; Pike, U. S. National Library of Medicine 8600 Rockville; MD, Bethesda; Usa, 20894 (2009). WHO-recommended handrub formulations (en inglés). World Health Organization. Consultado el 28 de diciembre de 2020.
- Organización Mundial de la Salud (OMS) (ed.). «Brote de enfermedad por coronavirus (COVID-19): orientaciones para el público». Archivado desde el original el 16 de marzo de 2020. Consultado el 16 de marzo de 2020.
- Organización Mundial de la Salud (OMS), ed. (17 de enero de 2020). «Nuevo coronavirus». Archivado desde el original el 20 de febrero de 2020. Consultado el 25 de enero de 2020.
- Organización Mundial de la Salud (OMS), ed. (9 de marzo de 2020). «Q&A on coronaviruses (COVID-19). Can humans become infected with the COVID-19 from an animal source?» (en inglés). Archivado desde el original el 22 de marzo de 2020. Consultado el 22 de marzo de 2020.
- The Straits Times, ed. (23 de enero de 2020). «Wuhan virus: Work to start on three possible vaccines, says epidemic response group» (en inglés). Archivado desde el original el 24 de enero de 2020. Consultado el 26 de enero de 2020.
- Coalición para las Innovaciones en Preparación para Epidemias (CEPI), ed. (23 de enero de 2020). «CEPI to fund three programmes to develop vaccines against the novel coronavirus, nCoV-2019» (en inglés). Archivado desde el original el 24 de enero de 2020. Consultado el 22 de marzo de 2020.
- Sagonowsky, Eric (23 de enero de 2020). FiercePharma, ed. «Inovio, Moderna score CEPI funding for vaccine work against deadly coronavirus» (en inglés). Archivado desde el original el 26 de enero de 2020. Consultado el 22 de marzo de 2020.
- Gregorio-Nieto, Brenda; Hobbs, Miriam (25 de enero de 2020). National Broadcasting Company (NBC), ed. «Local Biotech Company Developing Coronavirus Vaccine» (en inglés). Archivado desde el original el 8 de marzo de 2020. Consultado el 22 de marzo de 2020.
- «Making Sense of Mutation: What D614G Meansfor the COVID-19 Pandemic Remains Unclear». CellPress. Consultado el 28 de agosto de 2020.
- «En Malasia, detectan cepa de Covid-19, 10 veces más infecciosa». Forbes. 17 de agosto de 2020. Consultado el 28 de agosto de 2020.
- «Omicron: everything you need to know about new Covid variant». the Guardian (en inglés). 26 de noviembre de 2021. Consultado el 26 de noviembre de 2021.
Enlaces externos
 Wikimedia Commons alberga una categoría multimedia sobre SARS-CoV-2.
Wikimedia Commons alberga una categoría multimedia sobre SARS-CoV-2.- Coronavirus COVID-19 Global Cases by the Center for Systems Science and Engineering (CSSE) at Johns Hopkins University (JHU) Seguimiento en tiempo real de la cantidad de casos de COVID-19 a través de mapa interactivo
- Genoma completo en GENBANK
- Genoma de SARS-CoV-2, vía UCSC Genome Browser
- «SARS-CoV-2 related protein structures». Base de datos de proteínas de SARS-CoV-2.
- Una historia de coronavirus