Viroma humano
El viroma humano es la colección total de virus en y sobre el cuerpo humano.[3][4][5] Los virus en el cuerpo humano pueden infectar tanto las células humanas como otros microbios, como las bacterias (bacteriófagos).[6] Algunos virus causan enfermedades, mientras que otros pueden ser asintomáticos. Ciertos virus también se integran en el genoma humano como provirus o elementos virales endógenos.
| Microbiota humana | ||
|---|---|---|
| Flora humana | ||
| Trastornos y terapias | ||
| Relacionado: | ||
Los virus evolucionan rápidamente y, por lo tanto, el viroma humano cambia constantemente.[7] Cada ser humano tiene un viroma único con un equilibrio único de especies.[4][8] El estilo de vida, la edad, la ubicación geográfica e incluso la estación del año pueden afectar la exposición de un individuo a los virus, y la susceptibilidad de uno a cualquier enfermedad que pueda ser causada por esos virus también se ve afectada por la inmunidad preexistente y la genética viral y humana.[9]
El viroma humano está lejos de estar completamente explorado y con frecuencia se descubren nuevos virus. A diferencia de los aproximadamente 40 billones de bacterias en un microbioma humano típico,[10] todavía no se dispone de una estimación del número de partículas virales en un humano adulto sano, aunque los viriones generalmente superan en número a las bacterias individuales 10:1 en la naturaleza. El estudio del viroma se cree que proporcionará una comprensión de los microbios en general y cómo afectan la salud y la enfermedad humana.
Métodos y herramientas
Hay varios métodos disponibles para el aislamiento y el estudio de virus humanos:
- La secuenciación profunda es una técnica de secuenciación rápida de ADN que es útil para caracterizar la riqueza del viroma, la estabilidad, la función del gen y la asociación con fenotipos de enfermedades.[3][5] Esta tecnología crea grandes cantidades de información de secuencia y es capaz de detectar componentes raros de una comunidad microbiana. Los métodos actuales que combinan la eliminación de ADN humano y bacteriano de las muestras, la secuenciación a gran escala y la bioinformática son muy eficientes en la identificación de virus desconocidos.[11] A diferencia de otros métodos de descubrimiento, los virus no necesitan crecer en cultivos celulares. Sin ningún conocimiento previo de la secuencia del genoma o los métodos de crecimiento, se pueden descubrir nuevos virus. Por lo tanto, la secuenciación profunda es adecuada para la identificación rápida de un virus desconocido o inesperado involucrado en un brote de enfermedad o asociado con afecciones que no se consideran causadas por virus. La secuenciación profunda también permite proyecciones a gran escala con un mínimo esfuerzo práctico.[12] Una exploración sistemática de los virus que infectan a los humanos (el viroma humano) es importante y factible con estos métodos.
- La reacción en cadena de la polimerasa es una herramienta para amplificar y detectar secuencias de ADN específicas. Se puede usar para ayudar a caracterizar el viroma, pero está limitado por la necesidad de información de secuencia de ADN al menos parcial.
- El metagenoma humano incluye todos los organismos que viven en nosotros. Los virus contribuyen al metagenoma y establecen una infección crónica que infesta los cromosomas; Este método formulará una nueva estimación del número de genes que confieren susceptibilidad a un virus determinado y especificará alelos para algunos virus.[13][14]
- Los estudios de anticuerpos a gran escala con ELISA usando sangre donada podrían ayudar a determinar la exposición humana a virus particulares en diferentes regiones geográficas.[9]
Diversidad de virus humanos
El viroma humano no es estable y puede cambiar con el tiempo. De hecho, nuevos virus se descubren constantemente.[9][16][17] Con un número cada vez mayor de virus conocidos, el diagnóstico y el tratamiento de nuevas afecciones asociadas a virus también serán más fáciles.[18][19] Estudiar el viroma podría ayudar a mejorar el desarrollo de fármacos y limitar el uso de antibióticos.[4][20]
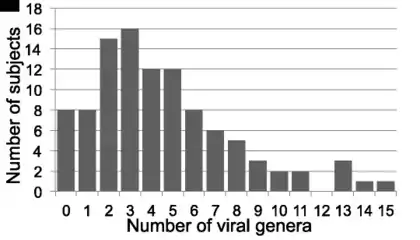
Uno de los primeros estudios que utilizó la secuenciación de ADN de alto rendimiento para describir la diversidad de los virus de ADNds eucariotas en individuos normales incluyó 706 muestras de 102 sujetos.[15] Este estudio detectó un promedio de 5.5 géneros virales en cada individuo y estos virus incluyeron herpesvirus, papilomavirus, poliomavirus, adenovirus, anellovirus, parvovirus y circovirus.

Cada individuo tenía un perfil viral distinto, lo que demuestra la alta diversidad interpersonal del viroma. Se detectaron de uno a 15 géneros virales (promedio 5.5) en el 92% de los 102 individuos muestreados (Figura 2).[15] La Figura 3 ilustra los viromas de los 102 individuos definidos mediante el muestreo de hasta cinco hábitats corporales principales, mostrando que se detectó una amplia gama de virus en personas sanas (Figura 3).
Los 102 individuos portaban siete familias distintas de virus de ADN humano (Figura 4A).[15] Las secuencias se detectaron predominantemente en la nariz y la piel, similitud con 17 géneros de virus del papiloma (Figura 4B). Los rosolovirus, predominantemente HHV-7 y, en menor medida, HHV-6, estuvieron presentes en el 98% de los individuos que proporcionaron muestras de boca.
Además, los mismos virus prevalecieron en múltiples hábitats corporales dentro de los individuos. Por ejemplo, los virus del papiloma beta y gamma fueron los virus más comúnmente encontrados en la piel y la nariz (Figura 4A, B), que pueden reflejar proximidad y similitudes en microambientes que apoyan la infección con estos virus.[15]

El viroma de la sangre humana
Los datos de secuenciación del genoma completo de sangre de 8,240 individuos sin ninguna enfermedad infecciosa clara revelaron 94 virus diferentes en el 42% de los participantes del estudio. Las secuencias incluyeron 19 virus de ADN humano, provirus y virus de ARN (herpesvirus, anellovirus, papilomavirus, tres poliomavirus, adenovirus, VIH, HTLV, hepatitis B, hepatitis C, parvovirus B19 y virus de la influenza).[21] De posible relevancia para la medicina de transfusiones, este estudio identificó el poliomavirus de células de Merkel en 49 individuos, el virus del papiloma en sangre de 13 individuos, el parvovirus B19 en 6 individuos y la presencia de herpesvirus 8 en 3 individuos.
Impacto en la salud humana
El viroma humano es parte de nuestros cuerpos y no siempre causará daño. Muchos virus latentes y asintomáticos están presentes en el cuerpo humano todo el tiempo. Los virus infectan todas las formas de vida; por lo tanto, las células bacterianas, vegetales y animales y el material en nuestro intestino también portan virus.[8] Cuando los virus causan daño al infectar las células del cuerpo, se puede desarrollar una enfermedad sintomática. Contrariamente a la creencia común, los virus dañinos pueden ser minoritarios en comparación con los virus benignos en el cuerpo humano. Es mucho más difícil identificar virus que identificar bacterias, por lo tanto, nuestra comprensión de los virus benignos en el cuerpo humano es muy rudimentaria.[4]
Los efectos sobre la salud de los virus en un individuo dependen del sistema inmune del individuo.[3][19]
La hipótesis de higiene
Investigaciones recientes han relacionado la idea emergente de la hipótesis de higiene con los virus. Esta hipótesis intenta explicar y justificar algunas de las altas incidencias de enfermedades como el asma[22] y el eccema[23] en el mundo occidental ante el uso excesivo de antibióticos y antivirales por parte de la sociedad occidental. Este uso excesivo altera potencialmente no solo las bacterias del intestino sino también los virus que han vivido en armonía con el cuerpo humano y que ahora juegan un papel en la regulación de la salud humana. Esta hipótesis generalmente se refiere a microorganismos, pero ahora se está extendiendo para incluir virus en el aire e infecciones virales comunes de la infancia que son cada vez menos comunes.[24]
Efecto de la localización de la piel
Diversos virus colonizan la piel humana y difieren según el sitio de la piel.[25] Estos viromas de la piel incluyen virus humanos (virus del papiloma humano) y bacteriófagos (virus bacterianos) que infectan bacterias cutáneas comensales como los estafilococos.[26] Las comunidades de virus difieren según los niveles de humedad y el grado de protección del ambiente externo.
Efecto de la dieta
Muchos estudios han demostrado que las bacterias y los virus en el intestino humano (el microbioma intestinal) pueden verse alterados por los cambios en la dieta.[27] Un estudio que se centró en virus bacterianos, llamados bacteriófagos, en el intestino encontró una relación significativa entre la dieta y el tipo de bacteriófagos presentes.[28] Esto se hizo comparando la distancia entre las comunidades intestinales de bacteriófagos en individuos antes y después de que comenzaran una dieta controlada. Los resultados fueron que la distancia entre las comunidades intestinales de bacteriófagos de individuos con la misma dieta fue significativamente menor al final de su tratamiento dietético que al principio, mientras que no hubo un aumento en la similitud de la comunidad para individuos con diferentes dietas a lo largo del tiempo.
Referencias
- «Human AND RNA Virus - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 15 de noviembre de 2016.
- «bacteriophage - Gene - NCBI». www.ncbi.nlm.nih.gov. Consultado el 15 de noviembre de 2016.
- Wylie, Kristine M.; Weinstock, George M.; Storch, Gregory A. (1 de octubre de 2012). «Emerging view of the human virome». Translational Research 160 (4): 283-290. PMC 3701101. PMID 22683423. doi:10.1016/j.trsl.2012.03.006.
- Williams, S. C. P. (6 de febrero de 2013). «The other microbiome». Proceedings of the National Academy of Sciences 110 (8): 2682-2684. Bibcode:2013PNAS..110.2682W. PMC 3581985. PMID 23388628. doi:10.1073/pnas.1300923110.
- Fontana, Judith M.; Alexander, Elizabeth; Salvatore, Mirella (2012). «Translational research in infectious disease: current paradigms and challenges ahead». Translational Research 159 (6): 430-453. PMC 3361696. PMID 22633095. doi:10.1016/j.trsl.2011.12.009.
- Pride, David T; Salzman, Julia; Haynes, Matthew; Rohwer, Forest; Davis-Long, Clara; White, Richard A; Loomer, Peter; Armitage, Gary C et al. (8 de diciembre de 2011). «Evidence of a robust resident bacteriophage population revealed through analysis of the human salivary virome». The ISME Journal 6 (5): 915-926. PMC 3329113. PMID 22158393. doi:10.1038/ismej.2011.169.
- Woolhouse, M.; Scott, F.; Hudson, Z.; Howey, R.; Chase-Topping, M. (10 de septiembre de 2012). «Human viruses: discovery and emergence». Philosophical Transactions of the Royal Society B: Biological Sciences 367 (1604): 2864-2871. PMC 3427559. PMID 22966141. doi:10.1098/rstb.2011.0354.
- Zimmer, Carl. «Your Inner Lions: Get to Know Your Virome». National Geographic. Consultado el 29 de abril de 2013.
- Delwart, Eric; Racaniello, Vincent (14 de febrero de 2013). «A Roadmap to the Human Virome». PLoS Pathogens 9 (2): e1003146. PMC 3573120. PMID 23457428. doi:10.1371/journal.ppat.1003146.
- Sender, R; Fuchs, S; Milo, R (January 2016). «Are We Really Vastly Outnumbered? Revisiting the Ratio of Bacterial to Host Cells in Humans». Cell 164 (3): 337-40. PMID 26824647. doi:10.1016/j.cell.2016.01.013.
- Thurber, Rebecca V; Haynes, Matthew; Breitbart, Mya; Wegley, Linda; Rohwer, Forest (2009). «Laboratory procedures to generate viral metagenomes». Nature Protocols 4 (4): 470-483. PMID 19300441. doi:10.1038/nprot.2009.10.
- Allander, T. (6 de septiembre de 2005). «From The Cover: Cloning of a human parvovirus by molecular screening of respiratory tract samples». Proceedings of the National Academy of Sciences 102 (36): 12891-12896. PMC 1200281. PMID 16118271. doi:10.1073/pnas.0504666102.
- Virgin, Herbert W.; Wherry, E. John; Ahmed, Rafi (1 de julio de 2009). «Redefining Chronic Viral Infection». Cell 138 (1): 30-50. PMID 19596234. doi:10.1016/j.cell.2009.06.036.
- Kristensen, David M.; Mushegian, Arcady R.; Dolja, Valerian V.; Koonin, Eugene V. (2010). «New dimensions of the virus world discovered through metagenomics». Trends in Microbiology 18 (1): 11-19. PMC 3293453. PMID 19942437. doi:10.1016/j.tim.2009.11.003.
- Wylie, Kristine M.; Mihindukulasuriya, Kathie A.; Zhou, Yanjiao; Sodergren, Erica; Storch, Gregory A.; Weinstock, George M. (1 de enero de 2014). «Metagenomic analysis of double-stranded DNA viruses in healthy adults». BMC Biology 12: 71. ISSN 1741-7007. PMC 4177058. PMID 25212266. doi:10.1186/s12915-014-0071-7.
- Anderson, Norman G.; Gerin, John L.; Anderson, N. Leigh (1 de julio de 2003). «Global Screening for Human Viral Pathogens». Emerging Infectious Diseases 9 (7): 768-773. PMC 3023425. PMID 12890315. doi:10.3201/eid0907.030004.
- Dalke, Kate. «The Human Virome». Genome News Network. Consultado el 2 de abril de 2013.
- «Pathogenic simian immunodeficiency virus infection is associated with expansion of the enteric virome.». Cell 151 (2): 253-66. 12 de octubre de 2012. PMC 3490196. PMID 23063120. doi:10.1016/j.cell.2012.09.024.
- Pennisi, E. (24 de marzo de 2011). «Going Viral: Exploring the Role Of Viruses in Our Bodies». Science 331 (6024): 1513. Bibcode:2011Sci...331.1513P. PMID 21436418. doi:10.1126/science.331.6024.1513.
- Moustafa, Ahmed; Xie, Chao; Kirkness, Ewen; Biggs, William; Wong, Emily; Turpaz, Yaron; Bloom, Kenneth; Delwart, Eric et al. (22 de marzo de 2017). «The blood DNA virome in 8,000 humans». PLOS Pathogens 13 (3): e1006292. ISSN 1553-7374. PMC 5378407. PMID 28328962. doi:10.1371/journal.ppat.1006292.
- Butler, Christopher C (September 2013). «Asthma prevalence and humoral immune response in Somali immigrants in the US: implications for the hygiene hypothesis.». Primary Care Respiratory Journal: Journal of the General Practice Airways Group 22 (3): 262-264. PMC 6442840. PMID 23959045. doi:10.4104/pcrj.2013.00081.
- Strachan, David P (10 de junio de 2014). «Siblings, Asthma, Rhinoconjunctivitis And Eczema: A Worldwide Perspective From The International Study Of Asthma And Allergies In Childhood.». Clinical and Experimental Allergy 45 (1): 126-136. PMC 4298795. PMID 24912652. doi:10.1111/cea.12349.
- Daley, D (October 2014). «The evolution of the hygiene hypothesis: the role of early-life exposures to viruses and microbes and their relationship to asthma and allergic diseases.». Current Opinion in Allergy and Clinical Immunology 14 (5): 390-396. PMID 25102107. doi:10.1097/ACI.0000000000000101.
- Hannigan, Geoffrey D.; Meisel, Jacquelyn S.; Tyldsley, Amanda S.; Zheng, Qi; Hodkinson, Brendan P.; SanMiguel, Adam J.; Minot, Samuel; Bushman, Frederic D. et al. (30 de octubre de 2015). «The Human Skin Double-Stranded DNA Virome: Topographical and Temporal Diversity, Genetic Enrichment, and Dynamic Associations with the Host Microbiome». mBio (en inglés) 6 (5): e01578-15. ISSN 2150-7511. PMC 4620475. PMID 26489866. doi:10.1128/mBio.01578-15.
- Grice, Elizabeth A.; Kong, Heidi H.; Conlan, Sean; Deming, Clayton B.; Davis, Joie; Young, Alice C.; Program, NISC Comparative Sequencing; Bouffard, Gerard G. et al. (29 de mayo de 2009). «Topographical and Temporal Diversity of the Human Skin Microbiome». Science (en inglés) 324 (5931): 1190-1192. Bibcode:2009Sci...324.1190G. ISSN 0036-8075. PMC 2805064. PMID 19478181. doi:10.1126/science.1171700.
- Focà, Alfredo; Liberto, Maria Carla; Quirino, Angela; Marascio, Nadia; Zicca, Emilia; Pavia, Grazia (1 de enero de 2015). «Gut Inflammation and Immunity: What Is the Role of the Human Gut Virome?». Mediators of Inflammation 2015: 326032. ISSN 0962-9351. PMC 4405218. PMID 25944980. doi:10.1155/2015/326032.
- Minot, S.; Sinha, R.; Chen, J.; Li, H.; Keilbaugh, S. A.; Wu, G. D.; Lewis, J. D.; Bushman, F. D. (31 de agosto de 2011). «The human gut virome: Inter-individual variation and dynamic response to diet». Genome Research 21 (10): 1616-1625. PMC 3202279. PMID 21880779. doi:10.1101/gr.122705.111.
