Anticorps
Un anticorps est une substance produite par le système immunitaire adaptatif dans un organisme vivant pour détecter et neutraliser les agents pathogènes de manière spécifique. Ce faisant, les anticorps se lient à l'agent pathogène et dirigent vers lui, dans le but de le détruire, les cellules immunitaires dites phagocytes (macrophages, polynucléaires neutrophiles) et/ou les agents du complément. L'organisme devient alors réfractaire à l’agent envahisseur : il s’immunise[1].
Pour l’article ayant un titre homophone, voir Anticor.
Le terme anticorps réfère à la fonction de lutte contre les agents étrangers à l'organisme qui sont eux appelés antigènes. Les antigènes et les anticorps, dont la combinaison est à la base de la réaction immunologique d’un organisme contre un agent extérieur, n’ont pas de définition en eux-mêmes, mais se définissent l’un par rapport à l’autre[2] :
- est un antigène toute substance contre laquelle le système immunitaire d’un individu produit des anticorps. Un antigène capable de provoquer une réponse immunitaire est dit immunogène. Toute substance étrangère ou tout microbe introduit dans l'organisme peut se comporter en antigène. Des substances de taille et de forme très variable, voire des substances chimiques synthétiques qui ne sont jamais trouvées dans la nature, peuvent provoquer la production d'anticorps ;
- est un anticorps une immunoglobuline sécrétée par les plasmocytes, stade final de différenciation des lymphocytes B. Les immunoglobulines sont un des constituants majeurs des protéines plasmatiques qui furent d'abord appelées γ-globulines suivant leur profil électrophorétique lors de la séparation des protéines du sang. On distingues 5 classes d'immunoglobulines dites isotypes : A, D, E, G et M.
Il arrive que l'organisme produise des anticorps contre des constituants de lui même, comme on peut l'observer dans les maladies auto-immunes. On parle alors d'auto-anticorps.
Structure générale des immunoglobulines
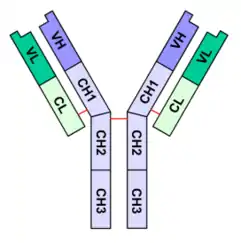
En bleu, Chaîne lourde avec une région variable (VH) suivie d'une région constante (CH1), une région charnière, et deux autres régions constantes (CH2 and CH3).
En vert, Chaîne légère avec une région variable (VL) et une constante (CL)
La structure générale des immunoglobulines a été décrite en 1959 par Porter à la suite des travaux d'Edelman. Ces deux chercheurs ont été associés pour le prix Nobel décerné en 1972. Les immunoglobulines sont des glycoprotéines formées de 4 chaînes polypeptidiques (150 000 uma ou dalton) : deux chaînes lourdes (H pour heavy de 50 000 uma chacune, en violet sur la figure 1), et deux chaînes légères (L pour light de 25 000 uma chacune, en vert) qui sont reliées entre elles par un nombre variable de ponts disulfures (en rouge) assurant une flexibilité de la molécule. Pour une molécule d'immunoglobuline donnée, les deux chaînes lourdes sont identiques, de même que les deux chaînes légères. Les immunoglobulines ont une forme en Y (chaque chaîne légère constitue pour moitié un bras du Y) et sont constituées de domaines immunoglobulines de 110 acides aminés environ. Chaque chaîne légère est constituée d'un domaine constant et d'un domaine variable ; les chaînes lourdes sont composées d'un domaine variable et de trois ou quatre domaines constants selon l'isotype. Les domaines de type immunoglobuline sont retrouvés dans de nombreuses protéines de fonctions très variables mais toujours impliquées dans des mécanismes d'interactions inter-cellulaires. Cela a amené à la définition d'une superfamille des immunoglobulines regroupant toutes ces protéines.

Domaines constants
Les domaines constants sont caractérisés par une séquence en acides aminés très proche d'un anticorps à l'autre, caractéristiques de l'espèce et de l'isotype. Chaque chaîne légère en possède un exemplaire noté CL. Les chaînes lourdes comportent, selon l'isotype, trois ou quatre domaines constants CH1, CH2, CH3, (CH4).
Les domaines constants ne sont pas impliqués dans la reconnaissance de l'antigène, mais interviennent dans l'activation du système du complément ainsi que dans l'élimination des complexes immuns (anticorps lié à son antigène) par les cellules immunitaires possédant les récepteurs aux fragments constants (RFc).
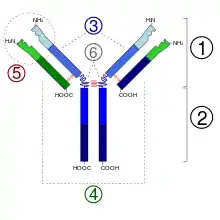
2. Fragment Fc
3. Chaîne lourde (en bleu) avec une région variable (VH) suivie d'une région constante (CH1), une région charnière, et deux autres régions constantes (CH2 and CH3).
4. Chaîne légère (en vert) avec une région variable (VL) et une constante (CL)
5. Paratope
6. Régions charnières.
Domaines variables
Un anticorps possède quatre domaines variables situés aux extrémités des deux « bras ». L'association entre un domaine variable porté par une chaîne lourde (VH) et le domaine variable adjacent porté par une chaîne légère (VL) constitue le site de reconnaissance (ou paratope) de l'antigène. Ainsi, une molécule d'immunoglobuline possède deux sites de liaison à l'antigène, soit un au bout de chaque bras. Ces deux sites sont identiques (mais destinés à différents épitopes), d'où la possibilité de lier deux molécules d'antigène par anticorps.
Fragments
Le clivage enzymatique spécifique des immunoglobulines permet d'isoler différents fragments :
- le fragment Fc (cristallisable). Il est le support des propriétés biologiques de l'immunoglobuline, en particulier sa capacité à être reconnu par des effecteurs de l'immunité ou à activer le complément. Il est constitué des fragments constants des chaînes lourdes (CH2) au-delà de la région charnière (« hinge », en anglais). Il ne reconnaît pas l'antigène ;
- le fragment Fv. C'est le plus petit fragment gardant les propriétés de l'anticorps que possède l'immunoglobuline. Il est constitué uniquement des régions variables VL et VH, il fixe donc l'antigène avec la même affinité que l'anticorps complet et est monovalent ;
- le fragment Fab. Il a la même affinité pour l'antigène que l'anticorps complet. Le fragment Fab est formé de la chaîne légère en entier (VL+CL) et d'une partie de la chaîne lourde (VH+CH1). Il est monovalent ;
- le fragment F(ab')2. Il correspond à l'association de deux fragments Fab reliés par une petite partie des parties constantes des chaînes lourdes, la région charnière (en anglais : hinge). Il a la même affinité que l'anticorps pour l'antigène et est divalent.
Isotypie, allotypie, idiotypie
C’est Jacques Oudin qui a forgé, à partir de 1956 les trois mots : isotypie, allotypie et idiotypie, qui sont maintenant utilisés par la communauté scientifique tout entière[6], et qui expriment l’infinie possibilité d’adaptation de notre système de défense immunitaire.
Isotypie
Les immunoglobuline sont subdivisées en classes ou « isotypes », selon la structure des domaines constants des chaînes lourdes : les chaînes γ, α, μ, ε et δ correspondent respectivement aux immunoglobulines IgG, IgA, IgM, IgE et IgD (voir Tableau 1). Il existe également des sous-classes d'immunoglobulines, reflétant des différences plus fines entre chaînes lourdes. L'être humain possède ainsi quatre sous-classes d'IgG et deux sous-classes d'IgA. Il existe également des isotypes de chaînes légères, celles-ci pouvant être κ (kappa) ou λ (lambda).
| IgG | IgA | IgM | IgE | IgD | |
| Localisation
membranaire |
Oui | Oui | Oui | Oui | Oui |
| Sécrétion | Oui | Oui
Monomère ou dimère |
Oui pentamère | Oui | Non |
| Valence1 | 2 | 2 à 4 | 2 à 10 | 2 | 2 |
| Localisation | sang | muqueuses sécrétions | Lymphocyte B sang | Granulocyte basophile mastocytes | Lymphocyte B |
| Proportion | 70 % à 75 % | 15 % à 20 % des anticorps sériques | 10 % | moins de 1 % | moins de 1 % |
| Rôles | neutralisation des toxines, bactéries et virus, voie classique du complément (sauf IgG4) | agglutination, neutralisation des bactéries, virus | agglutination, voie classique du complément | allergies, neutralisation de parasites | activation du lymphocyte B |
Tableau 1 : Propriétés des différents isotypes d'immunoglobulines.
- Les immunoglobulines M (IgM) sont présentes sous forme monomérique à la surface de la membrane des lymphocytes B naïfs. Ce sont les premières immunoglobulines rencontrées par un antigène lors de la première exposition de l'organisme à cet antigène. Elles peuvent ensuite être sécrétées par les plasmocytes et peuvent circuler alors associées en pentamères. Cependant cette valence de l'IgM de 10 n'est que théorique. En effet, bien que cet isotype d'anticorps soit de structure pentamérique, la gêne stérique occasionnée lors de la liaison aux épitopes des antigènes fait que la valence réelle est plus proche de 5, voire 6. Elles fixent le complexe C1 du complément et permettent son activation par la voie classique.
- Les immunoglobulines G (IgG) sont produites en réaction à un antigène lors de la réaction immunitaire secondaire. Elles sont générées à la suite de la commutation isotypique et révèlent un processus de maturation de la réponse anticorps de l'organisme. Elles protègent l'organisme contre les bactéries, les virus, et certaines toxines présentes dans le sang ou la lymphe. Elles fixent le complément (constituants du système immunitaire) et jouent un rôle dans la réponse mémoire, base de l'immunité durable, qui permet la vaccination. Elles traversent la barrière placentaire et permettent une « immunité passive » apportée par le système immunitaire de la mère au fœtus.
- Les immunoglobulines A (IgA) forment une barrière empêchant la plupart des pathogènes de se lier aux cellules des muqueuses et de l'épiderme. Elles sont présentes sous forme de dimères dans les sécrétions glandulaires et de type mucus, en particulier dans la salive, les larmes, le lait maternel (colostrum), les sécrétions nasales, les sécrétions gastrointestinales et celles du tractus respiratoire. Un déficit en IgA (qui peut avoir une origine génétique) peut conduire à des infections au niveau du tractus respiratoire supérieur (rarement au niveau inférieur), mais une élévation sérique des IgG et IgM peut pour partie compenser naturellement ce déficit[7],[8].
- Les immunoglobulines D (IgD) sont uniquement membranaire lymphocytes B où elles fixent les antigènes[8].
- Les immunoglobulines E (IgE) sont plus grosses que les immunoglobulines G. Produites par certains globules blancs (plasmocytes), dans la peau, le système digestif, les amygdales (ou tonsilles) et le tractus respiratoire. Elles sont reliées à deux types de globules blancs (les mastocytes et les granulocytes basophiles) par une sorte de tige. La capture d'un antigène, par cette immunoglobuline, entraîne la sécrétion des produits (dont de l'histamine) lançant une réaction inflammatoire et éventuellement allergique[8].
Allotypie
L’allotypie des protéines a été découverte, et nommée, en 1956 par Jacques Oudin. On pensait auparavant que tous les individus d’une même espèce animale avaient la même spécificité antigénique (il l’a alors appelé spécificité isotypique). Il a montré que les spécificités antigéniques (allotypiques) varient selon les groupes d’individus d’une même espèce et sont transmises héréditairement suivant les lois de Mendel[1].
C'est en 1956 que Grubb et Laurell ont découvert le système Gm, système de groupe des immunoglobulines IgG, par une technique d'inhibition d'antiglobuline. Les divers allotypes des chaînes lourdes constituent ce système. Il permet également de différencier les molécules des quatre sous-classes, IgG1, IgG2, IgG3, IgG4.
C. Ropartz et al. en 1961 ont découvert le système Km (à l'origine appelé Inv), porté par la chaîne légère Kappa, cet allotype est donc présent sur toutes les classes d'immunoglobulines.
Enfin le système ISF situé sur la chaîne lourde γ1 des IgG1, l'expression de cette spécificité augmentant avec l'âge, de 25 % des sujets avant 20 ans à 60 % au-delà de 70 ans, chez les caucasiens.
Les allotypes définis par le système Am sont situés sur les IgA, et plus précisément sur les chaînes α2. Il existe deux isotypes α1 et α2 de chaînes α, caractérisant les sous-classes Am1 et Am2 des IgA.
Idiotypie
L’idiotypie des anticorps a été découverte, et nommée, en 1963 par Jacques Oudin, en observant que les anticorps possèdent des spécificités antigéniques autres que les spécificités allotypiques et apparemment liées à la fonction anticorps. Il observa que certains immunsérums ne reconnaissaient que les anticorps ayant servi à leur préparation… Ainsi, les anticorps possédaient-ils des spécificités antigéniques apparemment liées à leur fonction de combinaison avec un antigène et à sa spécificité. Le système immunitaire de l’individu qui produit des anticorps est capable de réagir, dans des conditions non pathologiques, contre leurs spécificités idiotypiques et contribuer ainsi à la régulation de leur production[6],[9].
L'idiotype est un paratope propre à une molécule issue d'un seul clone. Cet épitope fait partie ou est très proche du site de reconnaissance de l'antigène, et est donc situé sur la partie variable, Fab (fragment antigen binding), de l'immunoglobuline. Autrement dit, le paratope ou sa région voisine d'une immunoglobuline peut être reconnu comme un épitope par certains lymphocytes. D'où la notion de réseau idiotypique.
Rôle des anticorps
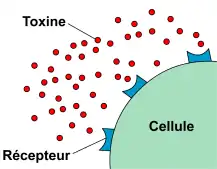
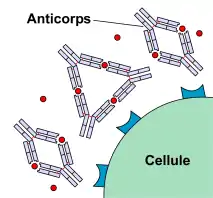
Au cours de la réponse immunitaire, les anticorps ont trois fonctions principales : se lier à l'antigène, activer le système du complément et recruter des cellules immunocompétentes.
Liaison à l'antigène
Les anticorps ont la capacité de reconnaître et de se fixer de manière spécifique sur un antigène. Cette spécificité est conférée par la présence de domaines extrêmement variables aux extrémités des anticorps. La reconnaissance entre antigène et anticorps est par exemple mise à profit dans la lutte contre les toxines bactériennes. Ces toxines agissent en se fixant sur des récepteurs présents à la surface des cellules de l'organisme, ce qui provoque des dérèglements importants de l'activité cellulaire. En se fixant sur ces toxines, les anticorps anti-toxine les neutralisent et empêchent les liaisons avec les récepteurs cellulaires (voir figure 2).
De la même manière, de nombreux virus et bactéries n'exercent leur pathogénicité qu'après fixation aux cellules de l'organisme. Les bactéries utilisent des adhésines qui sont des molécules d'adhésion aux membranes cellulaires et les virus possèdent des protéines de fixation sur leur enveloppe externe. Les anticorps anti-adhésines et anti-protéines de la capside virale bloquent l'action de ces agents pathogènes en se liant sur les molécules de fixation.
Activation du complément
Les anticorps protègent également l'organisme en déclenchant la cascade du complément. Il s'agit d'un ensemble de protéines du plasma dont l'activation (par la voie classique dans le cas d'anticorps) permet de détruire des bactéries par perforation et de faciliter la phagocytose, l'élimination des complexes immuns et la libération de molécules chimiotactiques. Ce qui amène à la lyse de l'élément pathogène. Ces protéines sériques sont des médiateurs de l'inflammation.
Activation de cellules immunocompétentes
Après avoir reconnu un antigène grâce à sa partie variable, un anticorps peut se lier à des cellules du système immunitaire par sa partie constante. Ces interactions revêtent une grande importance dans le déroulement de la réponse immunitaire. Ainsi, les anticorps fixés sur une bactérie peuvent se lier aux macrophages et déclencher une phagocytose. Les lymphocytes NK (Natural Killer) peuvent exercer leur cytotoxicité et lyser des bactéries opsonisées par des anticorps.
Synthèse des immunoglobulines
Généralités
Les gènes des immunoglobulines subissent une recombinaison V(D)J dans les lymphocytes B. Cette recombinaison est, en association avec les phénomènes d'hypermutation somatique et de variabilité jonctionnelle, la source de leur diversité. Cela conduit à l'apparition de clones de lymphocytes B, chaque clone exprimant une seule immunoglobuline spécifique d'un seul antigène. Dans le système lymphatique, les clones exprimant des immunoglobulines d'intérêt seront sélectionnés pour être conservés tandis que ceux exprimant des immunoglobulines reconnaissant des motifs protéiques appartenant au soi seront éliminés. C'est le principe de sélection clonale.
Synthèse des IgM
Les IgM sont les premières immunoglobulines produites par les lymphocytes B après la recombinaison V(D)J.
Synthèse des IgE, IgA et IgG
Lors de la maturation d'un lymphocyte B, et suivant les stimuli qui ont accompagné cette maturation, les clones de cellules B reconnaissant l'épitope subissent une commutation de classe. Les cellules B qui expriment des IgM et des IgD peuvent évoluer pour ne plus produire qu'un seul isotype (IgE, IgA et IgG), en opérant une recombinaison du gène codant le fragment constant (Fc) des chaînes lourdes, mais en gardant intact le fragment variable. Ce changement de fragment constant est également et plus souvent appelé commutation isotypique.
Ce phénomène est possible par le réarrangement des gènes codant les domaines CH : sur le génome, les segments de gène codant un isotype sont successifs et précédés d'une séquence de commutation. À la réception d'un signal extracellulaire de commutation, le lymphocyte B synthétise une recombinase qui forme une boucle non fonctionnelle entre les séquences de commutation : cette boucle rapproche un segment codant un domaine constant et l'association VDJ déjà formée.
Exemple : La production d'interleukine 4+++ aussi IL13 par le lymphocyte Th2 entraîne une commutation isotypique d'une IgM vers une IgE.
Anticorps monoclonaux et polyclonaux
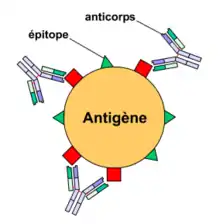
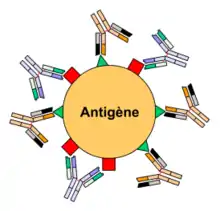
Un agent pathogène (bactérie, virus, etc.) est reconnu par le système immunitaire par l'intermédiaire d'antigènes. Un antigène possède généralement plusieurs épitopes différents qui sont autant de sites de liaison aux anticorps. On peut classer une population d'anticorps selon sa capacité à reconnaître un seul ou plusieurs épitopes. On parle alors respectivement d'anticorps monoclonaux et polyclonaux.
Anticorps monoclonaux
Les anticorps monoclonaux sont des anticorps ne reconnaissant qu'un seul type d'épitope sur un antigène donné (voir figure 3). Ils sont par définition tous identiques et produits par un seul clone de plasmocyte.
Les anticorps monoclonaux sont très largement utilisés en biologie et en médecine, à la fois comme outils de diagnostic et dans des buts thérapeutiques. Les anticorps monoclonaux utilisés comme médicaments ont tous une DCI se terminant par « mab », acronyme de monoclonal antibody par exemple le rituximab. Ils sont par exemple utilisés dans les tests de grossesse du commerce, ainsi que dans de nombreux domaines de la recherche en biologie et par de nombreuses techniques (cytométrie en flux, western blots…). Ils sont aussi de plus en plus utilisés dans les tests en laboratoire d'immuno-hématologie pour rehausser les réactions positives.
Produire des anticorps monoclonaux in vitro a longtemps été difficile, en raison de la faible durée de vie des cellules sécrétrices d'anticorps, les plasmocytes. Les anticorps étaient alors obtenus in vivo en injectant chez l'animal un antigène donné et en recueillant les anticorps dans son sang. Cette méthode coûteuse ne donnait qu'une faible quantité d'anticorps, pollués par de nombreuses impuretés.
À la fin des années 1970, César Milstein et Georges Köhler ont développé la technique des hybridomes. L'antigène est injecté chez l'animal, et des cellules de rate en sont prélevées après quelques semaines. Dans ces cellules se trouvent des plasmocytes sécrétant des anticorps dirigés spécifiquement contre l'antigène choisi. Ces plasmocytes sont alors fusionnés in vitro avec des myélomes, qui sont des cellules tumorales ayant la propriété de se multiplier indéfiniment. Les cellules hybrides obtenues (dites hybridomes) sont sélectionnées puis multipliées dans un milieu de culture approprié. Elles y produisent des anticorps monoclonaux, très purs et en quantités importantes.
Le génie génétique permet aujourd'hui de produire des anticorps monoclonaux utilisables en pratique clinique humaine. Mais la plupart des anticorps étant produits dans des cellules de rongeurs (souris, rat, hamster, lapin plus rarement poulet, mulet), ils déclenchent une réaction immunitaire lors de leur injection à un patient. Cette immunité inactive diminue progressivement l'action bénéfique de l'anticorps. Pour éviter cela, on cherche à produire des anticorps chimériques « humanisés », modifiés par génie génétique pour remplacer au maximum les fragments constants Fc de l'espèce d'origine par des fragments humains.
En cas de pandémie (de grippe aviaire) par exemple, l’immunothérapie passive des malades par des anticorps monoclonaux est une des solutions envisagées par les chercheurs qui en 2007 testaient déjà son efficacité sur l'animal[10], avec des résultats laissant penser que des anticorps monoclonaux d'origine humaine pourraient être produits à partir du sang de patients ayant guéri d'une grippe à H5N1 (ou de convalescents le cas échéants) et contribuer à enrayer une épidémie et à limiter le nombre de morts (en prophylaxie unique, ou comme traitement complémentaire).
Anticorps polyclonaux
Les anticorps polyclonaux sont un mélange d'anticorps reconnaissant différents épitopes sur un antigène donné, chaque idiotype étant sécrété par un clone de plasmocytes différent. Au cours de la réponse immunitaire, un organisme synthétise des anticorps dirigés contre plusieurs épitopes d'un antigène : la réponse est dite polyclonale. In vivo, la réponse est toujours polyclonale, sauf cas exceptionnels (vaccination par exemple). Cet exemple est en fait un exemple d'anticorps polyclonal monospécifique qui est en fait un anticorps qui reconnaît différents épitopes du même antigène. Un autre exemple concerne les anticorps anti-RH1. Une personne immunisée produit une multitude d'anticorps, donc polyclonaux, qui reconnaissent différents épitopes de la protéine RHD. Les anticorps monoclonaux utilisés au laboratoire ne reconnaissent chacun qu'un seul épitope de cette molécule. D'où le fait que certains variants de cette molécule peuvent être reconnus par un réactif — et donc étiquetés comme Rhésus Positif dans un laboratoire — et ne pas être reconnus par un second réactif — et donc étiquetés comme Rhésus Négatif dans un autre laboratoire.
Utilisations
Les anticorps comptent aujourd'hui « parmi les outils les plus couramment utilisés dans les sciences biologiques » et notamment dans le domaine biomédical, de la biologie moléculaire, de l'épigénétique, de la protéomique, mais aussi en microscopie à fluorescence, pour la sérologie ou encore comme abzyme. Le dosage sanguin des immunoglobulines spécifiques permet d'établir ou confirmer certains diagnostics médicaux.
Autrefois les chercheurs devaient produire eux-mêmes les anticorps qu'ils utilisaient, puis des entreprises les ont commercialisés[11] (vers 2010, environ 300 entreprises commercialisent plus de 2 millions d'anticorps, vendus aux chercheurs dans un marché qui en 2011 a atteint environ 1,6 milliard $, selon le consultant américain Frost & Sullivan[11]). Or, de nombreux travaux ont montré que beaucoup des anticorps commercialisés ne sont pas fiables (ex. : en 2011, une évaluation[12] de 246 anticorps utilisés en épigénétique a démontré qu'un quart de ces anticorps échouaient aux tests de spécificité (ce qui signifie qu'ils se lient à deux cibles ou plus)[12]. De plus, quatre de ces 246 anticorps se liaient très spécifiquement à une protéine, mais qui n'était pas la protéine-cible[12]).
Problème de standardisation et de mésusage
En octobre 2015, un sondage réalisé par le GBSI[13] montrait que 52 % des chercheurs n'arrivaient pas à authentifier l'identité de lignées cellulaires, qui peuvent facilement être contaminées avec des variétés indésirables de cellules à croissance rapide. C’est un problème qui peut induire de graves biais scientifiques d’interprétation. Une alerte était également donnée sur l'utilisation anarchique des anticorps[14]. Plusieurs études mettent en cause la reproductibilité de nombreuses expériences scientifiques[11]. De nombreux auteurs pensent que les anticorps sont l'un des moteurs importants de la « crise de reproductibilité » (ex. : les conclusions de 47 sur 53 travaux de recherche historiques sur le cancer basés sur l'utilisation de marquage par des anticorps n'ont pas pu être reproduites[11]. L'une des évaluations les plus larges a été faite par et pour l'Atlas des protéines humaines, un consortium suédois visant à produire des anticorps pour chaque protéine du génome humain. Il a étudié environ 20.000 anticorps commerciaux mis sur le marché et conclu que moins de 50 % pouvaient être utilisés efficacement pour cartographier la distribution des protéines dans des échantillons conservés de tissus ; souvent ils sont efficaces dans certaines conditions, mais sources d'erreurs catastrophiques dans d'autres)[11]. Selon des fabricants comme NeuroMab qui produit des anticorps pour les neurosciences, ses anticorps sont livrés avec une liste explicite des types d'expériences pouvant les utiliser, « mais les scientifiques ne suivent pas toujours ces instructions »[11]. Abgent (société de San Diego en Californie, vendant des anticorps) qui est aussi une filiale de WuXi AppTec basé à Shanghai a testé tous ses anticorps il y a environ un an[11]. Au vu des résultats, l'entreprise a retiré de son catalogue environ 1/3 de ses références pour monter en qualité devant ses clients, ce qui a permis de faire chuter le nombre de plaintes[11].
Un problème encore plus grave selon Leonard Freedman, président de la GBSI est qu’un nombre encore plus important de chercheurs ne valident pas les anticorps qu’ils utilisent de plus en plus souvent et massivement dans leurs expériences, or il est aussi essentiel que la recherche utilise des anticorps standardisés et ayant une cible précise et parfaitement connue dans les conditions où se fera l'expérimentation car des anticorps peu performants peuvent donner des faux positifs s’ils se lient à d’autres protéines que leur cible supposée. Ils peuvent aussi produire des « faux négatifs » quand ils ne parviennent pas à se lier à la protéine qu’ils sont supposés cibler.
Ces problèmes ont plusieurs fois conduit des scientifiques et des journaux à se rétracter pour des études déjà publiées. Ils ont aussi conduit des chercheurs à tirer des conclusions fausses ou contestables dans plusieurs études. Ceci explique en partie aussi le manque de reproductibilité constatée pour certaines études.
En 2016, un nouveau sondage[15] fait par le GBSI révèle que dans près d'un tiers des cas, les jeunes scientifiques ne prennent pas le temps de valider les anticorps qu’ils achètent aux fournisseurs commerciaux, même s'ils savent que la pertinence et l’exactitude de leurs résultats dépendent du bon fonctionnement et de l’adéquation de ces réactifs à leur travail. Deux raisons sont le manque de temps et une confiance excessive dans les produits du marché. Mais cette enquête révèle aussi que plus de 50 % ont répondu ne pas avoir reçu une formation spécifique sur la manière d'évaluer les anticorps.
« Ceci est très inquiétant », commente Matthias Uhlen (de l'Institut royal de technologie de Stockholm, qui dirige un groupe de travail international sur la validation d'anticorps, mais qui n'a pas été directement impliqué dans l'enquête publiée en 2016), notamment car il est important de vérifier par des essais l’adéquation d’un anticorps du commerce à la fonction qu’on veut lui attribuer, car par exemple certains anticorps détectent bien une protéine dans son « dénaturé » (dans les préparations cellulaires) mais pas dans sa forme naturelle qui est replié — ou inversement. De plus, un anticorps fonctionnant correctement pour un type de tissu ou dans une certaine préparation pourrait produire de faux signaux dans d'autres contextes[16].
Or valider un anticorps est plus compliqué qu'authentifier correctement une lignée cellulaire, et aussi parce que l’étude montre que les jeunes chercheurs (5 ans d’expérience ou moins) le font encore moins que leurs aînés : ils ne sont que 43 % à valider les anticorps qu’ils achètent dans le commerce (« la raison la plus souvent invoquée était le temps à passer pour le faire ») alors que 76 % des chercheurs ayant plus de 10 ans d’expérience disent le faire[16].
18 % des chercheurs en biologie médicale interrogés dans l'enquête de la GBSI ont reconnu qu'ils n’appliquaient aucune des procédures de validation requises, et 15 % des répondants n’étaient pas certains de le faire[16].
Selon Freedman, les problèmes de reproductibilité attribués aux anticorps peuvent être imputés à la fois à un déficit de formation et à l’usage d’anticorps inappropriés, deux causes encouragées par un manque de lignes directrices claires dans les bonnes pratiques. En outre, les entreprises qui vendent des anticorps ne donnent pas assez d’informations sur leurs performances en tant que réactif, et les auteurs d'articles ne citent pas toujours les numéros de catalogue ni les numéros de clone des anticorps qu'ils ont utilisé).
Pistes d'amélioration de la fiabilité des anticorps utilisés par la recherche
De multiples tentatives ont été faites pour améliorer à la fois la manière dont les chercheurs valident les anticorps[17],[18] et la manière dont ils rendent compte des anticorps. Les chercheurs qui utilisent des anticorps dans leurs travaux doivent les enregistrer correctement afin de permettre à leurs recherches d'être reproductibles (et donc testées et qualifiées par d'autres chercheurs). Moins de la moitié des anticorps de recherche référencés dans les articles universitaires peuvent être facilement identifiés[19]. Des articles publiés dans F1000 en 2014 et 2015 fournissent aux chercheurs un guide pour signaler l'utilisation d'anticorps de recherche[20],[21].
À partir de 2015, quelques groupes de chercheurs se sont organisés pour tenter de faciliter la circulation et validation des informations sur les anticorps du commerce[11] en créant les outils suivants :
- Antibodypedia, un portail collaboratif (en ligne) sur les réactifs : Antibodypedia (antibodypedia.com), maintenu en Suède par l'Atlas des protéines humaines, qui a catalogué plus de 1,8 million d'anticorps et évalué les données de validation disponibles pour diverses techniques expérimentales[11] ;
- anticorps en ligne (antibodies-online.com), un outil mis en place par et pour des laboratoires indépendants pour faire des études de validation, (généralement à la charge des vendeurs) ; sur 275 études, moins de la moitié des produits testés ont obtenu une « validation indépendante »[11] ;
- un Registre des anticorps (antibodyregistry.org), entretenu par une ONG qui attribue des identificateurs uniques à des anticorps, ce site propose aussi des liens vers d'autres ressources[11] ;
- pAbmAbs (pabmabs.com/wordpress), il s'agit d'une sorte de réseau social et plateforme de recommandations participatives, qui encourageant les scientifiques à examiner les anticorps qu'ils utilisent et à partager leurs informations[11] ;
- ImmPort anticorps Ontology Iniative Force11 (https://www.force11.org), projet visant à associer un identifiant unique à chaque anticorps (via Research ResourceIdentifiers ou RRIDs), utilisable dans les articles scientifiques et repérable par les moteurs de recherche[22]. L'article sur le RRID, est copublié dans 4 revues qui ont mis en œuvre la norme RRIDs pour la citation des ressources de recherche, qui tire des données de l'antibodyregistry.org comme source d'identifiants d'anticorps[23],[24].
Ces outils ne sont cependant pas très connus de la communauté scientifique[11].
Quelques revues scientifiques (dont le journal Nature) ont également commencé à demander à leurs auteurs de préciser si les anticorps utilisés dans leurs articles scientifiques ont été profilés pour l'application qu'ils en ont fait.
Un groupe de scientifiques a demandé en 2015 une révision radicale des procédés de fabrication et de vente des anticorps, et de diffusion de l'information associée. Ainsi, dans la revue Nature en 2015, Andrew Bradbury[25] (Los Alamos National Laboratory), avec plus de 100 cosignataires, demandait que l'on ne mette plus sur le marché que des anticorps définis jusqu'au niveau de la séquence d'ADN qui les produit, puis fabriqué dans des cellules génétiquement modifiées « recombinantes », de manière à obtenir que des produits strictement standardisés, et que l'on évite la variabilité introduite par la production du même anticorps chez les animaux d'élevage[25]. Cette proposition rendrait les anticorps 10 à 100 fois plus coûteux mais éviteraient des millions de dollars de pertes dues à des études ratées ou des résultats biaisés. Cependant, A. Bradbury réclame aussi des informations précises sur les anticorps individuels, or ces informations sont considérées par de nombreuses entreprises comme relevant du secret commercial. Il faudrait en outre une information complète sur les fonctions avérées de chacun de ces « anticorps recombinants »[11].
Une autre possibilité serait de les faire synthétiser par des virus[11].
En , ces questions seront à nouveau évoquées (et peut-être solutionnées) lors d'une réunion de 3 jours qui réunira des fournisseurs et utilisateurs d'anticorps ainsi que des bailleurs de fonds et des journaux scientifiques à Asilomar (Californie) en [26], lieu rendu célèbre pour la production de directives sur l'ADN recombinant[27].
Notes et références
- Martine Allain-Regnault, « La défense tous azimuts de l’organisme, deux hypothèses sur l’origine des anticorps », Le Monde, , p. 12 (lire en ligne)
- (en) Charles A. Janeway, Paul Travers, Mark Walport et J. Donald Capra, Immunobiology. The immune system in health and disease., Elsevier Science Ltd, , 635 p. (ISBN 0-8153-3217-3), p. 34-35
- (en) Eduardo Padlan, « Anatomy of the antibody molecule », Mol Immunol, vol. 31, no 3, , p. 169–217. (PMID 8114766, DOI 10.1016/0161-5890(94)90001-9)
- (en) « New Sculpture Portraying Human Antibody as Protective Angel Installed on Scripps Florida Campus » (consulté le )
- (en) « Protein sculpture inspired by Vitruvian Man » (consulté le )
- François Jacob, « La vie et l’œuvre de Jacques Oudin », La vie des sciences, Comptes rendus de l’Académie des sciences, 1987, série générale, tome 4, n°6,, p. 601-605
- (en) Calabrese E.J. - Pollutants and High-Risk Groups. New York, 1978
- Futura Science, les immunoglobulines
- (en) Simmons CP, Bernasconi NL, Suguitan Jr. AL, Mills K, Ward JM. et al., « Prophylactic and therapeutic efficacy of human monoclonal antibodies against H5N1 influenza », PLoS Med, vol. 4, no 5, , e178 (DOI 10.1371/journal.pmed.0040178)
- Monya Baker, « Reproducibility crisis: Blame it on the antibodies », Nature, vol. 521, (DOI 10.1038/521274a, lire en ligne [PDF])
- T.A. Egelhofer, A. Minoda, S. Klugman et al., « An assessment of histone-modification antibody quality », Nature Struct. Mol. Biol., vol. 18, , p. 91–93 (DOI 10.1038/nsmb.1972, lire en ligne [PDF])
- Global Biological Standards Institute (GBSI), une ONG basée à Washington DC, consacrée à l'amélioration de la recherche et des pratiques biomédicale – notamment en promouvant la reproductibilité des expériences et l’utilisation de réactifs plus fiables
- Monya Bake, « Antibody anarchy: A call to order », Nature, vol. 527, , p. 545–551 (DOI 10.1038/527545a, lire en ligne [PDF])
- Freedman L.P & al. BioTechniques 61, 16–18 (2016) ; sondage basé sur 400 chercheurs du domaine biomédical interrogés en ligne, présenté par le journal scientifique BioTechniques
- Monya Baker, « Biomedical researchers lax about validating antibodies for experiments », Nature, (DOI 10.1038/nature.2016.20192)
- (en) Saper CB, « An open letter to our readers on the use of antibodies », The Journal of Comparative Neurology, vol. 493, no 4, , p. 477–8 (PMID 16304632, DOI 10.1002/cne.20839, S2CID 14082678).
- (en) « NOT-OD-16-011: Implementing Rigor and Transparency in NIH & AHRQ Research Grant Applications », sur grants.nih.gov.
- (en) Vasilevsky NA, Brush MH, Paddock H, Ponting L, Tripathy SJ, Larocca GM et Haendel MA, « On the reproducibility of science: unique identification of research resources in the biomedical literature », PeerJ, vol. 1, , e148 (PMID 24032093, PMCID 3771067, DOI 10.7717/peerj.148).
- (en) Bandrowski A, Brush M, Grethe JS, Haendel MA, Kennedy DN, Hill S, Hof PR, Martone ME, Pols M, Tan S, Washington N, Zudilova-Seinstra E, Vasilevsky N, « The Resource Identification Initiative: A cultural shift in publishing », F1000Research, vol. 4, , p. 134 (PMID 26594330, PMCID 4648211, DOI 10.12688/f1000research.6555.2).
- (en) Helsby MA, Fenn JR, Chalmers AD, « Reporting research antibody use: how to increase experimental reproducibility », F1000Research, vol. 2, , p. 153 (PMID 24358895, PMCID 3829129, DOI 10.12688/f1000research.2-153.v2).
- https://scholar.google.com/scholar?q=RRID%3AAB_2298772)
- (en) « The Antibody Registry », sur antibodyregistry.org.
- (en) « Resource Identification Initiative », sur FORCE11, (consulté le ).
- A. Bradbury et A. Plückthun, « Reproducibility: Standardize antibodies used in research », Nature, vol. 518, , p. 27–29 (DOI 10.1038/518027a)
- Asilomar Conference Grounds 2016 : Antibody Validation: Standards, Policies, and Practices ; September 25-27 septembre 2016
- Paul Berg, « Meetings that changed the world: Asilomar 1975: DNA modification secured », Nature, vol. 455, , p. 290-291 (DOI 10.1038/455290a)
Voir aussi
Bibliographie
- (en) Jeremy Berg, John Tymoczko, Lubert Stryer, Biochemistry, W.H. Freeman and Company, New York, 2002. (ISBN 0-71674-684-0)
- Neil Campbell, Jane Reece, Biologie, De Boeck, 1995. (ISBN 2-80412-084-8)
- Charles Janeway, Paul Travers, Immunobiology, Garland Publishing, New York et Londres, 2001. (ISBN 0-81533-642-X)
Articles connexes
- Portail de la biologie
- Portail de la médecine