Chymotrypsine
La chymotrypsine est une peptidase digestive sécrétée par le pancréas, de la famille des protéases à sérine, qui catalyse des protéolyses. Elle est codée par deux gènes situés sur le chromosome 16, dont le produit est une proenzyme appelée chymotrypsinogène, qui doit être activé ultérieurement pour donner la chymotrypsine elle-même. Cette enzyme hydrolyse préférentiellement les protéines au niveau des liaisons peptidiques en aval d'un résidu de tyrosine, de tryptophane, de phénylalanine, de méthionine ou de leucine. Le site actif de la chymotrypsine comporte une poche hydrophobe dans laquelle se loge l'acide aminé du substrat. Ceci permet le positionnement de la liaison à cliver en regard de la sérine catalytique.
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| Chymotrypsinogène B1 | ||
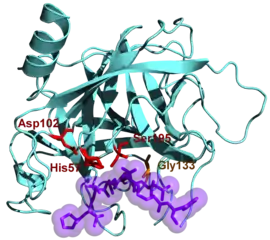 Représentation d'une α-chymotrypsine (PDB 4CHA) | ||
| Caractéristiques générales | ||
|---|---|---|
| Symbole | CTRB1 | |
| Homo sapiens | ||
| Locus | 16q23.1 | |
| Masse moléculaire | 27 870 Da[1] | |
| Nombre de résidus | 263 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Chymotrypsinogène B2 | ||
| Caractéristiques générales | ||
| Symbole | CTRB2 | |
| Homo sapiens | ||
| Locus | 16q23.1 | |
| Masse moléculaire | 27 923 Da[1] | |
| Nombre de résidus | 263 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Chymotrypsine C | ||
| Caractéristiques générales | ||
| Symbole | CTRC | |
| N° EC | 3.4.21.2 | |
| Homo sapiens | ||
| Locus | 1p36.21 | |
| Masse moléculaire | 29 484 Da[1] | |
| Nombre de résidus | 268 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
Synthèse et activation
La chymotrypsine est produite par le pancréas sous une forme inactive appelée le chymotrypsinogène. L'activation est provoquée par la trypsine qui coupe cette molécule en deux chaînes, puis par la chymotrypsine elle-même lors d'une trans-protéolyse, donnant à la fin une structure globulaire compacte de trois chaînes reliées par deux ponts disulfures et repliées en 2 domaines de 120 acides aminés.
Ces domaines adoptent la conformation d'un tonneau β formé de 6 brins β.
Le site actif est situé dans une crevasse bordée par les 2 domaines. Il fixe le substrat au niveau de 2 régions (la région 214-216, liaison non spécifique, et la région 189-216 et 226, liaison spécifique).
Mécanisme d'action
La chymotrypsine intervient dans la protéolyse des protéines du système digestif des mammifères et autres êtres vivants. Elle catalyse le clivage des chaînes polypeptidiques par hydrolyse, mécanisme au départ extrêmement lent sans un activateur.
La chymotrypsine attaque les groupes carbonyles potentiellement nucléophiles impliqués dans une liaison peptidique grâce à la sérine 195, laquelle se lie à son substrat pour former un intermédiaire substrat-enzyme covalent. Le site actif de l'enzyme fait également intervenir les acides aminés His57, Asp102 et Gly193.
Il a été montré que la réaction de la chymotrypsine avec son substrat se produit en deux phases : une phase initiale d'éclatement au tout début de la réaction, puis une phase d'état stationnaire qui suit la loi de Michaelis-Menten.
La chymotrypsine doit absolument être conservée au froid pour garder l'activité enzymatique.
 Mécanisme d'action de la chymotrypsine.
Mécanisme d'action de la chymotrypsine.
Séquence
Séquence du chymotrypsinogène B1 (gènes 16q23 et 16q24.1)
10 20 30 40 50 60
MASLWLLSCF SLVGAAFGCG VPAIHPVLSG LSRIVNGEDA VPGSWPWQVS LQDKTGFHFC
70 80 90 100 110 120
GGSLISEDWV VTAAHCGVRT SDVVVAGEFD QGSDEENIQV LKIAKVFKNP KFSILTVNND
130 140 150 160 170 180
ITLLKLATPA RFSQTVSAVC LPSADDDFPA GTLCATTGWG KTKYNANKTP DKLQQAALPL
190 200 210 220 230 240
LSNAECKKSW GRRITDVMIC AGASGVSSCM GDSGGPLVCQ KDGAWTLVGI VSWGSDTCST
250 260
SSPGVYARVT KLIPWVQKIL AAN
Séquence du chymotrypsinogène B2 (gène 16q23.1)
10 20 30 40 50 60
MAFLWLLSCW ALLGTTFGCG VPAIHPVLSG LSRIVNGEDA VPGSWPWQVS LQDKTGFHFC
70 80 90 100 110 120
GGSLISEDWV VTAAHCGVRT SDVVVAGEFD QGSDEENIQV LKIAKVFKNP KFSILTVNND
130 140 150 160 170 180
ITLLKLATPA RFSQTVSAVC LPSADDDFPA GTLCATTGWG KTKYNANKTP DKLQQAALPL
190 200 210 220 230 240
LSNAECKKSW GRRITDVMIC AGASGVSSCM GDSGGPLVCQ KDGAWTLVGI VSWGSRTCST
250 260
TTPAVYARVA KLIPWVQKIL AAN
Notes et références
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
Liens externes
- http://www.chem.qmul.ac.uk/iubmb/enzyme/EC34/3421.html#2101
- http://www.genome.jp/dbget-bin/www_bget?enzyme+3.4.21.1
- http://www.genome.jp/dbget-bin/www_bget?hsa:1504
- http://www.genome.jp/dbget-bin/www_bget?hsa:440387
- http://www.brenda-enzymes.org/php/result_flat.php4?ecno=3.4.21.1
- Portail de la biochimie