Consanguinité
En génétique des populations, la consanguinité est définie comme étant le résultat d’une reproduction sexuée entre deux individus apparentés (c'est-à-dire ayant un ou plusieurs ancêtres communs). Pour un descendant donné, elle est d’autant plus importante que le lien de parenté entre les géniteurs est étroit. Concept d'abord empirique et approximatif lié à l'élevage, ainsi qu'à certaines pratiques matrimoniales, il a été formalisé et précisé par la génétique avec la découverte des supports matériels de l'hérédité. En anthropologie de la parenté, la consanguinité est la relation entre les descendants d'un même ancêtre.
Consanguinité
Définitions et mesures de la consanguinité
La notion de consanguinité est définie comme étant le fruit d’une reproduction entre deux individus apparentés.
Chez les organismes diploïdes, le coefficient de consanguinité est la probabilité pour que deux allèles que possède un individu en un locus quelconque soient identiques par descendance. Il est noté F et est compris entre 0 et 1. Ceci suppose donc qu'il existe un ou plusieurs ancêtres (A1 et/ou A2) communs aux deux parents (P1 et P2) de l'individu (I) étudié, et on a alors :
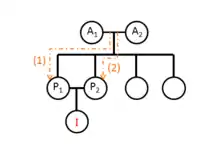
avec ni la somme du nombre de maillons reliant chacun des parents à leur ancêtre commun Ai : dans l'exemple du schéma ci-contre, la situation est symétrique, c'est-à-dire que chacun des parents est lié par le même nombre de maillons à l'ancêtre commun. De plus ceci est vrai pour chacun des ancêtres communs (A1 et A2),
on a donc et .
Les fractions représentent la probabilité de transmettre un même allèle à sa progéniture, ce qui se reproduit fois pour chaque ancêtre commun. De plus, si ces ancêtres communs A1 et A2 sont eux-mêmes consanguins avec un coefficient , on a une probabilité « augmentée » de transmettre un allèle identique à la génération suivante (le long des maillons fléchés (1) et (2) sur le schéma ci-contre). Cette probabilité s'exprimera alors
En ce qui concerne notre exemple (schéma ci-contre), si on suppose (cas théorique idéal) , alors on a
La consanguinité modifie donc les fréquences génotypiques mais laisse les fréquences alléliques non altérées : il s'agit d'un remaniement des associations d'allèles au sein des individus, et non d'une perte/fixation d'allèles. Puisque la consanguinité favorise l'union de gamètes semblables, on observe une perte d'hétérozygotie au sein des populations consanguines, comme le montre le tableau suivant de comparaison avec le modèle panmictique de Hardy-Weinberg :
| Type | équilibre Hardy-Weinberg | Population consanguine |
|---|---|---|
| Fréquence [AA] | ||
| Fréquence [aa] | ||
| Fréquence [Aa] |
où p et q sont les fréquences alléliques respectives des allèles A et a (on remarques qu'elles restent bien inchangées dans le cas d'une population consanguine, seules les fréquences génotypiques sont altérées).
NB : F peut donc s'interpréter comme la proportion d'individus qui auraient été hétérozygotes sous panmixie mais qui sont en réalité des homozygotes du fait des accouplements consanguins au sein de cette population.
En pratique, selon les populations étudiées, on distingue trois façons de définir et mesurer la consanguinité, toutes basées sur les F-statistiques (en) de Sewall Wright[1] :
- La consanguinité de pedigree
- (c'est le cas d'étude détaillé ci-dessus) un individu est dit consanguin si ses parents sont proches d’un point de vue généalogique. Le niveau de consanguinité est lié au degré de parenté qui existe entre les ascendants. La connaissance de la généalogie de l'individu est utilisée pour calculer son coefficient de consanguinité F, à rapprocher de l'indice de fixation FIT défini par les F-statistiques.
- La consanguinité perçue comme une association non aléatoire d’individus pour la reproduction
- cette définition se base sur la comparaison du degré de parenté de couples parentaux observés au sein d'une population, par rapport à celui de couples formés par l'association de deux individus pris au hasard dans la population (cas de panmixie). Dans ce cas, un individu est considéré comme consanguin si ses parents sont plus étroitement apparentés que deux individus pris au hasard. La mesure la plus utilisée est la déviance de l’hétérozygotie observée chez un individu par rapport à l’hétérozygotie attendue en cas de couplage aléatoire (notée FIS).
- La consanguinité résultant d’une subdivision de la population
- quand les populations sont subdivisées en groupes isolés, la consanguinité résulte alors de la restriction populationnelle qui induit des phénomènes de dérive génétique. La consanguinité peut être observée même en cas de couplage aléatoire au sein de la sous-population. Dans ce cas, on compare le degré de consanguinité attendu si les croisements se faisaient de manière aléatoire au sein de la métapopulation entière, au degré de consanguinité résultant d'un couplage aléatoire au sein des sous-populations : il s'agit du coefficient FST.
Effets de la consanguinité
La consanguinité a globalement pour effet de réduire la valeur sélective (fitness) des individus/populations concerné(e)s, mais ceci de manière très variable. Elle augmente le risque de survenue de maladies génétiques de transmission autosomique récessive uniquement. Ces effets vont fortement dépendre des espèces ou populations considérées et de la manière dont leurs génotypes interagissent avec l’environnement[2].
Dépression de consanguinité
La dépression de consanguinité est l'ensemble des conséquences de la perte d'hétérozygotie résultant de croisements consanguins[1]. En effet, les chances de trouver des individus homozygotes pour des allèles délétères jusqu'alors récessifs augmentent : l'expression de « mauvais » allèles (fardeau génétique) est accrue et la valeur sélective en pâtit.
- Mesure de la dépression de consanguinité
- Différence entre la valeur sélective d’un individu consanguin et celle d’un individu issu d’un croisement aléatoire , rapportée à cette dernière ; soit :
où est appelé coefficient de dépression de consanguinité ou coût de consanguinité[2].
Comment la consanguinité va-t-elle affecter la valeur sélective (fitness) d’un individu ?
Si les locus à l’origine de la dépression de consanguinité interagissent de manière indépendante et additive, la valeur sélective va décliner de façon linéaire pour des consanguinités croissantes[2] :
avec F est le coefficient de consanguinité, et b est un paramètre constant dépendant du gène, de la population et de l'environnement étudiés.
Si en revanche ces mêmes locus interagissent de manière indépendante et multiplicative, alors on s’attend à ce que la valeur sélective diminue de manière exponentielle avec la consanguinité[2] :
où B est un paramètre constant dépendant du gène, de la population et de l'environnement étudiés.
Effet de purge
La dépression de consanguinité se révèle dans certains cas bénéfique pour la population touchée. En effet, l’expression des allèles délétères et récessifs (par l’augmentation de l’homozygotie) peut avoir un effet de « purge » sur le génome, en éliminant par sélection naturelle les individus dont le fardeau génétique serait ainsi exprimé. Ceci conduirait ainsi (sous réserve du rétablissement de la population) à une augmentation de la valeur sélective des individus dans leur milieu à un temps donné.
Ce mécanisme de purge est cependant très limité par le fait qu’il dépende de nombreux facteurs génétiques et environnementaux (population non isolée, effet Hill-Robertson (en) des petites populations…)[1].
De plus il s'agit d'un phénomène à double tranchant, qui, s'il n'a pas lieu, peut favoriser les mécanismes de superdominance (en) liés à la dépression de consanguinité, mais qui est aussi susceptible d'entraîner une perte de diversité génétique qui pourra limiter les capacités d’adaptation des populations en cas de modification de leur environnement.
De façon générale, l’expression des mutations létales, portées par des allèles récessifs, conduit rapidement au phénomène de purge, alors que des allèles récessifs à effets modérés (semi-létaux) ne sont pas aussi exposés à la sélection naturelle.
Écologie et conservation
Ex situ
La conservation ex situ a des objectifs cruciaux dans la sauvegarde des espèces. Des programmes de reproduction ont été mis en place pour constituer des populations viables de 250 à 500 individus par espèces. L’objectif est de garantir plus de 90 % de la diversité génétique des individus fondateurs de la population sur 200 ans. Il est donc nécessaire d’éviter les phénomènes de consanguinité[2],[3].
Les programmes de reproduction sont constitués d’un studbook, d’un coordinateur et d’un plan de transfert défini par le coordinateur lui-même. Les studbooks, répertoriés par ISIS (International Species Information System), listent les individus présents dans les parcs animaliers et permettent de définir la généalogie de l’espèce ou sous-espèces en question. À l’échelle européenne, il existe les ESB (European Studbook) pour les espèces peu menacées, et les EEP (European Endangered species Program) pour les espèces en danger ou peu représentées. Les équivalents américains sont les SSP, et des studbook internationaux existent également (ISB). Pour chaque nouveau studbook, le coordinateur doit répertorier tous les individus ayant vécu en captivité, à partir des premiers individus fondateurs connus, et leur descendance. Ensuite il suffit de mettre à jour les nouveautés (naissances, transferts, morts) chaque année.
Le coordinateur est responsable de proposer des échanges entre les parcs à la suite d'analyses de pedigree[3], afin que chaque fondateur contribue de façon égale à la génération suivante et que les familles soient de taille similaire. Toute population captive présentant un certain degré de consanguinité, il est nécessaire d’adapter la reproduction pour égaliser la représentation des fondateurs : on utilise alors un outil statistique pour estimer le risque de perte d’allèle. Ceci permet d’identifier les individus les plus importants pour la reproduction, c'est-à-dire ayant les allèles les moins représentés. Pour planifier les accouplements, un second modèle recherche les paires optimales (faible coefficient de parenté) de façon à maximiser la diversité génétique résultante[3].
In situ : les relocations d’animaux
Les populations concernées par des mesures de conservation sont très généralement de petite taille. Elles ont de ce fait perdu en variation génétique et sont susceptibles d’avoir fixé des allèles délétères. Pour éviter que cette « détérioration » génétique ne mène à l’extinction, certaines populations peuvent bénéficier de l’introduction d’individus extérieurs[2] :
Réintroduction
L’action finale des programmes de reproduction ex-situ est la réintroduction des individus dans leur milieu naturel. Il est donc d’autant plus important de limiter la consanguinité dans ces programmes pour permettre la réintroduction d’individus riches génétiquement évitant le phénomène de dépression hybride (maladaptation dans le milieu). Idéalement, en conservant 90 % de la diversité génétique des premiers fondateurs, il est possible de garantir le succès des individus dans l’habitat ciblé[3]. Trois stratégies sont principalement utilisées. L’une d’entre elles est de relâcher des individus provenant d’une population connue pour son niveau d’hétérozygotie élevé. La deuxième est de choisir des individus hybrides (issues du croisement de deux populations). Et enfin la dernière est de relâcher des individus issus d’une population géographiquement proche du lieu de réintroduction.
Translocation
Il s’agit d’un second type de renforcement de population. Une translocation est le transfert d’un individu ou d’une population sauvage d’une région de son aire de répartition à une autre. Par exemple, dans des populations où la fitness est diminuée par la consanguinité, une translocation peut ré-augmenter sa diversité génétique. Dans la translocation comme dans la réintroduction, le grand débat est alors de savoir si le patrimoine génétique des individus transférés ne va pas diminuer la fitness des descendants. D’autre part, ces renforcements de population peuvent induire l’introduction de parasites ou de maladies.
Les pumas de Floride[4] sont un exemple réussi de translocation : en 1995, la population de panthères de Floride (Puma concolor coryi), isolée des populations des autres panthères de la même espèce, a subi une restauration génétique grâce à la translocation de huit femelles de panthère du Texas (Puma concolor stanleyana), sous-espèce la plus proche géographiquement. Les descendants hybrides avaient un taux de survie supérieur aux descendants purs (donc toujours affectés par la consanguinité) et la population de Floride a vu sa densité et son aire de répartition augmenter.
Couloirs écologiques ou corridors biologiques
La fragmentation de l’habitat et la diminution d’espace naturel est responsable d'une diminution du taux de dispersion des populations. Ceci diminue donc la diversité génétique des sous-populations et induit la consanguinité. Ainsi, pour permettre les échanges d’individus et donc de gènes, des corridors écologiques peuvent être mis en place pour soutenir la migration et/ou la survie des migrateurs. Par exemple, construire des ponts ou des tunnels reliant les deux côtés de la route visent à permettre à des cervidés ou des rongeurs de traverser sans se faire écraser .
Exemples célèbres de populations consanguines
Certains exemples reviennent souvent lorsque l'on parle de consanguinité ; en voici une petite liste non exhaustive.
Les guépards : leur espèce présente un taux bas d'hétérozygotie, ce qui, par extrapolation, a suggéré que l'espèce était consanguine et devait donc souffrir de dépression de consanguinité (mortalité juvénile, vulnérabilité à la maladie, altération du comportement…)[5]. Si la possibilité d'une telle conclusion n'est pas à exclure totalement, force est de constater que les justifications avancées ont été néanmoins remises en cause du fait du manque de vraisemblance de certaines des hypothèses sous-jacentes[6].
Les bisons d'Europe : ils représentent un très bon exemple de conservation fondée sur un programme de reproduction en captivité. Cette espèce se divisant en deux sous-espèces, dont l'une (Bison bonasus caucasinus) n'est plus actuellement représentée que par une lignée mixte (Bison bonasus caucasinus x Bison bonasus bonasus), ce cas met bien en valeur les problématiques liées à la difficulté d'éviter la consanguinité tout en gardant intactes des divisions taxonomiques aussi délicates que la sous-espèce.
Le loup gris[7] : à la suite des campagnes d’extermination du loup en Scandinavie, cet animal a été considéré comme éteint dans les années 1960. Mais on a découvert dans les années 1980 une meute de provenance inconnue. Les premières analyses génétiques ont mis en évidence une perte importante de la diversité génétique et montré que la population s’était constituée à partir de deux individus fondateurs. Malgré cela et grâce au maintien de conditions adéquates (proies disponibles, chasse limitée, etc.), on a observé à partir de 1991 une croissance exponentielle de la population et une hausse de la diversité génétique, ce qui a fait supposer que ces phénomènes étaient dus à l'immigration d’un unique individu mâle appartenant à une population voisine. Des migrations, même rares et limitées en nombre d’individus, peuvent donc être réellement salvatrices pour des populations consanguines.
Les tigres blancs en captivité sont tous issus du même individu : Mohan. La consanguinité est très élevée et conduit à des déformations graves dès la naissance.
Homologie de séquence ADN selon le degré de parenté
Le degré d'homologie[8] présenté dans cette section correspond à la proportion d'ADN qui est identique entre deux individus donnés. Il ne doit pas être confondu avec le degré de consanguinité, qui correspond à la proportion d'homozygotie chez un individu donné (c'est-à-dire à la proportion d'ADN qui présente une séquence identique sur les deux allèles d'un individu donné au sein de l'ensemble de son génome).
| Degré d'homologie | Lien de parenté entre les individus |
|---|---|
| 1 | jumeau identique |
| 0,5 (1/2) | parent – enfant |
| 0,25 (1/4) | grand-père, grand-mère, petit-fils et petite-fille |
| 0,125 (1/8) | arrière-grand-père, arrière-grand-mère, arrière-petit-fils et arrière-petite-fille |
| 0,0625 (1/16) | arrière-arrière-grand-père, arrière-arrière-grand-mère, arrière-arrière-petit-fils et arrière-arrière-petite-fille |
| 0,03125 (1/32) | arrière-arrière-arrière-grand-père, arrière-arrière-arrière-grand-mère, arrière-arrière-arrière-petit-fils et arrière-arrière-arrière-petite-fille |
| 0,5 (1/2) | frère ou soeur (bilatéral) |
| 0,25 (1/4) | demi-frère et demi-sœur |
| 0,25 (1/4) | oncle, tante, neveu et nièce |
| 0,125 (1/8) | demi-oncle, demi-tante, demi-neveu et demi-nièce |
| 0,125 (1/8) | grand-oncle, grand-tante, petit-neveu et petite-nièce |
| 0,0625 (1/16) | demi-grand-oncle, demi-grand-tante, demi-petit-neveu et demi-petite-nièce |
| 0,0625 (1/16) | arrière-grand-oncle, arrière-grand-tante, arrière-petit-neveu et arrière-petite-nièce |
| 0,03125 (1/32) | arrière-arrière-grand-oncle, arrière-arrière-grand-tante, arrière-arrière-petit-neveu et arrière-arrière-petite-nièce |
| 0,03125 (1/32) | demi-arrière-grand-oncle, demi-arrière-grand-tante, demi-arrière-petit-neveu et demi-arrière-petite-nièce |
| 0,015625 (1/64) | demi-arrière-arrière-grand-oncle, demi-arrière-arrière-grand-tante, demi-arrière-arrière-petit-neveu et demi-arrière-arrière-petite-nièce |
| 0,0625 (1/16) | demi-cousin et demi-cousine |
| 0,125 (1/8) | cousin et cousine |
| 0,1875 (3/16) | sesqui-cousin et sesqui-cousine |
| 0,25 (1/4) | double-cousin et double-cousine |
| 0,03125 (1/32) | demi-grand-cousin, demi-grand-cousine, demi-petit-cousin et demi-petite-cousine |
| 0,0625 (1/16) | grand-cousin, grand-cousine, petit-cousin et petite-cousine |
| 0,09375 (3/32) | sesqui-grand-cousin, sesqui-grand-cousine, sesqui-petit-cousin et sesqui-petite-cousine |
| 0,125 (1/8) | double-grand-cousin, double-grand-cousine, double-petit-cousin et double-petite-cousine |
| 0,015625 (1/64) | demi-cousin deuxième et demi-cousine deuxième |
| 0,03125 (1/32) | cousin deuxième et cousine deuxième |
| 0,046875 (3/64) | sesqui-cousin deuxième et sesqui-cousine deuxième |
| 0,0625 (1/16) | double-cousin deuxième et double-cousine deuxième |
| 0,078125 (1/128) | sester-cousin deuxième et sester-cousine deuxième |
| 0,09375 (3/32) | triple-cousin deuxième et triple-cousine deuxième |
| 0,109375 (7/64) | sesqua-cousin deuxième et sesqua-cousine deuxième |
| 0,125 (1/8) | quadruple-cousin deuxième et quadruple-cousine deuxième |
| 0,015625 (1/64) | demi-arrière-grand-cousin, demi-arrière-grand-cousine, demi-arrière-petit-cousin et demi-arrière-petite-cousine |
| 0,03125 (1/32) | arrière-grand-cousin, arrière-grand-cousine, arrière-petit-cousin et arrière-petite-cousine |
| 0,046875 (3/64) | sesqui-arrière-grand-cousin, sesqui-arrière-grand-cousine, sesqui-arrière-petit-cousin et sesqui-arrière-petite-cousine |
| 0,0625 (1/16) | double-arrière-grand-cousin, double-arrière-grand-cousine, double-arrière-petit-cousin et double-arrière-petite-cousine |
| 0,0078125 (1/128) | demi-grand-cousin deuxième, demi-grand-cousine deuxième, demi-petit-cousin deuxième et demi-petite-cousine deuxième |
| 0,015625 (1/64) | grand-cousin deuxième, grand-cousine deuxième, petit-cousin deuxième et petite-cousine deuxième |
| 0,0234375 (3/128) | sesqui-grand-cousin deuxième, sesqui-grand-cousine deuxième, sesqui-petit-cousin deuxième et sesqui-petite-cousine deuxième |
| 0,03125 (1/32) | double-grand-cousin deuxième, double-grand-cousine deuxième, double-petit-cousin deuxième et double-petite-cousine deuxième |
| 0,0390625 (5/128) | sester-grand-cousin deuxième, sester-grand-cousine deuxième, sester-petit-cousin deuxième et sester-petite-cousine deuxième |
| 0,046875 (3/64) | triple-grand-cousin deuxième, triple-grand-cousine deuxième, triple-petit-cousin deuxième et triple-petite-cousine deuxième |
| 0,0546875 (7/128) | sesqua-grand-cousin deuxième, sesqua-grand-cousine deuxième, sesqua-petit-cousin deuxième et sesqua-petite-cousine deuxième |
| 0,0625 (1/16) | quadruple-grand-cousin deuxième, quadruple-grand-cousine deuxième, quadruple-petit-cousin deuxième et quadruple-petite-cousine deuxième |
| 0,0078125 (1/128) | cousin troisième et cousine troisième |
Sociétés humaines
Reproduction consanguine
L'évitement de la consanguinité passerait, chez l'homme, par la mise en place de barrières psychologiques : Freud, ou encore Westermarck proposeront leur vision de ces mécanismes plus ou moins inconscients.
Durant l'Ancien Régime en France il n'était pas rare de voir dans certaines familles royales ou aristocratiques des mariages entre personnes de même famille. Aujourd'hui, en France, la consanguinité reste une pratique extrêmement marginale du fait entre autres, des progrès de la science dans le domaine de la génétique. Néanmoins certains foyers dits « traditionnels » demeurent, Paul Hervier dément l'idée reçue d'un nord de la France contemporainement consanguin, il explique que cela constitue une pratique socio-culturelle remontant à plusieurs siècles et cantonnée aujourd'hui aux campagnes reculées. Le véritable foyer de la consanguinité en France d'après P. Hervier est la Bretagne, ce qui s'explique par des racines historiques proches de la culture celtique. À l'échelle régionale cela reste très inégalement réparti, en effet ce phénomène est observable par les sociologues quasiment uniquement en dehors des grands pôles urbains (Rennes, Brest, etc.)[9].
Conséquences

- Non consanguins
- Consanguins
Un gène muté n'est pas nécessairement bénéfique ou nocif de façon intrinsèque, et une variation d'environnement peut changer la donne. Voir le double exemple de Biston betularia avant, pendant et après la révolution industrielle, ainsi que la section Risque de ne pas effectuer les bons choix dans l'article Eugénisme : la nature, ne planifiant pas, peut fort bien ne pas effectuer une meilleure sélection que les hommes. Le temps gommera simplement ses erreurs, quand il y en a eu.
Chez l'Homme
De nombreuses études scientifiques et médicales ont mis en évidence chez l'Homme des effets sur la santé de l'enfant « consanguin », avec (en moyenne) :
Croyances et réalités
Une relative consanguinité était fréquente dans de nombreuses sociétés rurales ou insulaires, induite par la difficulté matérielle qu'il y avait à chercher au loin un conjoint présentant des caractéristiques génétiques différentes ; les études de localisation géographique des noms de famille laissaient penser que le brassage génétique était autrefois plus limité qu'aujourd'hui. Des nombreux cas de croisements entre individus étroitement apparentés donnaient lieu à des enfants atteints de maladies génétiques mortelles et au XIXe siècle il est couramment admis que la tare héréditaire ou congénitale résulte fréquemment de l'influence néfaste d'une consanguinité importante. Cette consanguinité a été considérée comme un facteur explicatif de décadence, par exemple dans des familles royales (ce qui pourra être l'un des arguments pour leur disparition ou remplacement par un autre système de gouvernement lors de révolutions). La génétique a confirmé que la procréation consanguine concentre certaines caractéristiques génétiques défavorables.
La part de responsabilité du viol au sein de la famille et de l'inceste reste inconnue pour le passé et mal connue pour le présent, de même que le nombre de relations sexuelles hors du couple dans le cas de couples consanguins (par exemple mariés de force au sein de leur famille).
Des chercheurs et sociobiologistes estiment que l'évitement de l'inceste et des relations sexuelles entre parents du premier et du deuxième degré, est un processus inné, décrit par l'effet Westermarck, commun à toutes les sociétés humaines, qui indiquerait que la diversité génétique est une condition de survie pour l'espèce, la sélection naturelle nous poussant alors à éviter la consanguinité. Cette option se distingue de celle de Claude Lévi-Strauss qui y voit essentiellement un phénomène social : « donner » ses sœurs pour avoir l'appui de beaux-frères partout où l'on sera amené à passer, mais peut fort bien coexister avec elle. On sait aussi que la consanguinité induit chez les enfants une moindre capacité à se reproduire et une mortalité plus élevée[20],[21],[22].
Prévalence
Les tabous socioculturels et religieux autour de l'inceste ont longtemps rendu la fréquence et le degré de consanguinité très difficile à mesurer. Les progrès de la génomique permettent maintenant d'en savoir plus. Ainsi une étude[25] récente (2019) a pu préciser le degré de consanguinité qui prévaut actuellement au sein d'un groupe important d'humains (à partir d'une base de données biomédicales concernant environ 450 000 personnes), grâce à une étude génomique anonymisée à grande échelle ; cette étude a conclu qu'un seul cas sur 3652 européen(es) (issu(e)s de deux parents européens[26],[27] né(es) entre 1938 et 1967) est né de parents très proches (extrême consanguinité, soit plus de 10 % d'ADN commun, indiquant un couple de géniteurs de type frères-sœurs, parent-enfant ou grand-parent-petit-enfant…) Cette étude a permis de confirmer que les personnes ayant une haute consanguinité tendent à être plus petites, moins musclées et à avoir des capacités cognitives plus faibles que la moyenne[19]. Les auteurs signalent que la base de données (UKB) qu'ils ont utilisée présente un biais de représentativité, car elle inclut « notamment des participants en meilleure santé et plus éduqués que la population moyenne », ce qui pourrait avoir conduit à une sous-estimation du nombre réel de cas de consanguinité extrême[28].
Exemples célèbres
Vers 1775, le typhon Lengkieki a dévasté l'atoll de Pingelap, dans l'état de Pohnpei, un des États fédérés de Micronésie. Le typhon et la famine qui a suivi n'ont laissé qu'une vingtaine de survivants, dont l'un était porteur d'un gène de l'achromatopsie, maladie génétique caractérisée par une absence totale de vision des couleurs, une forte photophobie, une acuité visuelle très réduite et un nystagmus. Quelques générations plus tard, à cause de la consanguinité, entre 8 et 10 % de la population est atteinte d'achromatopsie, et environ 30 % des habitants de l'atoll sont porteurs sains du gène, alors que la fréquence normale de l'achromatopsie est de 1/33000[29]. Cette proportion inhabituelle d'achromates dans la population a attiré le neurologue Oliver Sacks dans l'île. À la suite de ce voyage, il a écrit son livre L'Île en noir et blanc[30] en 1997.
Études anthropologiques
Des anthropologues sont devenus célèbres pour avoir développé des théories anthropologiques sur les structures de parenté. C'est le cas de Claude Lévi-Strauss, fondateur du structuralisme, qui, dans sa thèse (Les structures élémentaires de la parenté, 1955), montre que toute société structure les liens de parenté de manière différente afin d'éviter l'inceste, lequel est un tabou universel. D'autres anthropologues, tel que le jésuite Léon de Sousberghe, contesteront cette loi universelle en étudiant des sociétés où les unions préférentielles augmentent la consanguinité.
Notes et références
- (en) Keller, L.F. and Waller, D.M. (2002) « Inbreeding effects in wild populations » Trends in Ecology & Evolution 17(5), 230-241
- (en) Hedrick, P.W. and Kalinowski, S.T. (2000) « Inbreeding depression in Conservation Biology » Annual Review of Ecology and Systematics 31, 139-162
- (en) Ebenhard, T. (1995) « Conservation breeding as a tool for saving animal species from extinction » Trends in Ecology & Evolution 10(11), 438-443
- Pimm, S.L., Dollar, L., Bass O.L. Jr (2006) « The genetic rescue of the Florida panther » Animal Conservation 9(2), 115–122
- (en) O'Brien, S.J., Roelke, M.E., Marker, L., Newman, A., Winkler, C.A., Meltzer, D., Colly, L., Evermann, J.F., Bush, M. and Wildt, D.E. (1985) « Genetic basis for species vulnerability in the cheetah » Science 227(4693), 1428-1434
- (en) Caughley, G. (1994) « Directions in conservation biology » Journal of Animal Ecology 63(2), 215-244
- (en) Vilà, C., Sundqvist, A.-K., Flagstad, Ø., Seddon, J., Bjö rnerfeldt, S., Kojola, I., Casulli, A., Sand, H., Wabakken, P. and Ellegren, H. (2003) « Rescue of a severely bottlenecked wolf (Canis lupus) population by a single immigrant » Proceedings of the Royal Society of London B: Biological Sciences 270, 91-97
- (en) « Average Percent DNA Shared Between Relatives »
- Paul Hervier, Les mariages consanguins
- (en) Mohd Fareed et Mohammad Afzal, « Estimating the Inbreeding Depression on Cognitive Behavior: A Population Based Study of Child Cohort », PLOS ONE, vol. 9, no 10, , e109585 (ISSN 1932-6203, PMID 25313490, PMCID PMC4196914, DOI 10.1371/journal.pone.0109585, lire en ligne, consulté le )
- Dorsten, L. E., Hotchkiss, L. & King, T. M. The effect of inbreeding on early childhood mortality: twelve generations of an amish settlement. Demography 36, 263–271 (1999).
- Yengo, L. et al. (2017) Detection and quantification of inbreeding depression for complex traits from SNP data. Proc. Natl Acad. Sci. USA 114, 8602–8607.
- Huisman, J., Kruuk, L. E. B., Ellis, P. A., Clutton-Brock, T. & Pemberton, J. M. (2016) Inbreeding depression across the lifespan in a wild mammal population. Proc. Natl Acad. Sci. 113, 3585–3590.
- Charlesworth, B. & Charlesworth, D. (1999) The genetic basis of inbreeding depression. Genet. Res. 74, 329–340
- Joshi, P. K. et al. Directional dominance on stature and cognition in diverse human populations. Nature 523, 459–462 (2015).
- Johnson, E. C., Evans, L. M. & Keller, M. C. (2018) Relationships between estimated autozygosity and complex traits in the UK Biobank. PLoS Genet. 14, e1007556.
- Saura, M. et al.(2015) Detecting inbreeding depression for reproductive traits in Iberian pigs using genome-wide data. Genet. Sel. Evol. 47, 1.
- Morton, N. E., Crow, J. F. & Muller, H. J(1956) An estimate of the mutational damage in man from data on consanguineous marriages. Proc. Natl Acad. Sci. 42, 855–863
- Extreme inbreeding’ revealed by massive human-genome survey ; Analysis suggests that roughly one in 3,600 people studied were born to closely related parents ; Nature Commun. (2019) Génomique ; publiée comme brève dans Nature le 06 septembre 2019
- Morton, N. E., Crow, J. F. & Muller, H. J. An estimate of the mutational damage in man from data on consanguineous marriages. Proc. Natl Acad. Sci. 42, 855–863 (1956).
- Fareed, M., Kaisar Ahmad, M., Azeem Anwar, M. & Afzal, M. Impact of consanguineous marriages and degrees of inbreeding on fertility, child mortality, secondary sex ratio, selection intensity, and genetic load: a cross-sectional study from Northern India. Pediatr. Res. 81, 18–26 (2017).
- Dorsten, L. E., Hotchkiss, L. & King, T. M (1999) The effect of inbreeding on early childhood mortality: twelve generations of an amish settlement. Demography 36, 263–271.
- (en) « Global prevalence tables », sur consang.net (consulté le )
- (en) Abdelmajid Hammami, Mohamed Elgazzeh, Noureddine Chalbi et Ben Abdallah Mansour, « Endogamy and consanguinity in Mauritania », La Tunisie Medicale, vol. 83, no 1, , p. 38–42 (ISSN 0041-4131, PMID 15881720, lire en ligne, consulté le )
- Yengo, L., Wray, N. R., & Visscher, P. M. (2019) Extreme inbreeding in a European ancestry sample from the contemporary UK population. Nature communications, 10(1), 1-11.
- base de données de la Biobanque britannique (UKB)
- Bycroft, C. et al. The UK (2018). Biobank resource with deep phenotyping and genomic data. Nature 562, 203
- Fry, A. et al. (2017) Comparison of sociodemographic and health-related characteristics of UK biobank participants with those of the general population. Am. J. Epidemiol. 186, 1026–1034.
- (en) J Francois, Heredity in ophthalmology, St. Louis, Mosby,
- (en) Oliver Sacks, The Island of the Colourblind, Sydney, Picador, , 345 p. (ISBN 978-0-330-35887-3, OCLC 37444083)
Voir aussi
Bibliographie
- Frankham, R. (2005) Genetics and extinction. Biological Conservation 126(2), 131-140
- Frankham, R. (2003) Genetics and conservation biology. Comptes Rendus Biologies 326(1), 22-29
- Reed, D.H. and Frankham, R. (2000) Correlation between fitness and genetic diversity. Conservation Biology 17(1), 230–237
- Ebenhard, T. (1995) Conservation breeding as a tool for saving animal species from extinction. Trends in Ecology & Evolution 10(11), 438-443
- Jean Sutter et Léon Tabah, « Fréquence et répartition des mariages consanguins en France », Population, vol. 3, no 4, , p. 607-630 (JSTOR 1524358)
- Brian Van DOORMAAL (2008), La consanguinité au fil du temps ; Réseau laitier canadien ; , 32e symposium sur les bovins laitiers ; Drummondville (PDF, 12 p.)
Articles connexes
Liens externes
- Notices dans des dictionnaires ou encyclopédies généralistes :
- Ressource relative à la santé :
- Cours complet sur la consanguinité et son coefficient
- Méthode de calcul du taux de consanguinité
- (en) Le pdf « IUCN Guidelines for Re-introductions » sur le site de téléchargements de l'IUCN
- (fr) Calcul du coefficient de consanguinité par la méthode des chemins
- (fr) Algérie : près de 38 % des mariages sont consanguins ()
- (en) Carte de prévalence de la consanguinité dans le monde
- Portail de la biologie
- Portail de l’anthropologie
- Portail de la médecine
