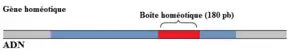
Boîte homéotique
Une boîte homéotique (appelée aussi homéoboîte, homeobox en anglais) est une séquence d'ADN qu'on retrouve dans certains gènes essentiels au développement embryonnaire (morphogenèse) des animaux, des champignons et des plantes. Ainsi, un gène contenant une boîte homéotique va coder un homéodomaine qui grâce à sa conformation se lie à l’ADN et permet d’activer en cascade d'autres gènes. C'est le cas, par exemple, de tous les gènes nécessaires pour l'identité et le développement d'un segment du corps, appelés gènes homéotiques chez Drosophila, une mouche à fruits. Il est important de faire la distinction suivante : un gène possédant une boîte homéotique n'est pas nécessairement un gène homéotique, même si tous les gènes homéotiques possèdent une séquence boîte homéotique.
Cet article possède un paronyme, voir Homebox (homonymie).
Structure et fonctions
Une boîte homéotique est composée d'environ 180 paires de bases (voir figure 1) codant un domaine protéique de 60 acides aminés, l’homéodomaine (ou domaine homéotique) qui est bien conservée (avec relativement les mêmes acides aminés dans les différents homéodomaines)[1]. Cet homéodomaine est capable de se lier à l'ADN pour contrôler l’expression des gènes. Ainsi, l'homéodomaine permet la reconnaissance d'une séquence spécifique d’ADN d’un gène. Les protéines contenant ces homéodomaines, les homéoprotéines, agissent comme facteurs de transcription régulant l'expression de ces gènes cibles durant le développement embryonnaire, la différenciation cellulaire et la spécification du destin de la cellule[2].
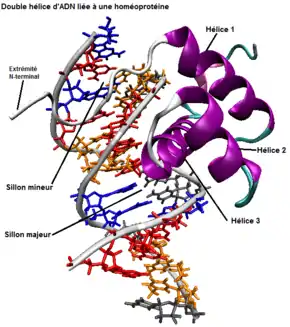
L’homéodomaine est une structure tridimensionnelle appelée hélice-tour-hélice (HTH) et comporte trois hélices alpha séparées par une boucle et un tour (voir figure 2). La troisième hélice alpha va venir s'insérer dans le sillon majeur de la double hélice d'ADN au moment de la reconnaissance et va rendre cette dernière active pour la transcription[3].
Les homéoprotéines en tant que facteurs de transcriptions remplissent une grande variété de fonctions biologiques. Il n’existe probablement pas de tissus dans les plantes ou les animaux qui ne requièrent pas ces protéines pour fonctionner correctement. Chez les animaux, elles agissent dès les premiers stades de développement et elles sont essentielles dans les cellules souches embryonnaires[4]. Plusieurs sont impliquées dans le développement du système nerveux et la perturbation des gènes homéotiques peut mener à divers troubles et maladies génétiques. Également chez les plantes, les gènes homéotiques régulent de nombreux aspects du développement comme la maintenance des cellules souches, la réponse au stress et la réponse à la lumière[1].
Reconnaissance de la séquence d’ADN par l’homéodomaine
La reconnaissance de la séquence par l’homéodomaine se fait grâce à l’hélice 3, mais la boucle entre les hélices 1 et 2 et le bras flexible N-terminal (i.e. extrémité de la protéine avec une fonction amine libre [-NH2]) de l’homéoprotéine aident à stabiliser cette dernière. Cette extrémité N-terminale de protéine homéotique va venir s’aligner dans le sillon mineur de la double hélice d’ADN (voir figure 2). L’hélice 3 et la boucle entre l’hélice 1 et 2 sont riches en arginine et en lysine qui forment des ponts hydrogène avec la structure de l’ADN. C’est grâce à ces liens hydrogène et à des interactions hydrophobes que l’homéoprotéine vient interagir avec l’ADN, s’y stabiliser et s’y attacher[3]. La majorité des séquences d’ADN qui se lient efficacement avec l’homéodomaine contiennent la série de bases azotées suivantes : ATTA (ou TAAT complémentaire) qui interagissent avec les acides aminés de l’homéodomaine. Toutefois, comme il existe une grande conservation de la séquence d’acides aminés dans l’homéodomaine, une même séquence nucléotidique de l’ADN peut être reconnue par plusieurs homéodomaines. Ainsi, la spécificité de la reconnaissance doit être assurée par des cofacteurs qui coopèrent avec l’homéoprotéine. De plus, ces protéines homéotiques possèdent la propriété de se dimériser, c’est-à-dire de s’associer entre elles. Ces deux possibilités forment des complexes protéiques qui affinent la spécificité de la reconnaissance des homéoprotéines sur le site de fixation de l’ADN[3],[5].
Historique et découverte des boîtes homéotiques
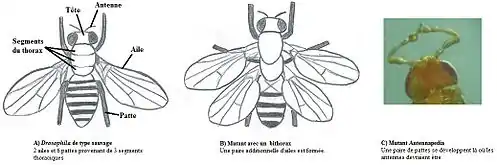
Un groupe de scientifiques composé de Michael Levine, William McGinnis et de Ernst Hafen et dirigé par Walter Jakob Gehring ont découvert la première boîte homéotique chez les eucaryotes en 1983[6]. Ce serait la plus grande avancée en biologie après la découverte de Watson et Crick sur la structure de l’ADN en 1953 et l’élucidation subséquente du code génétique dans les années 60. L’équipe de Gehring s’intéressait aux effets développementaux importants associés au gène Antennapedia (Antp) de l’organisme modèle Drosophila, une mouche à fruits. Normalement, le segment de la tête de cet organisme porte une paire d’antennes, mais lors de leur expérience, ce segment s’est développé plutôt en un segment de corps avec une paire d’appendices (voir figure 3). Gehring pensait que des duplications (c'est-à-dire mutations géniques caractérisées par le doublement du matériel génétique d'une large portion chromosomique, d'un gène ou bien d'une suite nucléotidique) dans les gènes régulateurs (i.e. gène structural qui code une protéine impliquée dans la régulation de l'expression d'autres gènes) pourraient résulter en des transformations des segments de manière partielle ou complète. En utilisant les méthodes présentes à l’époque pour étudier les chromosomes, Gehring a fait une recherche chromosomique en utilisant le mutant du gène Antennapedia et le mutant du gène Bithorax pour voir s’il y avait des copies de ces gènes dans le génome de Drosophila et il en a trouvé plusieurs. Ces résultats confirment ce qu’Edward B. Lewis avait postulé vers la fin des années 1970 en ce qui concerne la duplication de gènes en tandem (i.e. duplication d’une séquence donnant deux séquences identiques, l’une après l’autre, dans un segment d’un chromosome). Cela a encouragé Gehring à émettre l’hypothèse que ces mutations sont en fait des copies des gènes qui confèrent l’identité unique du segment dans lequel ils apparaissent[7].
Depuis la découverte des boîtes homéotiques de l’équipe de Gehring dans les années 1980, beaucoup plus d’attentions ont été dirigées vers une branche grandissante de la biologie, soit la biologie évolutive du développement (ou evo-devo qui est l’abréviation anglaise reconnue). Le but premier des scientifiques dans ce domaine est de combler le manque d’informations qui permettraient de lier l’ontogénie évolutive partagée entre différents taxons et la biologie développementale. En 1980, avant même la découverte des boîtes homéotiques, une recherche à grande échelle avait été conduite sur le modèle expérimental Drosophila melanogaster dans le but de trouver des mutations de gènes affectant le processus de développement de cette espèce. Certains phénotypes mutants résultant comme la larve piquante connue sous le nom de «hérisson» sont devenus célèbres depuis cette étude. Cette dernière a permis de faire passer la biologie développementale de sa forme primitive, très descriptive, comparative et expérimentale à une phase plus avancée, traitant de génétique. Depuis, grâce à un effort considérable de la part de toute une gamme de disciplines biologiques, la plupart des gènes homéotiques Antennapedia de Drosophila ont été localisés chez les vertébrés et invertébrés tels les poulets, les humains, les souris et même les oursins de mer. Ces découvertes ont depuis poussé les chercheurs à émettre des hypothèses sur le rôle fonctionnel de ces gènes dans la détermination du plan corporel au cours des premières étapes de l’embryogénèse[8].
Gènes homéotiques chez les plantes
Les gènes contenant des boîtes homéotiques retrouvées chez les plantes peuvent être divisés en différentes familles et sous-familles selon la conservation des acides aminés et la présence de séquences additionnelles. La séquence de 60 acides aminés relativement bien conservée de l’homéodomaine suggère que chaque groupe de protéines contenant un homéodomaine aurait été formé par duplication d’un gène ancien et unique. On note que le premier gène homéotique observé chez les plantes est Knottedl (kn1)[9]. On peut regrouper ces gènes chez les plantes selon 11 classes majeures soit DDT, HD-ZIP (contenant 4 sous-classes : I à IV), LD, NDX, PHD, PINTOX, PLINC, SAWADEE, TALE (contenant 2 classes : KNOX et BEL), WOX[1].
Gènes homéotiques chez les animaux
On retrouve chez les animaux 16 classes majeures de gènes homéotiques soit les suivantes : ANTP, CERS, CUT (contenant 4 sous-classes : CMP, CUX, ONECUT et SATB), HNF, LIM, POU, PRD, PRD-LIKE, PROS, SIX/SO, ZF et la super-classe TALE regroupant les classes IRO, MEIS, MKX, PBC et TGIF). Au total, il y aurait chez la plupart des vertébrés environ 250 gènes homéotiques et 15-30 % des facteurs de transcriptions des animaux seraient des homéodomaines[1].
La conservation des gènes homéotiques à travers le règne animal
L’importance des gènes homéotiques dans le développement peut être démontrée par le fait que des mutations géniques sur ces gènes peuvent causer des cancers ou des anomalies de développement sévères[10],[11]. Parmi les gènes homéotiques les plus connus, on note les gènes HOX, paraHOX, Dlx, Pax, NK et Otx qui ont été très étudiés chez plusieurs animaux incluant la souris, le poisson zèbre, les nématodes et la mouche à fruits. C’est d’ailleurs la comparaison de ces gènes entre plusieurs espèces qui a mené à l’une des découvertes les plus intéressantes du XXe siècle en biologie, soit la conservation remarquable des boîtes homéotiques et de d’autres gènes développementaux entre des embranchements divergents d’animaux[12],[13]. Toutefois, ce ne sont pas tous les gènes homéotiques qui sont ancestraux et l’ampleur de leur conservation évolutive varie considérablement. Par exemple, une étude sur le locus (emplacement physique précis sur un chromosome) de toutes les boîtes homéotiques du génome humain a révélé la présence de six nouveaux gènes (ARGFX, DUXA, DUXB, DPRX, LEUTX et TPRX1) qui ne sont pas des gènes orthologues (gènes semblables retrouvés chez deux espèces distinctes) à ceux du génome de la souris, ni du génome des invertébrés[14],[15].
Gènes humains
On distingue quatre groupe de gènes HOX selon le chromosome où ils sont situés :
| Nom | chromosome | Gène |
| HOXA (ou HOX1) | Chromosome 7 | HOXA1, HOXA2, HOXA3, HOXA4, HOXA5, HOXA6, HOXA7, HOXA9, HOXA10, HOXA11, HOXA13 |
| HOXB | Chromosome 17 | HOXB1, HOXB2, HOXB3, HOXB4, HOXB5, HOXB6, HOXB7, HOXB8, HOXB9, HOXB13 |
| HOXC | Chromosome 12 | HOXC4, HOXC5, HOXC6, HOXC8, HOXC9, HOXC10, HOXC11, HOXC12, HOXC13 |
| HOXD | Chromosome 2 | HOXD1, HOXD3, HOXD4, HOXD8, HOXD9, HOXD10, HOXD11, HOXD12, HOXD13 |
La famille de gènes homéotiques DLX (distal-less homeobox) inclut DLX1, DLX2, DLX3, DLX4, DLX5, et DLX6. Ces gènes sont impliqués dans le développement du système nerveux et des membres.
Voici une liste d'autres protéines humaines qui contiennent l'homéodomaine :
- ADNP ; ALX3 ; ALX4 ; ARGFX ; ARX ;
- NKX3-2 (BAPX1) ; BARHL1; BARHL2 ; BARX1 ; BARX2 ;
- ALX1 (CART1) ; CDX1 ; CDX2 ; CDX4 ; CRX ; CUTL1; CUTL2 ;
- DBX1 ; DBX2 ; DLX1; DLX2 ; DLX3 ; DLX4 ; DLX5 ; DLX6 ; DMBX1 ; DPRX ; DRGX ; DUX1 ; DUX2 ; DUX3 ; DUX4 ; DUX4c ; DUX5 ; DUXA ;
- EMX1 ; EMX2 ; EN1 ; EN2 ; ESX1L ; EVX1; EVX2 ;
- GBX1 ; GBX2 ; GSC ; GSC2 ; GSX1 ; GSX2 ;
- HDX ; HESX1 ; HHEX ; HLX1; HMBOX1 ; HMX1 ; HMX2 ; HMX3 ; HNF1A ; HNF1B ; HOMEZ; HOPX ;
- IRX1 ; IRX2 ; IRX3 ; IRX4 ; IRX5 ; IRX6 ; ISL1 ; ISL2 ; ISX ;
- LASS2 ; LASS3; LASS4 ; LASS5 ; LASS6 ; LBX1 ; LBX2 ; LHX1 ; LHX2 ; LHX3 ; LHX4 ; LHX5 ; LHX6 ; LHX8 ; LHX9 ; LMX1A ; LMX1B ;
- MEIS1 ; MEIS2 ; MEIS3 ; MEOX1 ; MEOX2 ; MIXL1 ; MKX ; MNX1 ; MSX1 ; MSX2 ;
- NANOG ; NANOGP1 ; NANOGP8 ; NKX2-1 ; NKX2-2 ; NKX2-3 ; NKX2-4 ; NKX2-5 ; NKX2-8 ; NKX3-1 ; NKX3-2 ; NKX6-1 ; NKX6-2 ; NKX6-3 ; NOBOX ; NOTO ;
- ONECUT1 ; ONECUT2 ; ONECUT3 ; OTP ; OTX1 ; OTX2 ;
- PAX3 ; PAX4 ; PAX6 ; PAX7 ; PBX1 ; PBX2 ; PBX3 ; PBX4 ; PDX1 ; PHOX2A ; PHOX2B ; PITX1 ; PITX2 ; PITX3 ; PKNOX1 ; PKNOX2 ; POU1F1 ; POU2F1 ; POU2F2 ; POU2F3 ; POU3F1 ; POU3F2 ; POU3F3 ; POU3F4 ; POU4F1 ; POU4F2 ; POU4F3 ; POU5F1 ; POU5F1P1 ; POU5F1P4 ; POU5F2 ; POU6F1 ; POU6F2 ; PROP1 ; PRRX1 ; PRRX2 ;
- RAX ; RAX2 ; RHOXF1 ; RHOXF2 ;
- SATB1 ; SATB2 ; SEBOX ; SHOX ; SHOX2 ; SIX1 ; SIX2 ; SIX3 ; SIX4 ; SIX5 ; SIX6 ;
- TGIF1 ; TGIF2 ; TGIF2LX ; TGIF2LY ; TLX1 ; TLX2 ; TLX3 ; TSHZ1 ; TSHZ2 ; TSHZ3 ;
- UNCX ; VAX1 ; VAX2 ; VENTX ; VSX1 ; VSX2 ;
- ZEB1 ; ZEB2 ; ZFHX2 ; ZFHX3 ; ZFHX4 ; ZHX1.
Notes et références
- Bürglin, T. R., & Affolter, M. (2015) Homeodomain proteins: an update. Chromosoma, 1-25
- Li, G., & Holland, P. W. (2010). The origin and evolution of ARGFX homeobox loci in mammalian radiation. BMC evolutionary biology, 10(1), 182.
- Schofield, P. N. (1987). Patterns, puzzles and paradigms: the riddle of the homeobox. Trends in Neurosciences, 10(1), 3-6
- Young, R. A. (2011). Control of the embryonic stem cell state. Cell, 144(6), 940-954
- Scott, M. P., Tamkun, J. W., & Hartzell, G. W. (1989). The structure and function of the homeodomain. Biochimica y Biophysica Acta (BBA)-Reviews on Cancer, 989(1), 25-48
- McGinnis, W., Levine, M. S., Hafen, E., Kuroiwa, A., & Gehring, W. J. (1983). A conserved DNA sequence in homoeotic genes of the Drosophila Antennapedia and bithorax complexes. Nature, 308(5958), 428-433
- Gehring, W. J., & Hiromi, Y. (1986). Homeotic genes and the homeobox. Annual review of genetics, 20(1), 147-173.
- Jacobson, B. (2012) Homeobox Genes and the Homeobox. Arizona State University. Embryo Project Encyclopedia.
- Chan, R. L., Gago, G. M., Palena, C. M., & Gonzalez, D. H. (1998). Homeoboxes in plant development. Biochimica et Biophysica Acta (BBA)-Gene Structure and Expression, 1442(1), 1-19.
- Nunes, F. D., Almeida, F. C. S. D., Tucci, R., & Sousa, S. C. O. M. D. (2003). Homeobox genes: a molecular link between development and cancer. Pesquisa Odontologica Brasileira, 17(1), 94-98.
- Del Bene, F., & Wittbrodt, J. (2005). Cell cycle control by homeobox genes in development and disease. Seminars in cell & developmental biology,16(3), 449-460.
- Kappen, C., & Ruddle, F. H. (1993). Evolution of a regulatory gene family: HOM/HOX genes. Current opinion in genetics & development, 3(6), 931-938.
- Manak, J. R., & Scott, M. P. (1994). A class act: conservation of homeodomain protein functions. Development, 1994(Supplement), 61-77.
- Booth, H. A. F., & Holland, P. W. (2007). Annotation, nomenclature and evolution of four novel homeobox genes expressed in the human germ line. Gene, 387(1), 7-14.
- Holland, P. W., Booth, H. A., & Bruford, E. A. (2007). Classification and nomenclature of all human homeobox genes. BMC biology, 5(1), 47.