Macrohaplogrupo L (ADNmt)
En genética humana, el macrohaplogrupo L es un paragrupo que equivale a la ascendencia mitocondrial africana de toda la humanidad, se distribuye ampliamente en el África Subsahariana y está relacionado con el origen de los humanos modernos por línea materna a partir de la Eva mitocondrial, por lo cual tendría su origen en África Oriental hace aproximadamente 190 000 años.[1] L es un grupo parafilético de haplogrupos (paragrupo) con relación a los macrohaplogrupos M y N, los cuales representan la expansión humana fuera de África.
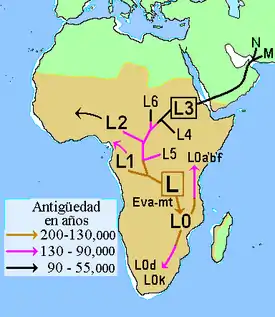
Los haplogrupos L son predominantes en el África negra, con frecuencias del 96-100 %, salvo en las áreas de difusión de las lenguas afroasiáticas, donde disminuye sensiblemente. Menores frecuencias se encuentran en África del Norte, Arabia y Medio Oriente en general; y en Europa especialmente al Sur. En América, L predomina entre los afroamericanos.
Clados
Los clados de L son L0, L1, L2, L3, L4, L5 y L6 relacionados filogenéticamente del siguiente modo:[2]
| Macrohaplogrupo L |
| |||||||||||||||||||||||||||||||||||||||||||||
Distribución por clados

- Macrohaplogrupo L (Eva mitocondrial)
- Haplogrupo L0: Típico de los pueblos khoisán y extendido en toda África.
- L0d
- L0a'b'f'k
- L0k
- L0a'b'f
- L0f
- L0a'b
- L1-6 o L1'2'3'4'5'6 (146, 182, 4312, 10664, 10915, 11914, 13276, 16230)
- Haplogrupo L1: Con el predominio más importante en los pigmeos binga y muy extendido en África Central y Occidental.
- L1b
- L1c
- L2-6 o L2'3'4'5'6
- Haplogrupo L5: Se presenta irregularmente hacia el este de África.
- L2'3'4'6
- Haplogrupo L2: Común en toda África y extendido en afroamericanos y parte de Medio Oriente.
- L2e
- L2a-d
- L2a
- L2b'c
- L2d
- L3'4'6
- Haplogrupo L6: En Yemen y Sudán.
- L3'4
- Haplogrupo L4: Predominante entre los hadza y sandawe; común en otras etnias de Tanzania.
- L4a (o L7)
- L4b
- Haplogrupo L3: Muy extendido en toda África, mayoritario en nigero-congoleños y es común en los países árabes.
- Haplogrupo L4: Predominante entre los hadza y sandawe; común en otras etnias de Tanzania.
- Haplogrupo L2: Común en toda África y extendido en afroamericanos y parte de Medio Oriente.
- Haplogrupo L1: Con el predominio más importante en los pigmeos binga y muy extendido en África Central y Occidental.
- Haplogrupo L0: Típico de los pueblos khoisán y extendido en toda África.
Véase también
| Eva mitocondrial (L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1–6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | A | I | O | R | S | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | R2'JT | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
Referencias
- P. Soares et al. 2009, Supplemental Data: An Improved Human Mitochondrial Molecular Clock. AJHG, Volume 84
- van Oven M, Kayser M. 2009. PhyloTree.org - mtDNA subtree L Archivado el 27 de julio de 2011 en Wayback Machine. doi:10.1002/humu.20921
- Alexandra Rosa et al. 2004, MtDNA Profile of West Africa Guineans: Towards a Better Understanding of the Senegambia Region.
- K. Abu-Amero et al. 2008, Mitochondrial DNA structure in the Arabian Peninsula. BMC Evolutionary Biology 2008, 8:45
- T. Kivisild et al. 2004, Ethiopian Mitochondrial DNA Heritage: Tracking Gene Flow Across and Around the Gate of Tears. AJHG 75, 5, 752-770
- Sadie Anderson 2006, Phylogenetic and phylogeographic analysis of African mitochondrial DNA variation.
- Sarah A. Tishkoff et al. 2007, History of Click-Speaking Populations of Africa Inferred from mtDNA and Y Chromosome Genetic Variation. Molecular Biology and Evolution 2007 24(10):2180-2195
- Antonio Salas et al. 2002, The Making of the African mtDNA Landscape. Am J Hum Genet. 2002 November; 71(5): 1082–1111.
- Yu-Sheng Chen et al. 1999, mtDNA Variation in the South African Kung and Khwe—and Their Genetic Relationships to Other African Populations. Am J Hum Genet. 2000 April; 66(4): 1362–1383.
- Quintana, Lluis et al 2003, MtDNA diversity in Central Africa: from hunter-gathering to agriculturalism
Enlaces externos
- Árbol filogenético de L* PhyloTree.org de van Oven & Kayser M. 2009.
- Mitochondrial DNA Site de Ian Logan