Haplogrupos del cromosoma Y humano
En genética poblacional, los haplogrupos del cromosoma Y humano (ADN-Y) están determinados por las diferencias que la evolución molecular produce en las secuencias de ADN del cromosoma Y humano a lo largo de milenios; lo que permite trazar la línea de descendencia patrilineal humana, es decir, la herencia genética transmisible exclusivamente de padre a hijo varón.
Las mutaciones identificadas constituyen los marcadores que pueden caracterizar el haplotipo de un hombre; un conjunto grande de haplotipos relacionados forma un haplogrupo, y estos a su vez han recibido una nomenclatura que desde 2002 los nombra con letras desde la A hasta la T. A las subdivisiones se le añaden números y letras minúsculas sucesivamente de acuerdo con la nomenclatura[1] definida por el Y Chromosome Consortium.
Los principales haplogrupos del cromosoma Y para Homo sapiens y su relación filogenética resumida se muestra en el siguiente cladograma:[2]
|
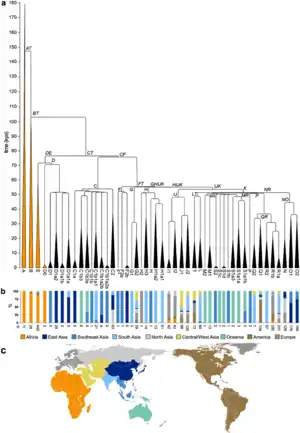
Dispersión y distribución
La distribución de los haplogrupos ancestrales refuerzan la teoría del origen de los humanos modernos en el África subsahariana y se puede trazar en forma aproximada las migraciones humanas prehistóricas a partir de África y la sucesiva colonización del resto del mundo, lo que puede resumirse en el siguiente mapa:

Haplogrupos en otras especies humanas
Restos humanos primitivos encontrados en Europa y Siberia, cuyo clima ha permitido la preservación de parte de su ADN, ha sido analizada genéticamente. El descubrimiento del hombre de Sidrón de hace 45000 años, brindó la oportunidad de realizar el análisis genético cromosómico a un individuo neandertal. Su estudio permitió concluir que la divergencia entre neandertales y humanos modernos habría ocurrido hace unos 590 mil años aproximadamente. En las poblaciones humanas modernas, no se ha encontrado haplogrupos neandertales, ni cromosómicos Y ni mitocondriales, por lo que es probable que estén extintos.[5]
El cromosoma Y en humanos arcaicos ha revelado resultados similares al análisis efectuado en el ADN mitocondrial, por lo que se han encontrado haplotipos que se relacionan con el de los humanos modernos del siguiente modo:[6]
|
La divergencia con los denisovanos habría ocurrido hace unos 700 mil años.[7]
Ascendencia africana temprana
Los linajes más antiguos se encuentran en África, por lo que el análisis genético del cromosoma Y refuerza la teoría del origen de los humanos modernos en África subsahariana.
Al ancestro común más reciente se le denomina Adán cromosómico, es de origen africano e inicialmente se le calculó entre 60 y 90 mil años de antigüedad.[8] Sin embargo, un estudio posterior (2011),[9] descubre más ramificaciones en el árbol filogenético dándole al Adán cromosómico 140.000 años de antigüedad, y más recientemente (2013), el descubrimiento del haplogrupo perdido A00 entre algunos pocos afroamericanos extendió su antigüedad a más de 300.000 años.[10]
Se considera que son africanos el ancestral paragrupo A y los haplogrupos B y E, los cuales se distribuyen ampliamente por todo el África subsahariana, dispersándose en épocas más recientes por otros continentes a través de migraciones y otros desplazamientos. Las relaciones filogenéticas entre los principales clados dentro y fuera de África es la siguiente:[2]
|
Fuera de África |
Paragrupo A
Los clados con las raíces más profundas pertenecen al paragrupo A, siendo los más antiguos A00, A0 y A1a en los humanos modernos. Estos se encuentran actualmente dispersos en muy pequeños porcentajes en especial en África Occidental y central, y está relacionado con los grandes clados que descienden del Adán cromosómico, por lo que tiene gran diversidad y una antigüedad aproximada de 300.000 años. Según ISOGG-2014[11] y PhyloTree,.[12] Las mayores frecuencias de A corresponden al haplogrupo A1b1 (A2'3 o A-L419), el cual se encuentra principalmente entre los pueblos joisán como los bosquimanos !khung (África austral) y los pueblos nilóticos (sursudaneses).
Haplogrupos B y E
El haplogrupo B se encuentra muy disperso en todo el África subsahariana, pero sus mayores frecuencias se hallan entre los pigmeos (binga y mbuti) y los nativos hadza. El haplogrupo E es el más frecuente en casi todo África, aparece en África oriental hace más de 50 mil años, se dispersó por todo el continente y alcanza las frecuencias más altas en el África occidental sahariana y subsahariana (más de 80%) y Etiopía (68%). El subclado E-M78, aparecido hace unos 18.600 años, se esparce por todo el Mediterráneo, por lo que es común en Europa y Cercano oriente. En Europa alcanza la presencia más alta en la comunidad étnica de los pasiegos con 41%.
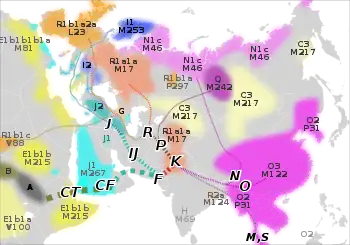
Expansión fuera de África
La evidencia filogeográfica del cromosoma-Y, nos permite deducir que se produjeron una o dos migraciones iniciales hace 60.000 años.[13] Estas migraciones se realizaron probablemente a través del estrecho Bab el-Mandeb desde el Cuerno de África en ruta costera hacia el Sur de Asia. Son dos los linajes que representan estas migraciones: D de donde desciende su principal clado el haplogrupo D1, y CF, de donde descienden macrohaplogrupos importantes como C, F y K.
El lugar más probable de origen de un haplogrupo está donde se distribuyen sus clados más antiguos, por lo que es factible que D1, C y F se originasen en la península del Indostán o en algún punto de la ruta costera desde África hacia el Sur de Asia. La siguiente tabla indica los linajes más antiguos descendientes de los macrohaplogrupos fuera de África, cuyo origen y subsecuente dispersión parece relacionarse con una temprana colonización del Subcontinente Indio.[4]
| Macrohaplogrupos | Eurasia Occidental | Indostán | Extremo Oriente | Sahul | América |
|---|---|---|---|---|---|
| D | - | D1 | D1 | - | - |
| C | C1a2 | C1b1 | C2, C1a1 | C1b2 | C2-P39 |
| F (exc. K) | G, IJ | H, F1, F3 | F2, F4 | - | - |
| K | R1, LT | LT, QR | NO, Q-M120 | MS | Q-L54 |
Haplogrupo D
Parte de estas primeras migraciones corresponden al haplogrupo D, el cual se encuentra casi exclusivamente en Asia y tendría relación con poblaciones antiguas como los negritos del Sudeste de Asia y los primeros pobladores del Asia Oriental, siendo en la actualidad especialmente frecuente en el Japón y el Tíbet. Son sus principales subclados (el marcador más representativa va entre paréntesis):
| D (CTS3946) |
| ||||||||||||||||||||||||
Grupos descendientes de CF
La mayor distribución fuera de África corresponde a los descendientes de CF (única mutación P143), el cual está relacionado con las primeras migraciones a regiones distantes como Australia, Europa y el resto del mundo, de manera que pueden encontrarse inclusive entre amerindios.
Haplogrupo C
El haplogrupo C (M130=RPS4Y, M216) tiene gran dispersión en todo Eurasia Oriental y Oceanía. Sus clados principales y su distribución más importante es del siguiente modo:
| C (M130) |
| ||||||||||||||||||||||||||||||||||||
Haplogrupo F
El haplogrupo F (M89) probablemente se originó en el Subcontinente indio hace unos 48.000 años. Los haplogrupos descendientes se encuentran en el 90% de la actual población mundial, pero casi exclusivamente fuera del África subsahariana. Son sus principales clados:
- Paragrupo F: los haplogrupos F1 al F4 y otros aún no definidos dentro de F* se encuentran en bajas frecuencias dispersos en Eurasia, pero principalmente en grupos tribales de la India.
- G (M201): característico de Eurasia Occidental. Se originó en el Cercano Oriente o Sur de Asia hace 30 mil años.
- H (M69): se origina en India hace 30 o 40 mil años y permaneció allí, siendo actualmente el más característico del Subcontinente indio. Se difundió en épocas históricas debido principalmente a la migración de los gitanos.
- IJK (L15)
- IJ (M429): tiene unos 45 mil años y podría relacionarse con el hombre de Cro-Magnon en Europa y Cercano Oriente.
- K (M9): se difundió por toda Eurasia y desde allí a Oceanía y América.
Haplogrupo K
Al haplogrupo K (M9) se le considera un "macrohaplogrupo" debido a su gran extensión filogeográfica. Su origen es impreciso, pero probablemente se originó al Sur de Asia hace unos 47.000 años. Sus principales ramas se distribuyen del siguiente modo:
- K* Paragrupo poco estudiado. Es lo que se presenta más frecuentemente en Micronesia y en algunas tribus filipinas. Es importante en nativos de Oceanía.
- LT (L298)
- L (M11): Encontrado principalmente en el sur de Asia.
- T (M184): Disperso en Eurasia Occidental, extendiéndose hacia la India y el Cuerno de África.
- K2 o K-M526
- K2c, K2d, K2e Son escasos.
- MS (P256) Predominante en Melanesia y extendido en Oceanía e Insulindia
- NO (M214): Apareció hace 35-40 mil años en el Extremo Oriente.
- N (M231): Se difundió en el Oriente, en toda Siberia y a Occidente hacia los Urales.
- O (M175): Predomina en Asia oriental y Sudeste de Asia.
- P (M45)
- Q (M242): Originario del Asia del Sur o Central, migró hacia América donde domina la variante Q-M3 entre los amerindios.
- R (M207): Predominante en Europa y Subcontinente indio.
- R1 (M173)
- R1a (M420): Se le ha relacionado con la expansión de las lenguas indoeuropeas. Se encuentra principalmente en Asia central y occidental, India, y entre los pueblos eslavos de Europa oriental.
- R1b (M343): Es muy común entre la población europea y más frecuente en las islas británicas y España; la variedad R1b-S116/P312 alcanza frecuencias importantes en Vasconia (País Vasco español y francés).
- R2 (M479): En India, Irán y Asia central.
- R1 (M173)
Nomenclatura
Desde 1999 se intentó designar a cada haplogrupo con varias denominaciones, hasta que llegó la propuesta histórica del Y Chromosome Consortium de 2002, la cual tuvo gran aceptación. Sin embargo, aún se está lejos de tener una nomenclatura estable, pues es susceptible a los cambios producidos por el descubrimiento de nuevos haplotipos. La siguiente tabla de conversión resume la nomenclatura dada a los principales haplogrupos:
| Haplogrupo | Su 1999[14] |
Jobling 2000[15] |
Underhill 2000[16] |
Semino 2000[17] |
Hammer 2001[18] |
Karafet 2001[19] |
Capelli 2001[20] |
YCC 2002[21] |
ISOGG 2010[22] |
ISOGG 2012[23] | VanOven 2014[24] | ISOGG 2020[25] |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A00 (AF6) | – | – | – | – | – | – | – | – | – | – | A00 | A00 |
| A0 (V148) | – | – | – | – | – | – | – | – | – | A0 | A0 | A0 |
| A1a (M31) | H1 | 7 | I | – | 1A | 1 | A | A1 | A1a | A1a | A1 | A1a |
| A1b-M6 | H1 | 27 | I | – | 2 | 3, 4 | A | A2 | A2 | A1b1a1a | A2-M6 | A1b1a1a |
| A1b-M51 | H1 | 7 | I | – | 1A | 1 | A | A3b1 | A3b1 | A1b1b2a | A3-M51 | A1b1b2a |
| A1b-M13 | H1 | 7 | I | Eu1 | 1A | 2 | A | A3b2 | A3b2 | A1b1b2b | A3-M13 | A1b1b2b |
| B (M60) | H1 | 2, 6 | II | – | 1B | 5 a 10 | B | B | B | B | B | B |
| C (M216) | H1 | 10 | V | Eu6 | 1F | 16 a 19 | C | C | C | C | C | C |
| C-M38 | H1 | 10 | V | Eu6 | 1F | 16,18 | C | C2 | C2 | C2 | C1b-M38 | C1b2a |
| C-M347 | H1 | 10 | V | Eu6 | 1F | 16 | C | C* | C4 | C4 | C1b-M347 | C1b2b |
| C-M217 | H1 | 10 | V | Eu6 | 1F | 17 | C | C3 | C3 | C3 | C2 | C2 |
| D-M174 | H2-3 | 4 | IV | Eu5 | 3G | 11, 12 | B | D | D | D | D | D1 |
| D-M55/M64 | H2 | 4 | IV | Eu5 | 3G | 11 | B | D2 | D2 | D2 | D2 | D1a2a |
| E (M96) | H2 | – | III | Eu2-4 | – | 13 a 15 | B | E | E | E | E | E |
| E-M33 (M132) | H2 | 21 | III | Eu3 | 3A | 13 | B | E1 | E1a | E1a | E1a | E1a |
| E-M2 (P1) | H2 | 8 | III | Eu2 | 5 | 15 | B | E3a | E1b1a | E1b1a1 | E1b1a-M2 | E1b1a1 |
| E-M35 | H2 | 25 | III | Eu4 | 4 | 14 | B | E3b | E1b1b1 | E1b1b1 | E1b1b-M35 | E1b1b1 |
| E-M75 | H2 | 21 | III | Eu3 | 3A | 13 | B | E2 | E2 | E2 | E2 | E2 |
| F (M89) | H4-17 | – | VI a X | Eu7-22 | – | 20 a 45 | – | F | F | F | F | F |
| G (M201) | H4 | 2 | VI | Eu11 | – | – | B | G | G | G | G | G |
| G-L1324 | H4 | 2 | VI | Eu11 | 1R | 20 | B | G1 (P20) | G1a | G1a1 | – | G1a1a1a |
| G-P15 | H4 | 2 | VI | Eu11 | 1Ha,1Hb | 22 | B | G2 | G2a | G2a | G2a | G2a |
| H-M69 | H4 | – | VI | – | 1R | 20 | B | H | H | H | H1 | H1a |
| H-M52 | H4 | 35 | VI | Eu12 | 1R | 20 | B | H | H1 | H1 | H1-M52 | H1a1 |
| H-Apt | H4 | 15 | VI | Eu10 | 1R | 20 | B | F1 | H2 | H2 | H1-Apt | H1a2a |
| I (M170) | H4 | 2 | VI | Eu7 | 1R | 21 | B | I | I | I | I | I |
| J (12f2.1) | H4 | 9 | VI | Eu9-10 | Med | 23-24 | B | J | J | J | J | J |
| J1-M267 | H4 | 9 | VI | Eu10 | Med | 23 | B | J1 | J1 | J1 | J1 | J1 |
| J2-M172 | H4 | 9 | VI | Eu9 | Med | 24 | B | J2 | J2 | J2 | J2 | J2 |
| K (M9) | H5-17 | – | VII a X | Eu13-22 | – | 25 a 45 | – | K | K | K | KLT | K |
| K-M256 | – | – | – | – | – | – | – | – | MNOPS | K(xLT) | K | K2 |
| L (M20) | H5 | 28 | VIII | Eu17 | 1U | 27 | F | L | L | L | L | L |
| M (P256, SK1828) | – | – | VIII | Eu16 | – | – | – | – | M | M | M1 | M |
| M-M4 | H17 | 24 | VIII | Eu16 | 1U | 37 a 39 | E | M | M1 | M1 | M1-M5 | M1 |
| M-SRY9138 | H5 | 23 | VIII | Eu16 | 1E | 25 | F | K1 | M2a | M2a | M1-M177 | M2a |
| N (M231) | H5 | 12, 16 | VIII | Eu13-14, Eu16 | 1U, 1I | 25, 26 | F | N | N | N | N | N |
| N-Tat | H5 | 16 | VIII | Eu13-14 | 1I | 26 | F | N3 | N1c | N1c1 | N-M46 | N1a1 |
| O (M175) | H5-12 | – | VII | Eu16 | 1U | 29 a 36 | - | O | O | O | O | O |
| O1-MSY2 (M116) | H9-10 | 26 | VII | Eu16 | 1U | 32 | H | O1 | O1 | O1 | O1 | O1a |
| O2-P31 | H5, H11-H12 | 5, 20, 26 | VII | Eu16 | 1U | 33 a 36 | G, I | O2 | O2 | O2 | O2 | O1b |
| O3-M122 | H6-8 | 26, 3 | VII | Eu16 | 1U | 29 a 31 | L | O3 | O3 | O3 | O3 | O2 |
| P-M45 | H13-16 | – | IX-X | Eu18-22 | – | 40 a 45 | D | P | P | P | QR | P1 |
| Q (M242) | – | – | X | – | – | – | D | - | Q | Q | Q | Q |
| Q-P36 (xQ-M3) | H3, H14 | 1 | X | Eu20, Eu22 | 1C | 40 | D | Q | Q1 | Q1 | Q1 | – |
| Q-M3 | H15 | 18 | X | Eu22 | 1G | 41 | D | Q3 | Q1a3a1 | Q1a3a1 | Q1a2-M3 | Q1b1a1a |
| R (M207) | – | – | – | – | – | – | D | R | R | R | R | R |
| R1a-M17 | H16 | 3 | IX | Eu19 | 1D | 45 | D | R1a1 | R1a1a | R1a1a | R1a-M17 | R1a1a |
| R1b-P25/L754 | H14 | 1 | IX | Eu18 | 1L | 44 | D | R1b | R1b1 | R1b1 | R1b1 | R1b1 |
| R2-M124 | H14 | 1 | X | Eu21 | 1C | 40 | D | P1 | R2a | R2a | R2a | R2a |
| S-M230 | – | – | – | – | – | – | – | K* | S | S | M2-M230 | S1a1b |
| S-P60/P308 | – | – | – | – | – | – | – | K* | K2 | K1 | M2-P60 | S1a1a1 |
| T-M70 | H5 | 26 | VIII | Eu15 | 1U | 25 | F | K2 | T | T1a | T-M70 | T1a |
Véase también
| Adán cromosómico | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| C1 | C2 | G | H | IJK | ||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| L | T | MS | P | NO | ||||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||
Referencias
- The Y Chromosome Consortium 2002, A Nomenclature System for the Tree of Human Y-Chromosomal Binary Haplogroups Genome Res. 2002. 12: 339-348 doi: 10.1101/gr.217602
- Y-DNA Haplogroup Tree ISOGG, última revisión: Ago 2013
- Hallast, P., Agdzhoyan, A., Balanovsky, O. et al. A Southeast Asian origin for present-day non-African human Y chromosomes. Hum Genet 140, 299–307 (2021). https://doi.org/10.1007/s00439-020-02204-9
- Kivisild et al 2003, The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste Populations
- Mendez, F. L., Poznik, G. D., Castellano, S., & Bustamante, C. D. (2016). The Divergence of Neandertal and Modern Human Y Chromosomes. American journal of human genetics, 98(4), 728–734.
- Y-DNA Haplogroup A and its Subclades - 2019-2020 ISOGG (International Society of Genetic Genealogy)
- Martin Petr, Mateja Hajdinjak, Qiaomei Fu, Elena Essel, Hélène Rougie et al. 2020, The evolutionary history of Neanderthal and Denisovan Y chromosomes. Science 25 Sep 2020: Vol. 369, Issue 6511, pp. 1653-1656 DOI: 10.1126/science.abb6460
- Mitochondrial Eve and Y-chromosomal Adam The Genetic Genealogist
- Cruciani, Fulvio et al 2011, A Revised Root for the Human Y Chromosomal Phylogenetic Tree: The Origin of Patrilineal Diversity in Africa
- The New Root – Haplogroup A00 DNAeXplained – Genetic Genealogy 2012
- ISOGG: Y-DNA Haplogroup A and its Subclades (última revisión: May 2014)
- Oven M. et al 2014. Minimal reference phylogeny for the human Y chromosome (version 9-Apr-2014)
- Chiaroni J. et al 2009, Y chromosome diversity, human expansion, drift, and cultural evolution
- Su, B., Xiao, J. et al 1999, Y-Chromosome evidence for a northward migration of modern humans into Eastern Asia during the last Ice Age. Am. J. Hum. Genet. 65: 1718–1724.
- Jobling, Mark A.; Tyler-Smith, Chris (2000). "New uses for new haplotypes". Trends in Genetics 16 (8): 356–62. doi:10.1016/S0168-9525(00)02057-6. PMID 10904265.
- Underhill, P.A., et al. 2000, Y chromosome sequence variation and the history of human populations. Nat. Genet. 26: 358–361.
- Semino, O. et al 2000, The genetic legacy of paleolithic homo sapiens in extant Europeans: A Y chromosome perspective. Science 290: 1155–1159.
- Hammer, M.F. et al 2001, Hierarchical patterns of global human y-chromosome diversity. Mol. Biol. Evol. 18: 1189–1203.
- Karafet, T. et al 2001, Paternal population history of East Asia: Sources, patterns, and microevolutionary rocesses. Am. J. Hum. Genet. 69: 615–628.
- Capelli et al. 2001. Capelli, C; Wilson, J; Richards, M; Stumpf, M; Gratrix, F; Oppenheimer, S; Underhill, P; Pascali, V et al. (2001). «A Predominantly Indigenous Paternal Heritage for the Austronesian-Speaking Peoples of Insular Southeast Asia and Oceania». The American Journal of Human Genetics 68: 432-43. PMC 1235276. PMID 11170891. doi:10.1086/318205.
- The Y Chromosome Consortium 2002, A Nomenclature System for the Tree of Human Y-Chromosomal Binary Haplogroups Genome Res. 2002 12: 339-348
- ISOGG 2010 Y-DNA Haplogroup Tree
- ISOGG 2012 Y-DNA Haplogroup Tree 2012
- van Oven M. et al 2014. Seeing the wood for the trees: a minimal reference phylogeny for the human Y chromosome. Hum Mutat. 2014 Feb;35(2):187-91. doi: 10.1002/humu.22468. Epub 2013 Nov 14. Full Y
- Y-DNA Haplogroup Tree 2019-2020 International Society of Genetic Genealogy
- Jobling, Mark A. y Chris Tyler-Smith 2003 El Cromosoma Y humano: un marcador evolutivo que llega a su mayoría de edad. "The human Y chromosome: an evolutionary marker comes of age"; Nature Reviews Genetics 4 (8): 598-612.
- A Nomenclature system for the Tree of Human Y-Chromosomal Haplogroups, Genome.org
Enlaces externos
- Árbol ISOGG de haplogrupos de ADN-Y de la International Society of Genetic Genealogy
- Yfull tree Experimental rev. 2020
- Árbol FTDNA de haplogrupos de ADN-Y de Family Tree DNA
- Mapa de haplogrupos J.D. Mc Donald 2005
- Origins, age, spread and ethnic association of European haplogroups and subclades. de Eupedia.com
- Overview of Y-DNA testing de Genebase
- DNA Consulting's Conversion Chart for Male Haplogroups Nomenclaturas ADN-Y (PDF) 2008
- YCC Stammbaum 2008 Filogenia Karafet et al
- Minimal reference phylogeny for the human Y chromosome (version 2014)
 Wikimedia Commons alberga una categoría multimedia sobre Haplogrupos del cromosoma Y humano.
Wikimedia Commons alberga una categoría multimedia sobre Haplogrupos del cromosoma Y humano.
- Las frecuencias de haplogrupos del cromosoma Y por regiones, pueden encontrarse en las siguientes listas (en inglés):