Histona H3
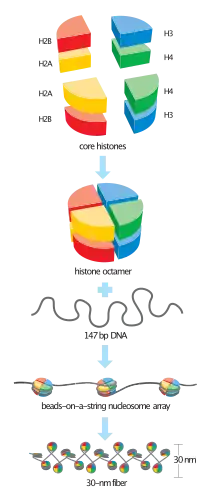
La histona H3 es una de las cinco familias principales de proteínas histonas, involucradas en mantener y regular la estructura de la cromatina en células eucariotas.[1][2] Se compone de un dominio globular principal y una cola larga N-terminal. H3 está involucrada en la estructura de "cuentas en cuerda" de los nucleosomas con el ADN. Las histonas son proteínas que experimentan frecuentemente modificaciones postraduccionales, siendo H3 la que sufre más modificaciones de las cinco histonas. El término Histona H3 es deliberadamente ambiguo, ya que no permite distinguir entre las variantes de secuencia o estados de modificación postraduccional. H3 es una proteína importante en el campo emergente de la epigenética, ya que se cree que sus variantes de secuencias y estados variables de modificación cumplen un papel en la regulación génica a largo plazo.
| Histona H3, familia 3B (H3.3B) | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | Buscar ortólogos: PDBe, RCSB | |||
| Identificadores | ||||
| Símbolo | H3F3B (HGNC: 4765) | |||
| Identificadores externos | ||||
| Locus | Cr. 17 q25 | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
Las otras familias de histonas son: H1, H2A, H2B y H4.
| Histona H3, familia 3A (H3.3A) | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | Buscar ortólogos: PDBe, RCSB | |||
| Identificadores | ||||
| Símbolos | H3F3A (HGNC: 4764) H3F3 | |||
| Identificadores externos | ||||
| Locus | Cr. 1 q41 | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
Epigenética y modificaciones postraduccionales
La cola N-terminal de H3 sobresale del nucleosoma central y es susceptible a modificaciones postraduccionales que influyen en procesos celulares. Estas modificaciones incluyen la unión covalente de grupos metilo o acetilo a aminoácidos lisina y arginina y la fosforilación de serinas o treoninas. La di- y trimetilación de la lisina en posición 9 se asocia con la represión de la expresión génica y la heterocromatina (ver H3K9me2 y H3K9me3), mientras que la monometilación de la lisina K4 (ver H3K4me1), se asocia con genes activos.[3][4] La acetilación de H3 en diferentes posiciones de lisinas en la cola están regulada por enzimas histona acetiltransferasas (HATs). La acetilación de la lisina K14 se ha identificado comúnmente en genes en transcripción activa en ARN (ver H3K14ac).
Variantes de secuencia
Se han identificado variantes de la histona canónica H3 en células de mamíferos. Estas se denominan Histona H3.1, H3.2, H3.3 H3.4 (H3T), H3.5, H3.X y H3.Y. Estas tienen secuencias altamente conservadas, diferenciándose entre sí tan solo en unos pocos aminoácidos.[5][6] Se ha comprobado que H3.3 cumple un rol importante en el mantenimiento de la integridad del genoma en el desarrollo de mamíferos.[7] Las variantes de histonas de diferentes organismos, así como su clasificación y características específicas se pueden encontrar en la base de datos HistoneDB - with Variants.
Genética
La histona H3 está codificada por diferentes genes en el genoma humano, incluyendo los siguientes:
- H3.1: HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J
- H3.2: HIST2H3A, HIST2H3C, HIST2H3D
- H3.3: H3F3A, H3F3B
Véase también
Referencias
- Reche, Pedro A.; Keskin, Derin B.; Hussey, Rebecca E.; Ancuta, Petronela; Gabuzda, Dana; Reinherz, Ellis L. (2006). «Recognition and classification of histones using support vector machine». Journal of Computational Biology 13 (1): 102-12. PMID 16472024. doi:10.1089/cmb.2006.13.102.
- Hartl, Daniel L.; Freifelder, David; Snyder, Leon A. (1988). Basic Genetics. Boston: Jones and Bartlett Publishers. ISBN 978-0-86720-090-4. Consultado el 23 de octubre de 2022.
- Rosenfeld, Jeffrey A.; Wang, Zhibin; Schones, Dustin E.; Zhao, Keji; DeSalle, Rob; Zhang, Michael Q. (Marzo de 2009). «Determination of enriched histone modifications in non-genic portions of the human genome». BMC Genomics 10: 143. PMC 2667539. PMID 19335899. doi:10.1186/1471-2164-10-143. Consultado el 23 de octubre de 2022.
- Lachner, Monika; O'Carroll, Dónal; Mechtler, Karl; Jenuwein, Thomas (Marzo de 2001). «Methylation of histone H3 lysine 9 creates a binding site for HP1 proteins». Nature 410 (6824): 116-20. Bibcode:2001Natur.410..116L. PMID 11242053. doi:10.1038/35065132.
- Mazrluff, William F.; Gongidi, Preetman; Woods, Keith R.; Jin, Jianping; Maltais, Lois J. (Noviembre de 2002). «The human and mouse replication-dependent histone genes». Genomics 80 (5): 487-98. PMID 12408966. doi:10.1016/S0888-7543(02)96850-3. Consultado el 23 de octubre de 2022.
- Hake, Sandra B.; García, Benjamin A.; Duncan, Elizabeth M.; Kauer, Monika; Dellaire, Graham; Shabanowitz, Jeffrey; Bazett-Jones, David P.; Allis, C. Davis et al. (Enero de 2006). «Expression patterns and post-translational modifications associated with mammalian histone H3 variants». The Journal of Biological Chemistry 281 (1): 559-68. PMID 16267050. doi:10.1074/jbc.M509266200. Consultado el 23 de octubre de 2022.
- Jang, Chuan-Wei; Shibata, Yoichiro; Starmer, Joshua; Yee, Della; Magnuson, Terry (Julio de 2015). «Histone H3.3 maintains genome integrity during mammalian development». Genes & Development 29 (13): 1377-92. PMC 4511213. PMID 26159997. doi:10.1101/gad.264150.115.