Histona H4
La histona H4 es una de las cinco familias principales de proteínas histonas, involucradas en el mantenimiento y regulación de la estructura de la cromatina en células eucariotas. Se compone de un dominio globular central y una cola larga N-terminal. H4 está involucrada en la estructura en forma de "cuentas en cuerda" de los nucleosomas y el ADN. Las histonas experimentan frecuentemente modificaciones postraduccionales. Las modificaciones incluyen la unión covalente de grupos acetilo y metilo en la cola N-terminal. Estas modificaciones pueden alterar la expresión de genes asociados con el octámero de histonas. [1] [2] H4 es una proteína importante para la estructura y función de la cromatina, ya que se cree que sus variantes de secuencia y estados variables de modificación juegan un papel en la regulación de genes a largo plazo.
| Histona H4 | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | Buscar ortólogos: PDBe, RCSB | |||
| Identificadores | ||||
| Símbolo | H4F3 (HGNC: 4780) | |||
| Identificadores externos | ||||
| Ortólogos | ||||
| Especies |
| |||
| UniProt |
| |||
| RefSeq (proteína) NCBI |
| |||
Genética
La histona H4 está codificada por múltiples genes en diferentes loci, incluidos: HIST1H4A, HIST1H4B, HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4G, HIST1H4H, HIST1H4I, HIST1H4J, HIST1H4K, HIST1H4L, HIST2H4A, HISTH4B .
Evolución
Las histonas son de las proteínas más conservadas evolutivamente en organismos eucariotas. Por ejemplo, la secuencia de aminoácidos de H4 de un guisante y una vaca difieren en solo 2 de sus 102 aminoácidos. Esta conservación sugiere que las funciones biológicas que cumplen involucran a casi todos de sus aminoácidos, siendo cualquier cambio dañino para la células. La mayoría de cambios en secuencias de histonas son letales; los pocos que no son letales causan cambios en el patrón de expresión génica así como otras anomalías. [3]
Estructura
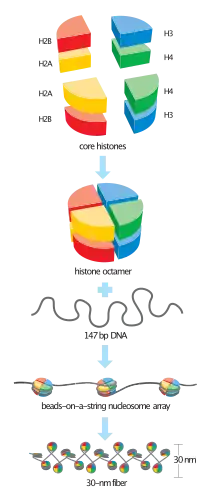
La histona H4 tiene una secuencia de entre 102 y 135 aminoácidos, con un motivo estructural, conocido como el plegamiento de histonas y formado por tres alfa-helices conectados por dos bucles. Las histonas H3 y H4 se unen para formar el heterodímero H3-H4, que a su vez se combinan en pareja para formar un tetrámero. Este tetrámero se combina con heterodímeros H2A-H2B para formar un octámero de histonas. [3]
Variantes de secuencia
La histona H4 es unas de las proteínas de evolución más lenta. Existen genes que se expresan de manera constitutiva a lo largo del ciclo celular y que expresan proteínas que son idénticas en secuencia a la histona canónica H4. [4] Las variantes de H4 en humanos se identificaron recientemente y aún se conocen poco. [5] [6] [7]
Se han identificado variantes patogénicas de novo de pérdida de sentido en seis genes codificantes de H4 (HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4I, HIST1H4J) en un total de 33 individuos, presentando todos estos deficiencias intelectuales, motoras en el desarrollo del sistema nervioso. Diez aminoácidos se vieron afectados, de los cuales seis se identificaron recurrentemente. Estas mutaciones se localizaban en el dominio globular central de H4 (involucrado en interacciones proteína-proteína) o en la cola C-terminal (involucrada en modificaciones postraduccionales).[5] [6] [7]
Traducción alternativa
El Péptido de Crecimiento Osteogénico (OGP) es un péptido de 14 aminoácidos producto de la traducción alternativa de la histona H4, compartiendo la misma secuencia de la cola C-terminal ALKRQGRTLYGFGG. La traducción comienza en el aminoácido en posición 85 del ARN mensajero de la histona H4, produciendo un péptido de 19 aminoácidos (preOGP). Este se transforma en OGP mediante la escisión de cinco residuos del extremo N-terminal. [8] Este péptido se encuentra en la sangre de humanos y ratas, así como en médula ósea en regeneración. En el suero sanguíneo se encuentra unido a α2M junto con otras dos proteínas de unión que no se han identificado con precisión. No se ha encontrado aún un receptor específico, aunque sí algunas rutas de señalización involucradas en la regeneración de tejido óseo.[9]
Modificaciones postraduccionales
Los organismos eucariotas pueden producir pequeñas cantidades de histonas variantes especializadas, las cuales difieren en la secuencia de aminoácidos de las histonas canónicas. Estas variantes con modificaciones covalentes en el extremo N-terminal se pueden añadir a las histonas, creando diferentes estructuras en la cromatina, necesarias para la función del ADN en organismos eucariotas superiores. Las modificaciones potenciales incluyen: metilación (mono-, di- o trimetilación) o acetilación en las colas. [3]
Metilación
La metilación de histonas ocurre en argininas, lisinas e histidinas. Se ha descubierto mono-, di- y trimetilación en las histonas H2A, H3 y H4. [10] Se ha asociado la metilación de histonas con diferentes funciones celulares, tales como la transcripción y replicación del ADN, así como la respuesta a daños en este, incluyendo reparación, formación de heterocromatina y reprogramación de células somáticas. Entre estas funciones biológicas, las más estudiadas son la represión y activación transcripcional. [10] La metilación de H4R3, por la histona metiltransferasa PRMT1, parece ser esencial para el establecimiento y mantenimiento in vivo de un amplio rango de modificaciones activas en la cromatina. Sin embargo, la acetilación de H4 inhibe su metilación por PRMT1. [11]
Acetilación
Se cree que la acetilación de histonas relaja la condensación de la heterocromatina ya que la carga negativa de los grupos acetilo repelen las cargas de los grupos fosfato del ADN. Esto reduce la afinidad de unión de las histonas por el ADN. Esta hipótesis fue validada con el descubrimiento de las enzimas histona acetiltransferesas (HATs) de diferentes complejos activadores de la transcripción. [10] La acetilación en histonas influencia en la estructura de la cromatina de diferentes maneras. En primer lugar, puede ser una diana de unión para proteínas que contienen regiones que reconocen las colas acetiladas. En segundo lugar, pueden bloquear la función de los remodeladores de cromatina.[12] En tercer lugar, neutralizan las cargas positivas en lisinas. [12] La acetilación de histonas H4 en la lisina 16 (H4K16ac) es especialmente importante para la estructura y función de la cromatina en variedad de eucariotas y es catalizada por HATs específicas. H4K16 es el único sitio de la cola N-terminal que se puede acetilar, y puede influir en la condensación de la cromatina. [12] La hipoacetilación de H4K16 parece causar un reclutamiento retardado de proteínas reparadoras del ADN a regiones dañadas en este en modelos de ratón que sufren del síndrome de Hutchinson Gliford, también denominado progeria. [13] H4K16ac también está involucrada en la activación de la transcripción y el mantenimiento de la eucromatina. [14]
Lista de modificaciones de la histona H4
- H4S1p
- H4R3me
- H4K5ac
- H4K8ac
- H4K12ac
- H4K16ac
- H4K16adp
- H4K20me
- H4S47o-p
- H4K91ac
- H4K91ub [15]
Véase también
- Nucleosoma
- Histona
- Cromatina
- Otras proteínas histonas involucradas en la cromatina:
Referencias
- Reche, Pedro A.; Keskin, Derin B.; Hussey, Rebecca E.; Ancuta, Petronela; Gabuzda, Dana; Reinherz, Ellis L. (18 de mayo de 2006). «Recognition and classification of histones using support vector machine». Journal of Computational Biology 13 (1): 102-112. PMID 16472024. doi:10.1089/cmb.2006.13.102.
- Daniel L., Hartl (1988). Basic Genetics (en inglés). Boston: Jones and Bartlett Publishers. ISBN 978-0-86720-090-4.
- Bruce, Alberts; John H., Wilson; Tim, Hunt (2008). Molecular Biology of the Cell (5th edición). ISBN 978-0-8153-4105-5. OCLC 82473851.
- Kamakaka, Rohinton T.; Biggins, Sue (febrero de 2005). «Histone variants: deviants?». Genes & Development 19 (3): 295-310. PMID 15687254. doi:10.1101/gad.1272805.
- Tessadori, Federico; Giltay, Jacques C.; Hurst, Jane A.; Massink, Maarten P.; Duran, Karen; Vos, Harmjan R. et al. (noviembre de 2017). «Germline mutations affecting the histone H4 core cause a developmental syndrome by altering DNA damage response and cell cycle control». Nature Genetics 49 (11): 1642-1646. PMID 28920961. doi:10.1038/ng.3956.
- Tessadori, Federico; Rehman, Atteeq U.; Giltay, Jacques C.; Xia, Fan; Streff, Haley; Duran, Karen et al. (mayo de 2020). «A de novo variant in the human HIST1H4J gene causes a syndrome analogous to the HIST1H4C-associated neurodevelopmental disorder». European Journal of Human Genetics 28 (5): 674-678. PMC 7171094. PMID 31804630. doi:10.1038/s41431-019-0552-9.
- Tessadori, Federico; Duran, Karen; Knapp, Karen; Fellner, Matthias; Smithson, Sarah; Meireles, Ana B. et al. (abril de 2022). «Recurrent de novo missense variants across multiple histone H4 genes underlie a neurodevelopmental syndrome». American Journal of Human Genetics 109 (4): 750-758. PMC 9069069. PMID 35202563. doi:10.1016/j.ajhg.2022.02.003.
- Bab, I.; Smith, E.; Gavish, H.; Attar-Namdar, M.; Chorev, M.; Chen, Y. C. et al. (mayo de 1999). «Biosynthesis of osteogenic growth peptide via alternative translational initiation at AUG85 of histone H4 mRNA». The Journal of Biological Chemistry 274 (20): 14474-14481. PMID 10318873. doi:10.1074/jbc.274.20.14474.
- Pigossi, Suzane C.; Medeiros, Marcell C.; Saska, Sybele; Cirelli, Joni A.; Scarel-Caminaga, Raquel M. (noviembre de 2016). «Role of Osteogenic Growth Peptide (OGP) and OGP(10-14) in Bone Regeneration: A Review». International Journal of Molecular Sciences 17 (11): 1885. PMC 5133884. PMID 27879684. doi:10.3390/ijms17111885.
- Kim, J. K.; Samaranayke, M.; Pradhan, S. (febrero de 2009). «Epigenetic mechanisms in mammals». Cellular and Molecular Life Sciences 66 (4): 596-612. PMC 2780668. PMID 18985277. doi:10.1007/s00018-008-8432-4.
- Huang, Suming; Litt, Michael; Felsenfeld, Gary (agosto de 2005). «Methylation of histone H4 by arginine methyltransferase PRMT1 is essential in vivo for many subsequent histone modifications». Genes & Development 19 (16): 1885-1893. PMC 1186188. PMID 16103216. doi:10.1101/gad.1333905.
- Taylor, Gillian C. A.; Eskeland, Ragnhild; Hekimoglu-Balkan, Betül; Pradeepa, Madapura M.; Bickmore, Wendy A. (diciembre de 2013). «H4K16 acetylation marks active genes and enhancers of embryonic stem cells, but does not alter chromatin compaction». Genome Research 23 (12): 2053-2065. PMC 3847775. PMID 23990607. doi:10.1101/gr.155028.113.
- Krishnan, Vaidehi; Ying Chow, Maggie Zi; Wang, Zimei; Zhang, Le; Liu, Baohua; Liu, Xinguang; Zhou, Zhongjun (julio de 2011). «Histone H4 lysine 16 hypoacetylation is associated with defective DNA repair and premature senescence in Zmpste24-deficient mice». Proceedings of the National Academy of Sciences of the United States of America 108 (30): 12325-12330. Bibcode:2011PNAS..10812325K. PMC 3145730. PMID 21746928. doi:10.1073/pnas.1102789108.
- Shogren-Knaak, M.; Ishii, H.; Sun, Jian-Min; Pazin, M.; Davie, J.; Peterson, C. (febrero de 2006). «Histone H4-K16 acetylation controls chromatin structure and protein interactions». Science 311 (5762): 844-847. Bibcode:2006Sci...311..844S. PMID 16469925. doi:10.1126/science.1124000.
- «Epigenetic modifications poster | Abcam». Consultado el 25 de noviembre de 2019.