Protéines LSm
En biologie, les protéines LSm sont une famille de protéines se liant à l'ARN trouvées dans pratiquement tous les organismes. LSm est une contraction de Like Sm (« comme Sm »), parce que les premiers membres identifiés de cette famille ont été les protéines Sm. Les protéines LSm sont définies par leur structure tridimensionnelle caractéristique et leur assemblage en anneaux de six ou sept molécules.
Pour les articles homonymes, voir LSM.
| Protéines LSm | ||
 Le tore de l'hexamère Hfq (une LSm) montrant l'ossature peptidique, chaque protéine étant d'une couleur différente, chaque feuillet bêta étant représenté par un ruban et l'hélice alpha par un cylindre. L'arc à 300° au centre représente l'oligonucléotide d'ARN. | ||
| Caractéristiques générales | ||
|---|---|---|
| Profils d'expression de l’ARN | ||
| Espèces du Vivant | ||
Ces protéines ont été découvertes comme des antigènes ciblés par des anticorps anti-Sm chez une patiente ayant une forme de lupus érythémateux disséminé (LED), une maladie auto-immune débilitante. Elles ont été nommées Sm en hommage à cette patiente, Stéphanie Smith. D'autres protéines ayant des structures très similaires ont ensuite été découvertes et nommées protéines LSm. De nouvelles protéines LSm continuent d'être découvertes.
Les protéines avec des structures similaires sont regroupées en hiérarchie : famille, super-famille, sous-familles et folds (type de repliement). La structure des protéines LSm est un exemple de protéines pliées en feuillets bêta en forme de tube court. Chaque protéine s'assemble en un cycle de six ou sept sous-unités formant un tore qui se lie généralement à une petite molécule d'ARN pour former un complexe ribonucléoprotéique. Le tore aide la molécule d'ARN à conserver sa structure tridimensionnelle. Selon le type de protéines et d'ARN impliqués, ces complexes facilitent une grande variété d'étapes de la maturation de l'ARN dont la dégradation, l'édition, l'épissage et la régulation.
D'autres noms équivalents comme LSm folds, Sm-like, LSm sont fréquemment utilisés et tout aussi acceptables.
Historique
Découverte de l'antigène Smith
L'histoire de la découverte des premières LSm commence avec une jeune femme, Stéphanie Smith, chez qui fut diagnostiqué en 1959 un lupus érythémateux disséminé (LED) et qui finit par succomber des complications de la maladie en 1969 à l'âge de 22 ans. Pendant cette période, elle fut traitée au New York Hospital de l'Université Rockefeller par les docteurs Henry Kunkel et Eng Tan. Les patients atteints de cette maladie auto-immune produisent des anticorps contre des antigènes situés dans le noyau de leurs cellules, le plus souvent contre leur propre ADN. Toutefois, les Dr Kunkel et Tan constatèrent en 1966 que Mme Smith produisait des anticorps contre un ensemble de protéines nucléaires, qu'ils ont appelé l'« antigène Smith » (Sm Ag)[1]. Environ 30 % des patients atteints de LED produisent des anticorps contre ces protéines, par opposition à ceux qui en fabriquent contre l'ADN double brin. Cette découverte a amélioré les tests diagnostiques de LED mais la nature et la fonction de cet antigène étaient inconnues.
Les protéines Sm, RNPsn, le splicéosome et l'épissage de l'ARN messager
Les recherches se sont poursuivies pendant les années 1970 et 1980. L'antigène Smith s'est révélé être un complexe d'acide ribonucléique (ARN) et de multiples protéines. De petits fragments d'ARN nucléaire non codants riches en uridine - les actuels snRNAs - faisaient partie de ce complexe et ont reçu les noms U1, U2, U4, U5 et U6. Puis on a découvert que quatre de ces ARNsn (U1, U2, U4 et U5) étaient étroitement liées à plusieurs petites protéines nommées SmB, SmD, SmE, SmF et SmG par ordre décroissant de taille. SMB possède un variant à pliage antiparallèle appelé SmB', et une protéine très similaire, SmN, remplace SmB/B' dans certains tissus (surtout nerveux). On a aussi découvert que SmD était un mélange de trois protéines, nommées SmD1, SmD2 et SmD3. Ces neuf protéines (SmB, SmB', SmN, SmD1, SmD2, SmD3, SmE, SmF et SmG) sont connues sous le nom de Sm core proteins ou tout simplement de protéines Sm. Les ARNsn forment des complexes avec des protéines Sm et d'autres protéines pour former dans le noyau de la cellule des particules appelées petites ribonucléoprotéines nucléaires ou Small Nuclear Ribonucleoproteins (snRNP). Au milieu des années 1980, on a compris que ces snRNP participaient à la formation d'un grand (4,8 MD de poids moléculaire) complexe protidique, le splicéosome, autour du pré-ARNm, coupant (excision) des parties du pré-ARNm appelées introns et épissant les parties codantes (exons)[2]. Après quelques modifications supplémentaires, le pré-ARNm épissé devient l'ARN messager (ARNm) qui est ensuite exporté du noyau vers le cytoplasme et sera traduit en une protéine par les ribosomes.
Découverte de protéines similaires aux protéines Sm
L'ARNsn U6 (contrairement aux autres ARN du même groupe) ne s'associe pas aux protéines Sm, même si le snRNP U6 est un élément central du splicéosome. En 1999, on a identifié un complexe protéique qui se lie spécifiquement à U6 et qui se compose de sept protéines clairement homologues aux protéines Sm. On a appelé ces protéines LSm (Like Sm, « comme Sm ») (LSm1, LSm2, LSm3, LSm4, LSm5, LSm6 et LSm7) avec la protéine LSm8 qui sera découverte ultérieurement. Le génome de Saccharomyces cerevisiae (la levure de boulanger) a été séquencé au milieu des années 1990, fournissant une ressource précieuse pour identifier des homologues de ces protéines humaines. Par la suite comme on a su séquencer les génomes d'autres eucaryotes, on a constaté que les eucaryotes, en général, possédaient des protéines homologues à la série des sept Sm et des huit LSm[3]. Peu de temps après, des protéines homologues aux protéines LSm des eucaryotes ont été trouvées chez les archées (Sm1 et Sm2) et chez les bactéries (Hfq et YlxS)[4]. Fait intéressant, les LSm des Archées sont plus proches des LSm des eucaryotes que de celles des bactéries. Les LSm décrites jusqu'ici sont plutôt de petites protéines, allant de 76 acides aminés (8.7 kD de poids moléculaire) pour la SmG humaine à 231 acides aminés (29 kD de poids moléculaire) pour la SmB humaine. Mais récemment, on a découvert de plus grosses protéines possédant un domaine structurel LSm en plus d'autres domaines structurels (telles que LSm10, LSm11, LSm12, LSm13, LSm14, LSm15, LSm16, ataxin-2 ainsi que la Sm3 des archées).
Découverte des LSM folds
Vers 1995, les comparaisons entre les diverses LSm identifiées ont mis en évidence deux séquences de motifs, l'une de 32 et l'autre de 14 acides aminés, très similaires dans les différentes molécules et séparées par une région variable de longueur variable. Cela montrait l'importance de ces deux motifs de séquence (nommés Sm1 et Sm2) et suggérait que tous les gènes des LSm évoluaient à partir d'un gène ancestral commun[5]. En 1999, on a pu obtenir des cristaux de protéines recombinantes Sm permettant, par diffractométrie de rayons X, la détermination leur structure atomique en trois dimensions[6]. On a ainsi pu constater qu'elles se composaient toutes d'une structure tridimensionnelle formée d'une courte hélice α et de un à cinq feuillets β, structure dénommée par la suite LSm fold. D'autres études ont montré que les protéines LSm s'assemblaient en un tore (sorte d'anneau) de six ou sept protéines et que l'ARN se fixait à l'intérieur du tore, avec un nucléotide lié à chaque protéine.
Caractéristiques

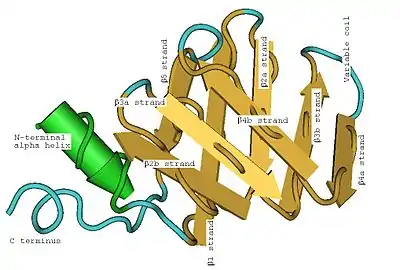
Les LSm sont caractérisées par un feuilletage β (structure secondaire), replié sur lui-même (structure tertiaire), rassemblé en six ou sept unités (structure quaternaire) et se liant à des oligonucléotides d'ARN[7].
Structure secondaire
La structure secondaire d'une protéine LSm est un feuillet de cinq brins anti-parallèles β, avec les brins identifiés à partir de l'extrémité N-terminale vers l'extrémité C-terminale par β1, β2, β3, β4, β5. La séquence de motifs Sm1 correspond aux brins β1, β2, β3 et la séquence Sm2 correspond aux brins β4 et β5. Les quatre premiers brins β sont adjacents entre eux, mais β5 est adjacent à β1, transformant la structure d'ensemble en un court cylindre. Cette topologie structurelle est décrite comme 51234. De courte longueur (deux à quatre tours de spire) l'hélice α N-terminale est également présente dans la plupart des LSm. Les brins β3 et β4 sont courts dans certaines LSm et séparés par un portion non structurés de longueur variable. Les brins β2, β3 et β4 sont courbés à 120° en leur milieu, les coudes correspondant souvent à la présence de glycine et les chaînes latérales internes au cylindre à des résidus hydrophobes: valine, leucine, isoleucine et méthionine.
Structure tertiaire
La structure tertiaire en tonneau des LSm est due aux brins β2, β3 et β4 fortement courbés (environ 120°) et au brin β1 qui vient fermer l'anneau avec le brin β5. Pour bien comprendre cette structure tertiaire, on peut imaginer dérouler l'anneau et se représenter une structure plane. Chaque brin β plié peut être considéré comme deux brins βa et βb plus courts. Le pliage de la LSm peut alors être considéré comme un pliage en deux plans (on parle de pliage en sandwich) de huit brins β anti-parallèles, avec cinq brins dans un plan et les trois autres dans un plan parallèle, les brins du premier plan faisant un angle d'environ 45° avec ceux du second. L'hélice α est placée à une extrémité du sandwich. L'hélice et les brins peuvent être étiquetés (de l'extrémité N-terminale à l'extrémité C-terminale) α, β1, β2a, β2b, β3a, β3b, β4a, β4b, β5 où les a et b se référent aux deux moitiés d'un brin tordu. Chacun des huit brins β est formé de cinq résidus d'acides aminés. En comptant les coudes, les boucles entre les brins et l'hélice α, environ 60 acides aminés contribuent à la formation d'une LSm, mais ce nombre varie entre homologues en raison de variations dans les boucles inter-brins, l'hélice α et même les longueurs des brins β3b et β4a.
Structure quaternaire
Les monomères de LSm s'assemblent en général en un anneau formé de six ou sept unités, tore d'environ 7 nanomètres de diamètre avec un trou de 2 nanomètres. Les LSm ancestrales sont des homohexamères ou homoheptamères c'est-à-dire que les 6 ou 7 unités du tore sont identiques. Chez les eucaryotes, on a affaire à des hétéroheptamères formés de sept unités toutes différentes, comme les protéines Sm par exemple. On comprendra mieux les liaisons entre les unités si l'on se rappelle, la description déroulée faite au paragraphe précédent. Les cinq brins β du premier plan d'une unité s'apparient avec les trois brins du second plan de l'unité adjacente, formant un feuillet torsadé Aβ4a/Aβ3b/Aβ2a/Aβ1/Aβ5/Bβ4b/Bβ3a/Bβ2b, où A et B se rapportent aux deux unités différentes. En plus des liaisons hydrogène entre les brins Aβ5 and Bβ4b des deux unités, il y a des contacts énergétiques favorables entre les chaînes latérales hydrophobes d'acides aminés à l'intérieur de la zone de contact et d'autres entre les chaînes latérales hydrophiles à la périphérie de l'aire de contact.
Liaison à l'ARN
Les anneaux de LSm forment des complexes avec des oligonucléotides d'ARN qui varient de liaisons fortes donnant des complexes très stables (tels que les snRNP de classe Sm) à des liaisons faibles de complexes transitoires. Pour autant que l'on connaisse les détails de ces liaisons, l'ARN se place généralement à l'intérieur du trou du tore LSm, un seul nucléotide étant fixé à une unité LSm, mais on a noté aussi d'autres liaisons des nucléotides avec l'extrémité (côté hélice α) des protéines. La nature chimique exacte de ces liaisons est variable, mais on a notamment des empilements alternés de base nucléique (souvent de l'uracile) entre deux acides aminés des chaînes latérales, des liaisons hydrogène entre acides aminés et base nucléique et/ou ribose, et des ponts salins entre acides aminés et groupement phosphate.
Références
- E. M. Tan and H. G. Kunkel, « Characteristics of a soluble nuclear antigen precipitating with sera of patients with systemic lupus erythematosus ». The Journal of Immunology (1966) 96: 464-471.
- C. L. Will and R. Luhrmann, "Spliceosomal UsnRNP biogenesis, structure and function". Current Opinion in Cell Biology (2001) 13: 290-301.
- W. He and R. Parker, "Functions of Lsm proteins in mRNA degradation and splicing". Current Opinion in Cell Biology (2000) 12: 346-350
- I. Toro, S. Thore, C. Mayer, J. Basquin, B. Seraphin, D. Suck, "RNA binding in an Sm core domain: X-ray structure and functional analysis of an archaeal Sm protein complex". The EMBO Journal (2001) 20:2293-2303.
- H. Hermann, P. Fabrizio, V.A. Raker, K. Foulaki, H. Hornig, H. Brahms, R. Luhrmann, "snRNP Sm proteins share two evolutionarily conserved sequence motifs which are involved in Sm protein-protein interactions". The EMBO Journal (1995) 14:2076-2088.
- C. Kambach, S. Walke, R. Young, J. M. Avis, E. de la Fortelle, V. A. Raker, R. Luhrmann, K. Nagai, "Crystal structures of two Sm protein complexes and their implications for the assembly of the spliceosomal snRNPs". Cell (1999) 96: 375-387.
- National Center for Biotechnology Information Structure Database PDB codes 1B34, 1D3B, 1I5L, 1KQ2, 1N9S, 1IB8.
- Portail de la biologie cellulaire et moléculaire