Réparation par excision de base
La réparation par excision de base (ou BER pour Base excision repair en anglais) est l'un des mécanismes de réparation de l'ADN utilisé par les cellules vivantes pour restaurer l'intégrité de l'ADN. Il est utilisé pour réparer les modifications chimiques survenues au niveau d’une base individuelle. Une telle lésion est réparée par simple élimination de la base, suivi du clivage du désoxyribose, et se termine par une nouvelle synthèse d'ADN intact remplaçant le nucléotide endommagé[1].
Pour les articles homonymes, voir BER.
Le mécanisme se déroule en trois ou quatre étapes suivant la nature de la lésion. Tout d'abord une ADN glycosylase élimine la base endommagée et produit un site abasique (ou site AP). Une endonucléase spécifique clive ensuite le désoxyribose de ce site abasique[2]. Une ADN polymérase remplit à nouveau l’espace libéré en utilisant la base intacte du brin opposé comme matrice. Enfin, une ADN ligase suture le brin réparé.
Donc, même si une seule base est endommagée, une série d’enzymes différentes sont nécessaires pour assurer une réparation correcte. La dernière étape de ce processus est également utilisée pour la réparation de simples cassures de la chaîne d’ADN.
La réparation par excision de base est limitée à quelques types de bases déterminés et se produit très rapidement. Différentes ADN glycosylases correspondent chacune spécifiquement à l’élimination d’un type de base endommagée. C’est ainsi que l’uracile-ADN glycosylase n’éliminera que les bases d’uracile et certains produits d’oxydation de l’uracile, tandis que d'autres ADN-glycosylases seront spécifiques d'autres types de bases altérées (8-oxoguanine...).
La réparation par excision de base est un mécanisme essentiel de maintien de l'intégrité de l'information génétique, conservé dans l'évolution. Elle fonctionne suivant les mêmes principes mécanistiques chez les bactéries et les eucaryotes. La première mise en évidence d'une uracile-ADN glycosylase spécifique a d'ailleurs été faite chez Escherichia coli[3].
Les types de lésions réparées par excision de base
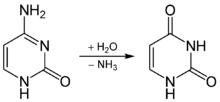
Le mécanisme de réparation par excision de base répare plusieurs types de lésion intervenant sur des bases. Il s'agit le plus souvent de modifications chimiques affectant les groupements fonctionnels de bases nucléotidiques, par oxydation, désamination ou alkylation. Parce que ces groupements fonctionnels sont impliqués dans la formation des liaisons hydrogène dans les appariements de bases, ces lésions présentent un caractère mutagène et doivent donc être réparées pour éviter une altération de l'information génétique.
La réparation par excision de base permet également à la cellule de réparer les sites abasiques, conséquences des réactions de dépurination spontanée (hydrolyse de la liaison glycosidique entre les bases puriques, A et G, et le désoxyribose). Dans ce dernier cas, la première étape faisant intervenir une ADN glycosylase n'est pas nécessaire, puisque le site abasique est produit directement par la lésion.
Les autres lésions réparées par excision de base sont :
- Les uraciles, produits par désamination spontanée des cytosines lors d'expositions chimiques ou enzymatiques (et aussi l'hypoxanthine, produit de désamination du A)
- Certaines bases oxydées : 8-oxoguanine (produit d'oxydation du G), thymine glycol (produit d'oxydation du T)
- Certaines bases alkylées : 7-méthylguanosine,…
Les étapes du mécanisme
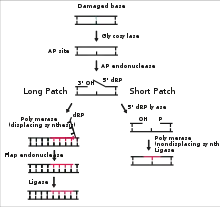
La première étape du mécanisme de réparation par excision de base est l'enlèvement de la base endommagée par une ADN glycosylase spécifique de chaque type de dommage : uracile-ADN glycosylase, oxoguanine-ADN glycosylase, méthylpurine-ADN glycosylase… On a ainsi identifié 11 ADN-glycosylases différentes chez les mammifères[4]. Ces ADN glycosylases reconnaissent et ôtent la base modifiée par hydrolyse de la liaison N-glycosidique entre la base et le désoxyribose. Ceci produit un site abasique ou site AP (apurique, apyrimidinique) sur l'ADN, un «trou» d'une base dans le duplex.
Le site AP est reconnu par une endonucléase spécifique, l'AP endonucléase, au niveau du site AP. Cette nucléase coupe le squelette phosphodiester en laissant un hydroxyle 3’-OH et un 5’-phosphate du côté du désoxyribose-5-phosphate. La position correspondant à la base manquante est synthétisée par une ADN polymérase, en utilisant le brin intact comme matrice et l'extrémité 3'-OH libérée par l'AP endonucléase comme amorce.
Deux voies de synthèse sont possibles[5] : soit une synthèse courte, limitée à un ou deux nucléotides, effectuée par une ADN polymérase spécialisée, peu processive, comme l'ADN polymérase bêta chez les eucaryotes ou l'ADN polymérase I chez les bactéries; soit une synthèse longue, effectuée par une ADN polymérase processive (ADN polymérase delta, chez les eucaryotes). Le désoxyribose et/ou le brin déplacé par cette synthèse est excisé par une nucléases.
La réparation se termine par la suture du brin réparé par une ADN ligase, ce qui rétablit la continuité du brin réparé.
Découverte
Le mécanisme de réparation par excision de base a été entre autres découvert par Tomas Lindahl qui a été récompensé à ce titre par le prix Nobel de chimie en 2015.
Notes et références
- Xavier Coumoul, Frédéric Dardel et Étienne Blanc, Mémo visuel de Biochimie : l'essentiel en fiches, Paris, Dunod, , 226 p. (ISBN 978-2-10-074226-4), p. 130
- Tomas Lindahl, « DNA glycosylases, endonucleases for apurinic/apyrimidinic sites, and base excision-repair », Progress in Nucleic Acid Research and Molecular Biology, vol. 22, , p. 135–192 (ISSN 0079-6603, PMID 392601, lire en ligne, consulté le )
- T. Lindahl, « An N-glycosidase from Escherichia coli that releases free uracil from DNA containing deaminated cytosine residues », Proceedings of the National Academy of Sciences of the United States of America, vol. 71, no 9, , p. 3649–3653 (ISSN 0027-8424, PMID 4610583, PMCID 433833, lire en ligne, consulté le )
- (en) Angelika L. Jacobs et Primo Schär, « DNA glycosylases: in DNA repair and beyond », Chromosoma, vol. 121, no 1, , p. 1–20 (ISSN 0009-5915 et 1432-0886, PMID 22048164, PMCID 3260424, DOI 10.1007/s00412-011-0347-4, lire en ligne, consulté le )
- Paola Fortini et Eugenia Dogliotti, « Base damage and single-strand break repair: mechanisms and functional significance of short- and long-patch repair subpathways », DNA repair, vol. 6, no 4, , p. 398–409 (ISSN 1568-7864, PMID 17129767, DOI 10.1016/j.dnarep.2006.10.008, lire en ligne, consulté le )
Voir aussi
Articles connexes
Liens externes
- Conférence Nobel de Tomas Lindahl, découvreur de la réparation par excision de base, sur le site Nobel
- Portail de la biologie cellulaire et moléculaire