فيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الشديدة
فيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الشديدة (SARSr-CoV)[ملاحظة 1] هو نوع من فيروسات كورونا يصيب البشر والخفافيش وبعض الثدييات الأخرى.[4][5]، وهو فيروس رنا مفرد السلسلة موجب الاتجاه مغلف يدخل خلايا مضيفه عبر الارتباط بمستقبل ACE2. وينتمي إلى جنس فيروس كورونا بيتا والجنس الفرعي فيروس ساربيكو.[6][7]
اضغط هنا للاطلاع على كيفية قراءة التصنيف فيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الشديدة | |
|---|---|
 صورة بالمجهر الإلكتروني النافذ لفيروسات تاجية متعلقة بسارس الناشئة من الخلايا المضيفة المستنبتة في المختبر | |
| المرتبة التصنيفية | نوع[1][2] |
| التصنيف العلمي | |
| فوق النطاق | حيويات |
| مملكة | Pisoniviricetes |
| رتبة | فيروسات عشية |
| فصيلة | فيروسات تاجية |
| فُصيلة | فيروس كورونا |
| جنس | فيروس كورونا بيتا |
| جُنيس | ساربيكوفيروس |
| الاسم العلمي | |
| Severe acute respiratory syndrome-related coronavirus[1][2] | |
| السلالات | |
| |
| مرادفات | |
| SARS coronavirus SARS-related coronavirus | |
تسببت سلالتين من الفيروس في تفشيات لأمراض تنفسية شديدة لدى البشر: فيروس كورونا السارس (سارس-كوف) الذي تسبب في تفشي المتلازمة التنفسية الحادة الوخيمة (السارس) بين 2002 و2003، وفيروس كورونا السارس 2 الذي يُحدث منذ أواخر 2019 جائحة مرض فيروس كورونا 2019 (كوفيد 19).[8][9] توجد المئات من السلالات الأخرى من فيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الشديدة، ومعروف بأن جميعها يصيب أنواعا غير بشرية. الخفافيش هي الخزان الرئيسي للعديد من سلالات فيروسات كورونا المرتبطة بالسارس، وتم تحديد العديد من السلالات في زباد النخل والتي على الأرجح هي أسلاف لفيروس كورونا السارس.[8][10]
فيروس كورونا المرتبط بالسارس هو أحد عدة فيروسات صنفتها منظمة الصحة العالمية في 2016 بأنها من الممكن أن تسبب وباء مستقبليا في مخطط جديد طُوِّر بعد وباء إيبولا للبحث والتطوير العاجل -قبل وأثناء الوباء- لاختبارات تشخيص، لقاحات وأدوية. تحقق هذا التنبؤ مع حدوث جائحة فيروس كورونا 2019–20.[11][12]
التصنيف
فيروس كورونا المرتبط بالسارس هو عضو من جنس فيروس كورونا بيتا (المجموعة 2) والجنس الفرعي فيروس كورونا ساربي (Sarbecovirus) (المجموعة الفرعية B).[13] فيروسات كورونا ساربي وعلى خلاف فيروسات كورونا إيمبي أو فيروسات كورونا ألفا لا تملك سوى ببتيداز شبيه بالباباين وحيد بدل اثنين في إطار القراءة المفتوح 1 (ORF1).[14] حُدِّد أن فيروس كورونا المرتبط بالسارس هو تفرعٌ باكرٌ من مجموعة فيروسات كورونا بيتا وذلك استنادا على مجموعة من النطاقات المحفوظة التي يشترك فيها مع المجموعة.[15][16]
الخفافيش هي المخزن المضيف الرئيسي لفيروس كورونا المرتبط بالسارس، حيث تطور الفيروس بشكل مشترك مع خفاش مضيف عبر مدة طويلة من الزمن.[17] ومؤخرا فقط، تطورت سلالات من فيروس كورنا المرتبط بالسارس وقامت بالانتقال عبر الأنواع من الخفافيش إلى البشر، كما هو الحال في سلالتي فيروس كورونا السارس، وفيروس كورونا السارس 2.[18][19] تنحدر كلا هاتين السلالتين من سلف واحد لكنهما قاما بالانتقال عبر الأنواع بشكل منفصل. فيروس كورونا السارس 2 ليس سليلا مباشرا من فيروس كورونا السارس.[8]
الجينوم
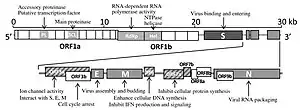
فيروس كورونا المرتبط بالسارس هو فيروس رنا مفرد السلسلة موجب الاتجاه مغلف، طول جينومه حوالي 30 ألف قاعدة وهو أحد أطول فيروسات الرنا. يملك هذا الفيروس 14 إطار قراءة مفتوح متداخلة في بعض الحالات.[20] للجينوم قبعة 5' ممثيلة وذيل عديد أدينين 3'،[21] ويوجد 265 نوكليوتيد في المنطقة 5' غير المترجمة (5′UTR) و342 نوكليوتيد في المنطقة 3' غير المترجمة (3′UTR).[20]
تسمح القبعة 5' الممثيلة وذيل عديد الأدينين 3' لجينوم الرنا موجب الاتجاه بأن يُترجم مباشرة بواسطة ريبوسومات الخلية المضيفة عند دخول الفيروس.[22] فيروس كورونا المرتبط بالسارس مماثل لفيروسات كورونا الأخرى في أن التعبير عن جينومه يبدأ بترجمة ريبوسومات الخلية لإطاري القراءة المفتوحين الكبيرين المتداخلين 1a و1b الذين ينتج كلاهما عديد بروتين.[20]
وظيفة العديد من البروتينات الفيروسية معروفة.[23] حيث يشفر إطارا القراءة 1a و1b عديد البروتين الخاص ببوليميراز الرنا المعتمد على الرنا (ريبليكاز الرنا) المعروف كذلك باسم منتسخة الرنا (ترنسكريبتاز)، وتشفر إطارت القراءة: 2a و4a و5a و9a على التوالي البروتينات البنيوية الرئيسية الأربعة: الحسكة، الغلاف، الغشاء والقفيصة المنواة.[24] وتشفر إطارات القراءة التالية من 3a حتى 9b ثمانية بروتينات فريدة تعرف باسم البروتينات الملحقة أو الإضافية والتي لا يوجد نظير لمعظمها لدى فيروسات أخرى. الوظائف المختلفة للبروتينات الملحقة ليست مفهومة جيدا.[23]
| وظيفة بروتينات جينوم فيروس السارس (SARS-CoV) من إطار القراءة المفتوح 1a حتى 9b (orf1a to orf9b). | |
|---|---|
| إطار القراءة المفتوح | الوظيفة[25][26][27] |
| orf1a وorf1b | عديد بروتين ريبليكاز الرنا/ منتسخة الرنا (pp1ab)، نسخ الجينوم لإنشاء فيروسات أخرى. (بروتين غير بنيوي) |
| orf2 | بروتين الحسكة (S)، ارتباط الفيروس بالمستقبل ودخوله للخلية. (بروتين بنيوي) |
| orf3a | يتآثر مع البروتينات البنيوية: الحسكة S والغلاف E والغشاء M. يملك نشاط قناة أيونية. ينظم بالزيادة السيتوكينات والكيموكينات مثل إنترلوكين 8 وCCL5. ينظم بالزيادة NF-κB و JNK يحدث الاستماتة وتوقف دورة الخلية عبر كاسباز 8 و9، وبواسطة BAX وبي53، وكذلك كيناز MAP P38. |
| orf3b | ينظم بالزيادة السيتوكينات والكيموكينات بواسطة RUNX1. يثبط إنتاج إنترفيرون نوع 1 والتأشير بواسطته. يحدث الاستماتة وتوقف دورة الخلية. |
| orf4 | بروتين الغلاف (E)، تجميع الفيروس وتبرعمه (خروجه من الخلية). (بروتين بنيوي) |
| orf5 | بروتين الغشاء (M)، تجميع الفيروس وتبرعمه (خروجه من الخلية). (بروتين بنيوي) |
| orf6 | يحسن من التخليق الخلوي للدنا. يثبط إنتاج إنترفيرون نوع 1 والتأشير بواسطته. |
| ORF7a | يثبط التخليق الخلوي للبروتين. يحدث استجابة التهابية عبر NF-κB ومعزز إنترلوكين 8. ينظم بالزيادة الكيموكينات مثل إنترلوكين 8 و.CCL5. ينظم بالزيادة JNK وكيناز MAP P38. يحدث الاستماتة وتوقف دورة الخلية. |
| orf7b | غير معروف |
| orf8a | يحدث الاستماتة عبر مسارٍ للمتقدرات |
| orf8b | يحسن من التخليق الخلوي للدنا. |
| orf9a | بروتين القفيصة المنواة (N)، تغليف الرنا الفيروسي. (بروتين بنيوي) |
| orf9b | يُحدث الاستماتة الخلوية |
تطور السلالات
أظهرت دراسات تطور السلالات أن الفرع التطوري المكون من فيروسي كورونا الخفاشيين BtKY72 وBM48-31 كان المجموعة القاعدية للشجرة التطورية الخاصة بالفيروسات المرتبطة بالسارس، والتي انفصلت عن الفيروسات المرتبطة بالسارس قبل ظهور سارس-كوف وسارس-كوف-2.[28][29]
| فيروس كورونا المرتبط بالسارس |
| ||||||||||||||||||||||||||||||||||||||||||
المرتبطة بسارس-كوف-1
شجرة تطور السلالات الخاصة بسلالة سارس-كوف والسلالات القريبة لها بناء على دراسات سلسلة كامل الجينوم.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
سارس-كوف-2 79 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
فيروس خفاشي فيروس زباد النخل فيروس بشري
المرتبطة بسارس-كوف-2
شجرة تطور السلالات الخاصة بسلالة سارس-كوف-2 والسلالات القريبة لها[37][38] :
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
سارس-كوف 79% | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
فيروس خفاشي فيروس آكل النمل الحرشفي فيروس بشري
الشكل البنيوي

مورفولوجيا فيروس كوونا المرتبط بالسارس هي سمة مميزة لكل عائلة فيروسات كورونا. وفيروسات كورونا هي جسيمات كروية متعددة الأشكال ذات امتدادات بصلية تظهر على شكل تاج (ومنه جاءت التسمية الفيروسات التاجية) حول الجسيمات في الصور المجهرية.[46] حجم الفيروس يترواح بين 80-90 نانومتر، ويبدو غلاف الفيروس في الصور المجهرية الإلكترونية كزوج مميز من أغشية كثافة الإلكترون.[47]
يتكون الغلاف الفيروسي من ليبيد ثنائي الطبقة تكون فيه بروتينات الغشاء (M) والغلاف (E) والحسكة (S) منغرسة أو راسية.[48] بروتينات الحسكة هي الامتدادت البصلية الخارجة من سطح الفيروس والتي تعطيه شكله المسنن التاجي. تآثر بروتينات الحسكة مع المستقبلات المتممة لها الموجودة على سطح الخلية هو أمر أساسي لتحديد الانتحاء النسيجي، الإعداء ومجال الأجناس التي يصيبها الفيروس.[49][50]
داخل الغشاء توجد القفيصة المنواة التي تتكون من عدة نسخ من بروتين القفيصة (N)، ترتبط هذه البروتينات مع جينوم الرنا الخاص بالفيروس (30 ألف قاعدة) أحادي السلسلة موجب الاتجاه في هيئية بنيوية من نوع خرزات على خيط.[51][52] يحمي غشاء اللبيد ثنائي الطبقة والبروتينات الغشائية والقفيصة المنواة الفيروس حين يكون خارج مضيفه.[53]
دورة الحياة
يتبع فيروس كورونا المرتبط بالسارس إستراتيجية التضاعف (التنسخ) النموذجية عند كل فيروسات كورونا.[21][54][55][56][57]
الارتباط والدخول
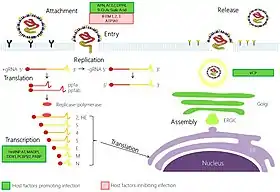
يحدث ارتباط فيروس كورونا السارس بالخلية المضيفة بواسطة بروتين الحسكة (spike protein) ومستقبِله،[58] يتعرف نطاق الارتباط بالمستقبل (RBD) الخاص ببروتين الحسكة على مستقبل الإنزيم المحول للأنجيوتنسين 2 (ACE2) ويرتبط به.[19] بعد الارتباط يدخل الفيروس إلى الخلية المضيفة عبر مسارين مختلفين. المسار الذي يتخذه الفيروس يعتمد على البروتياز المتواجد في الخلية المضيفة والذي يقص وينشط بروتين الحسكة المرتبط بالمستقبل.[59]
المسار الأول الذي يتخذه فيروس كورونا السارس لدخول الخلية المضيفة يتم بواسطة الإدخال الخلوي وأخذ الفيروس في جسيم داخلي. بعد ذلك يُفعَّل بروتين الحسكة المرتبط بالمستقبل بواسطة بروتياز السيستين كاتيبسين L المعتمد على الأس الهيدروجيني والخاص بالخلية المضيفة. يسبب تنشيط بروتين الحسكة المرتبط بالمستقبل تغيرا هيئيا يتلوه اندماج الغلاف الفيروسي مع غشاء الجسيم الداخلي.[59]
في المسار الثاني، يستطيع الفيروس دخول الخلية مباشرة عبر قصٍ حالٍ لبروتين الحسكة المرتبط بالمستقبل بواسطة بروتيازي السيرين الخاصين بالمضيف: بروتياز السيرين عبر الغشائي 2 وTMPRSS11D وذلك في سطح الخلية.[60][61] لدى فيروس كورونا السارس، يُحفِّز تنشيط جزء النهاية الكربوكسيلية من بروتين الحسكة اندماجَ الغلاف الفيروسي مع الغشاء الخلوي عبر إحداث تغيرات هيئية غير مفهومة بشكل كامل.[62]
ترجمة الجينوم
| وظيفة البروتينات اللابنيوية (nsps) لفيروس كورونا[63] | |
|---|---|
| البروتين | الوظيفة |
| nsp1 | يعزز تفكيك الرنا الرسول الخاص بالمضيف، يوقف الترجمة لدى المضيف. يوقف الاستجابة المناعية الفطرية. |
| nsp2 | يرتبط ببروتينات بروهبتين. غير معروف الوظيفة. |
| nsp3 | بروتين عبر غشائي عديد النطاقات، يتفاعل مع البروتين N ويعزز التعبير عن السيتوكينات. يقص نطاق باباين عديد البروتين pp1ab ويوقف الاستجابة المناعية الفطرية للمضيف. نطاقاته الأخرى غير معروفة الوظيفة. |
| nsp4 | بروتين سقالة عبر غشائي. يسمح باتخاذ البنية الصحيحة للحويصلات مزدوجة الغشاء (DMVs). |
| nsp5 | يقص 3CLPro عديد البروتين pp1ab. |
| nsp6 | بروتين سقالة عبر غشائي. وظيفة غير معروفة. |
| nsp7 | يشكل مركب سداسي القسمات مع nsp8، ملقاط عملياتية بوليميراز الرنا المعتمد على الرنا (RdRp) (nsp12). |
| nsp8 | يشكل مركب سداسي القسمات مع nsp7، ملقاط اضطراد بوليميراز الرنا المعتمد على الرنا (nsp12)، يعمل كبريماز. |
| nsp9 | بروتين مرتبط بالرنا (RBP). |
| nsp10 | عامل مرافق لـnsp16 وnsp14 ويشكل مثنوي مغاير مع كليهما. يحث نشاط مثيلة 2'-O (nsp16) وريبونوكلياز خارجي (nsp14). |
| nsp11 | وظيفة غير معروفة |
| nsp12 | بوليميراز الرنا المعتمد على الرنا (RdRp) |
| nsp13 | هيليكاز الرنا، 5' ثلاثي الفوسفاتاز. |
| nsp14 | ناقلة الميثيل N7، 3'-5' ريبونوكلياز خارجي (ExoN)، تضيف ناقلة الميثيل N7 القبعة 5'. يصحح الريبونوكلياز الخارجي (ExoN) الجينوم. |
| nsp15 | ريبونوكلياز داخلي (NendoU). |
| nsp16 | ناقلة ميثيل-2'-O (2-O-MT). يحمي الرنا الفيروسي من MDA5. |
بعد الاندماج تنتقل القفيصة إلى السيتوبلازم ويُحرر الجينوم الفيروسي هناك. يعمل الجينوم الفيروسي كرنا رسول ويقوم ريبوسوم الخلية بترجمة ثلثي الجينوم وهما إطارا القراءة المفتوح ORF1a وORF1b إلى عديدي بروتين كبيرين متداخلين pp1a وpp1ab
عديد البروتين الكبير pp1ab هو نتيجة انزياح الإطار الريبوسومي بـ -1 يُحدثه التسلسل الزلق UUUAAAC وعقدة رنا كاذبة مع المسار في نهاية إطار القراءة المفتوح ORF1a.[64] يسمح انزياح الإطار الريبوسومي بالترجمة المستمرة للإطار ORF1a متبوعا بـORF1b.[65]
تحتوي عديدات البروتين على البروتيازات الخاصة بها: PLpro و3CLpro اللذان يقصان عديدات البروتين في مواقع محددة مختلفة. ينتج عن قص عديد البروتين pp1ab 16 بروتينا لابنيويا (nsp) (nsp1 حتى nsp16). تشمل البروتينات الناتجة العديد من بروتينات النسخ والتضاعف مثل: بوليميراز الرنا المعتمد على الرنا (RdRp)، هيليكاز الرنا والريبونوكلياز الخارجي (ExoN).[56][65]
التضاعف والنسخ

يتكتل عددٌ من بروتينات التضاعف اللابنيوية لتشكيل معقد البروتين ريبليكاز-ترانسكريبتاز (RTC) عديد البروتينات.[65] البروتين الرئيسي في مركب الريبليكاز-ترانسكريبتاز هو بوليميراز الرنا المعتمد على الرنا (RdRp)، ويعمل الأخير بشكل مباشر على مضاعفة جينوم الرنا لتشكيل جينومات أخرى وكذلك نسخ إطارات القراءات لتخليق جزيئات الرنا الرسول الخاصة بالبروتينات الفيروسية، بينما تساعد البروتينات اللابنيوية الأخرى في عملية النسخ والتضاعف.[63]
البروتين اللابنيوي 15 (nsp15) هو 3'-5' ريبونوكلياز خارجي يعمل على تصحيح عملية النسخ وإضفاء دقة وصحة أكبر عليها. ويوفر للمعقد خاصية التصحيح التي لا يملكها بوليميراز الرنا المعتمد على الرنا. يشكل بروتيني nsp8 وnsp7 ملقاطا منزلقا وهو جزء من المعقد يزيد بشكل كبير عملياتية [الإنجليزية] بوليميراز الرنا المعتمد على الرنا.[63] تتطلب فيروسات كورونا دقة وعملياتية مرتفعة أثناء تخليق الرنا بسبب جينومها الكبير نسبيا مقارنة بفيروسات أخرى.[66]
أحد الوظائف الرئيسية لمعقد الريبليكاز-ترانسكريبتاز هو نسخ الجينوم الفيروسي. يقوم بوليميراز الرنا المعتمد على الرنا بالتخليق المباشر لجزيئات الرنا الجينومية الفرعية سالبة الاتجاه من الرنا الجينومي موجب الاتجاه، ثم يلي ذلك نسخ جزيئات الرنا الجينومية الفرعية سالبة الاتجاه هذه إلى جزيئات رنا رسول موجبة الاتجاه مقابلة لها تنتج عنها البروتينات الفيروسية البنيوية واللابنيوية بعد الترجمة.[67]
وظيفة مهمة أخرى لمركب الريبليكاز-ترانسكريبتاز هي مضاعفة الجينوم الفيروسي، يقوم بوليميراز الرنا المعتمد على الرنا بالتخليق المباشر لسلسلة الرنا الجينومية سالبة الاتجاه من الجينوم الفيروسي موجب الاتجاه، ثم يلي ذلك مضاعفة جينوم الرنا سالب الاتجاه هذا إلى جينومات رنا موجبة الاتجاه.[67]
تصبح جينومات الرنا المضاعفة جينوماتٍ لفيروسات جديدة. مختلف جزيئات الرنا الرسول الصغيرة هي نُسخٌ من الثلث الأخير من جينوم الفيروس والتي تلي إطاري القراءة ORF1a وORF1b. تُترجم جزيئات الرنا الرسول هذه إلى أربع بروتينات بنيوية (S وE وM وN) والتي ستصبح جزءا من الفيروسات الجديدة، وإلى ثمانية بروتينات ملحقة أخرى (orf3 to orf9b) والتي تساعد الفيروس في عمله.[68]
التجميع والتحرير
تحدث ترجمة الرنا داخل الشبكة الإندوبلازمية، وتنتقل البروتينات البنيوية S وE وM على طول المسار الإفرازي إلى التكتل الوسطي لجهاز غولجي، وهناك يقوم البروتين الغشائي M بتوجيه معظم تآثرات بروتين-بروتين المطلوبة لتجميع الفيروسات بعد ارتباطها بالقفيصة المنواة.[69]
تُحرَّر فيروسات النسل (الجديدة) من الخلية المضيفة بواسطة الإخراج الخلوي عبر حويصلات إفرازية.[69]
ملاحظات
- يُستخدم المصطلحان SARSr-CoV أي فيروس كورونا المرتبط (الذي يسبب أو له صلة) بالسارس وSARS-CoV أي فيروس كورونا السارس في بعض الأحيان بشكل مترادف، خاصة قبل اكتشاف SARS-CoV-2 أي فيروس كورونا السارس 2.
مراجع
- المحرر: اللجنة الدولية لتصنيف الفيروسات — العنوان : ICTV Master Species List 2013 v2 — العمل الكامل مُتوفِّر في: http://talk.ictvonline.org/files/master-species-lists/m/msl/4911
- المحرر: اللجنة الدولية لتصنيف الفيروسات — العنوان : ICTV Master Species List 2014 v4 — العمل الكامل مُتوفِّر في: https://talk.ictvonline.org/files/master-species-lists/m/msl/5208
- "ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus"، International Committee on Taxonomy of Viruses (ICTV) (باللغة الإنجليزية)، مؤرشف من الأصل (html) في 22 فبراير 2020، اطلع عليه بتاريخ 27 يناير 2019.
- Branswell, Helen (09 نوفمبر 2015)، "SARS-like virus in bats shows potential to infect humans, study finds"، Stat News، مؤرشف من الأصل في 21 فبراير 2020، اطلع عليه بتاريخ 20 فبراير 2020.
- "Global Epidemiology of Bat Coronaviruses"، Viruses، 11 (2): 174، فبراير 2019، doi:10.3390/v11020174، PMC 6409556، PMID 30791586،
Most notably, horseshoe bats were found to be the reservoir of SARS-like CoVs, while palm civet cats are considered to be the intermediate host for SARS-CoVs [43,44,45].
- "Virus Taxonomy: 2018 Release"، International Committee on Taxonomy of Viruses (ICTV) (باللغة الإنجليزية)، أكتوبر 2018، مؤرشف من الأصل في 9 مارس 2020، اطلع عليه بتاريخ 13 يناير 2019.
- "Coronavirus genomics and bioinformatics analysis"، Viruses، 2 (8): 1804–20، أغسطس 2010، doi:10.3390/v2081803، PMC 3185738، PMID 21994708،
Figure 2. Phylogenetic analysis of RNA-dependent RNA polymerases (Pol) of coronaviruses with complete genome sequences available. The tree was constructed by the neighbor-joining method and rooted using Breda virus polyprotein.
- Coronaviridae Study Group of the International Committee on Taxonomy of Viruses (مارس 2020)، "The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2"، Nature Microbiology، doi:10.1038/s41564-020-0695-z، PMID 32123347.
- Kohen؛ Kupferschmidth (28 فبراير 2020)، "Strategies shift as coronavirus pandemic looms"، Science، 367 (6481): 962–963، doi:10.1126/science.367.6481.962، PMID 32108093.
- "Ecoepidemiology and complete genome comparison of different strains of severe acute respiratory syndrome-related Rhinolophus bat coronavirus in China reveal bats as a reservoir for acute, self-limiting infection that allows recombination events"، Journal of Virology، 84 (6): 2808–19، مارس 2010، doi:10.1128/JVI.02219-09، PMC 2826035، PMID 20071579.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - Kieny، "After Ebola, a Blueprint Emerges to Jump-Start R&D"، Scientific American Blog Network، مؤرشف من الأصل في 20 ديسمبر 2016، اطلع عليه بتاريخ 13 ديسمبر 2016.
- "LIST OF PATHOGENS"، World Health Organization، مؤرشف من الأصل في 20 ديسمبر 2016، اطلع عليه بتاريخ 13 ديسمبر 2016.
- "Global Epidemiology of Bat Coronaviruses"، Viruses، 11 (2): 174، فبراير 2019، doi:10.3390/v11020174، PMC 6409556، PMID 30791586،
See Figure 1.
- "Coronavirus genomics and bioinformatics analysis"، Viruses، 2 (8): 1804–20، أغسطس 2010، doi:10.3390/v2081803، PMC 3185738، PMID 21994708،
See Figure 1.
- "Coronavirus genomics and bioinformatics analysis"، Viruses، 2 (8): 1804–20، أغسطس 2010، doi:10.3390/v2081803، PMC 3185738، PMID 21994708،
Furthermore, subsequent phylogenetic analysis using both complete genome sequence and proteomic approaches, it was concluded that SARSr-CoV is probably an early split-off from the Betacoronavirus lineage [1]; See Figure 2.
- "Coronaviridae - Figures - Positive Sense RNA Viruses - Positive Sense RNA Viruses (2011)"، International Committee on Taxonomy of Viruses (ICTV) (باللغة الإنجليزية)، مؤرشف من الأصل في 16 يناير 2020، اطلع عليه بتاريخ 06 مارس 2020،
See Figure 2.
- "SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory"، Infection, Genetics and Evolution، 11 (7): 1690–702، أكتوبر 2011، doi:10.1016/j.meegid.2011.06.021، PMID 21763784،
Betacoronaviruses-b ancestors, meaning SARSr-CoVs ancestors, could have been historically hosted by the common ancestor of the Rhinolophidae and Hipposideridae and could have later evolved independently in the lineages leading towards Rhinolophidae and Hipposideridae betacoronaviruses.
- "Evolutionary relationships between bat coronaviruses and their hosts"، Emerging Infectious Diseases، 13 (10): 1526–32، أكتوبر 2007، doi:10.3201/eid1310.070448، PMC 2851503، PMID 18258002.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor"، Nature، 503 (7477): 535–8، نوفمبر 2013، Bibcode:2013Natur.503..535G، doi:10.1038/nature12711، PMC 5389864، PMID 24172901.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage"، Journal of Molecular Biology، 331 (5): 991–1004، أغسطس 2003، doi:10.1016/S0022-2836(03)00865-9، PMID 12927536،
The SARS-CoV genome is ∼29.7 kb long and contains 14 open reading frames (ORFs) flanked by 5′ and 3′-untranslated regions of 265 and 342 nucleotides, respectively (Figure 1).
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466.
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization"، Methods in Molecular Biology، Springer، 1282: 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466.
- "The role of severe acute respiratory syndrome (SARS)-coronavirus accessory proteins in virus pathogenesis"، Viruses، 4 (11): 2902–23، نوفمبر 2012، doi:10.3390/v4112902، PMC 3509677، PMID 23202509.
- "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage"، Journal of Molecular Biology، 331 (5): 991–1004، أغسطس 2003، doi:10.1016/S0022-2836(03)00865-9، PMID 12927536،
See Figure 1.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "The role of severe acute respiratory syndrome (SARS)-coronavirus accessory proteins in virus pathogenesis"، Viruses، 4 (11): 2902–23، نوفمبر 2012، doi:10.3390/v4112902، PMC 3509677، PMID 23202509،
See Table 1.
- "Differential stepwise evolution of SARS coronavirus functional proteins in different host species"، BMC Evolutionary Biology، 9: 52، مارس 2009، doi:10.1186/1471-2148-9-52، PMC 2676248، PMID 19261195.
- Narayanan, Krishna؛ Huang؛ Makino (أبريل 2008)، "SARS coronavirus Accessory Proteins"، Virus Research، 133 (1): 113–121، doi:10.1016/j.virusres.2007.10.009، ISSN 0168-1702، PMC 2720074، PMID 18045721،
See Table 1.
- Lu R, Zhao X, Li J, Niu P, Yang B, Wu H؛ وآخرون (2020)، "Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding."، Lancet، 395 (10224): 565–574، doi:10.1016/S0140-6736(20)30251-8، PMC 7159086، PMID 32007145.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Coronaviridae Study Group of the International Committee on Taxonomy of Viruses (2020)، "The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2."، Nat Microbiol، 5 (4): 536–544، doi:10.1038/s41564-020-0695-z، PMC 7095448، PMID 32123347.
- Kim Y, Son K, Kim YS, Lee SY, Jheong W, Oem JK (2019)، "Complete genome analysis of a SARS-like bat coronavirus identified in the Republic of Korea."، Virus Genes، 55 (4): 545–549، doi:10.1007/s11262-019-01668-w، PMC 7089380، PMID 31076983.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Li, W. (2005)، "Bats Are Natural Reservoirs of SARS-Like Coronaviruses"، Science، 310 (5748): 676–679، doi:10.1126/science.1118391، ISSN 0036-8075.
- Xing‐Yi Ge, Ben Hu, and Zheng‐Li Shi (2015)، "BAT CORONAVIRUSES"، في Lin-Fa Wang and Christopher Cowled (المحرر)، Bats and Viruses: A New Frontier of Emerging Infectious Diseases, First Edition.، John Wiley & Sons.
{{استشهاد بكتاب}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - He B, Zhang Y, Xu L, Yang W, Yang F, Feng Y؛ وآخرون (2014)، "Identification of diverse alphacoronaviruses and genomic characterization of a novel severe acute respiratory syndrome-like coronavirus from bats in China."، J Virol، 88 (12): 7070–82، doi:10.1128/JVI.00631-14، PMC 4054348، PMID 24719429.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Lau, Susanna K. P.؛ Feng, Yun؛ Chen, Honglin؛ Luk, Hayes K. H.؛ Yang, Wei-Hong؛ Li, Kenneth S. M.؛ Zhang, Yu-Zhen؛ Huang, Yi؛ وآخرون (2015)، "Severe Acute Respiratory Syndrome (SARS) Coronavirus ORF8 Protein Is Acquired from SARS-Related Coronavirus from Greater Horseshoe Bats through Recombination"، Journal of Virology، 89 (20): 10532–10547، doi:10.1128/JVI.01048-15، ISSN 0022-538X.
- Xing-Yi Ge؛ Jia-Lu Li؛ Xing-Lou Yang؛ وآخرون (2013)، "Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor"، Nature، 503 (7477): 535–8، Bibcode:2013Natur.503..535G، doi:10.1038/nature12711، PMC 5389864، PMID 24172901.
- Yang XL, Hu B, Wang B, Wang MN, Zhang Q, Zhang W؛ وآخرون (2016)، "Isolation and Characterization of a Novel Bat Coronavirus Closely Related to the Direct Progenitor of Severe Acute Respiratory Syndrome Coronavirus."، J Virol، 90 (6): 3253–6، doi:10.1128/JVI.02582-15، PMC 4810638، PMID 26719272.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Wacharapluesadee S, Tan CW, Maneeorn P, Duengkae P, Zhu F, Joyjinda Y, وآخرون (فبراير 2021)، "Evidence for SARS-CoV-2 related coronaviruses circulating in bats and pangolins in Southeast Asia"، Nature Communications، 12 (1): 972، doi:10.1038/s41467-021-21240-1، PMC 7873279، PMID 33563978.
- Hul V, Delaune D, Karlsson EA, Hassanin A, Tey PO, Baidaliuk A, وآخرون (26 يناير 2021)، "A novel SARS-CoV-2 related coronavirus in bats from Cambodia"، bioRxiv (باللغة الإنجليزية)، ص. 2021.01.26.428212، doi:10.1101/2021.01.26.428212.
- Murakami, Shin؛ Kitamura, Tomoya؛ Suzuki, Jin؛ Sato, Ryouta؛ Aoi, Toshiki؛ Fujii, Marina؛ Matsugo, Hiromichi؛ Kamiki, Haruhiko؛ Ishida, Hiroho؛ Takenaka-Uema, Akiko؛ Shimojima, Masayuki؛ Horimoto, Taisuke (2020)، "Detection and Characterization of Bat Sarbecovirus Phylogenetically Related to SARS-CoV-2, Japan"، Emerging Infectious Diseases، 26 (12): 3025–3029، doi:10.3201/eid2612.203386، ISSN 1080-6040.
- Hu, Dan؛ Zhu, Changqiang؛ Ai, Lele؛ He, Ting؛ Wang, Yi؛ Ye, Fuqiang؛ Yang, Lu؛ Ding, Chenxi؛ Zhu, Xuhui؛ Lv, Ruicheng؛ Zhu, Jin؛ Hassan, Bachar؛ Feng, Youjun؛ Tan, Weilong؛ Wang, Changjun (2018)، "Genomic characterization and infectivity of a novel SARS-like coronavirus in Chinese bats"، Emerging Microbes & Infections، 7 (1): 1–10، doi:10.1038/s41426-018-0155-5، ISSN 2222-1751.
- Lam, Tommy Tsan-Yuk؛ وآخرون (2020)، "Identifying SARS-CoV-2-related coronaviruses in Malayan pangolins"، Nature، 583 (7815): 282–285، doi:10.1038/s41586-020-2169-0، ISSN 0028-0836.
- Xiao, Kangpeng؛ Zhai, Junqiong؛ Feng, Yaoyu؛ Zhou, Niu؛ Zhang, Xu؛ Zou, Jie-Jian؛ Li, Na؛ Guo, Yaqiong؛ Li, Xiaobing؛ Shen, Xuejuan؛ Zhang, Zhipeng؛ Shu, Fanfan؛ Huang, Wanyi؛ Li, Yu؛ Zhang, Ziding؛ Chen, Rui-Ai؛ Wu, Ya-Jiang؛ Peng, Shi-Ming؛ Huang, Mian؛ Xie, Wei-Jun؛ Cai, Qin-Hui؛ Hou, Fang-Hui؛ Chen, Wu؛ Xiao, Lihua؛ Shen, Yongyi (09 يوليو 2020)، "Isolation of SARS-CoV-2-related coronavirus from Malayan pangolins"، Nature، 583 (7815): 286–289، doi:10.1038/s41586-020-2313-x.
- Zhou, Hong؛ Chen, Xing؛ Hu, Tao؛ Li, Juan؛ Song, Hao؛ Liu, Yanran؛ Wang, Peihan؛ Liu, Di؛ Yang, Jing؛ Holmes, Edward C.؛ Hughes, Alice C.؛ Bi, Yuhai؛ Shi, Weifeng (2020)، "A Novel Bat Coronavirus Closely Related to SARS-CoV-2 Contains Natural Insertions at the S1/S2 Cleavage Site of the Spike Protein"، Current Biology، 30 (11): 2196–2203.e3، doi:10.1016/j.cub.2020.05.023، ISSN 0960-9822.
- Zhou, Peng؛ Yang, Xing-Lou؛ Wang, Xian-Guang؛ Hu, Ben؛ Zhang, Lei؛ Zhang, Wei؛ Si, Hao-Rui؛ Zhu, Yan؛ وآخرون (2020)، "A pneumonia outbreak associated with a new coronavirus of probable bat origin"، Nature، 579 (7798): 270–273، doi:10.1038/s41586-020-2012-7، ISSN 0028-0836.
- Temmam, Sarah؛ Vongphayloth, Khamsing؛ Baquero, Eduard؛ Munier, Sandie؛ Bonomi, Massimiliano؛ Regnault, Béatrice؛ Douangboubpha, Bounsavane؛ Karami, Yasaman؛ Chrétien, Delphine؛ Sanamxay, Daosavanh؛ Xayaphet, Vilakhan؛ Paphaphanh, Phetphoumin؛ Lacoste, Vincent؛ Somlor, Somphavanh؛ Lakeomany, Khaithong؛ Phommavanh, Nothasin؛ Pérot, Philippe؛ Dehan, Océane؛ Amara, Faustine؛ Donati, Flora؛ Bigot, Thomas؛ Nilges, Michael؛ Rey, Félix A.؛ van der Werf, Sylvie؛ Brey, Paul T.؛ Eloit, Marc (16 فبراير 2022)، "Bat coronaviruses related to SARS-CoV-2 and infectious for human cells"، Nature، doi:10.1038/s41586-022-04532-4.
- "Ultrastructural characterization of SARS coronavirus"، Emerging Infectious Diseases، 10 (2): 320–6، فبراير 2004، doi:10.3201/eid1002.030913، PMC 3322934، PMID 15030705،
Virions acquired an envelope by budding into the cisternae and formed mostly spherical, sometimes pleomorphic, particles that averaged 78 nm in diameter (Figure 1A).
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy"، Journal of Virology، 80 (16): 7918–28، أغسطس 2006، doi:10.1128/JVI.00645-06، PMC 1563832، PMID 16873249،
Particle diameters ranged from 50 to 150 nm, excluding the spikes, with mean particle diameters of 82 to 94 nm; Also See Figure 1 for double shell.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "The molecular biology of coronaviruses"، Advances in Virus Research، 48: 1–100، 1997، doi:10.1016/S0065-3527(08)60286-9، ISBN 9780120398485، PMID 9233431.
- The molecular biology of coronaviruses، Advances in Virus Research، Academic Press، ج. 66، 01 يناير 2006، ص. 193–292، doi:10.1016/S0065-3527(06)66005-3، ISBN 9780120398690، PMID 16877062،
Nevertheless, the interaction between S protein and receptor remains the principal, if not sole, determinant of coronavirus host species range and tissue tropism.
- "Origin and evolution of pathogenic coronaviruses"، Nature Reviews. Microbiology، 17 (3): 181–192، مارس 2019، doi:10.1038/s41579-018-0118-9، PMID 30531947،
Different SARS-CoV strains isolated from several hosts vary in their binding affinities for human ACE2 and consequently in their infectivity of human cells76,78 (Fig. 6b)
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization"، Methods in Molecular Biology، Springer، 1282: 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See section: Virion Structure.
- "The SARS coronavirus nucleocapsid protein--forms and functions"، Antiviral Research، 103: 39–50، مارس 2014، doi:10.1016/j.antiviral.2013.12.009، PMID 24418573،
See Figure 4c.
- "A structural analysis of M protein in coronavirus assembly and morphology"، Journal of Structural Biology، 174 (1): 11–22، أبريل 2011، doi:10.1016/j.jsb.2010.11.021، PMC 4486061، PMID 21130884،
See Figure 10.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "Structures and Functions of Coronavirus Proteins: Molecular Modeling of Viral Nucleoprotein." (PDF)، مؤرشف من الأصل (PDF) في 18 فبراير 2020.
- Lal, Sunil K, المحرر (2010)، Molecular Biology of the SARS-Coronavirus، doi:10.1007/978-3-642-03683-5، ISBN 978-3-642-03682-8.
- "Structural genomics and interactomics of 2019 Wuhan novel coronavirus, 2019-nCoV, indicate evolutionary conserved functional regions of viral proteins."، bioRxiv، يناير 2020، doi:10.1101/2020.02.10.942136.
- "Complete genome characterisation of a novel coronavirus associated with severe human respiratory disease in Wuhan, China."، bioRxiv، يناير 2020، doi:10.1101/2020.01.24.919183.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See section: Coronavirus Life Cycle – Attachment and Entry
- "Proteolytic activation of the SARS-coronavirus spike protein: cutting enzymes at the cutting edge of antiviral research"، Antiviral Research، 100 (3): 605–14، ديسمبر 2013، doi:10.1016/j.antiviral.2013.09.028، PMC 3889862، PMID 24121034،
See Figure 2.
- "TMPRSS2 and ADAM17 cleave ACE2 differentially and only proteolysis by TMPRSS2 augments entry driven by the severe acute respiratory syndrome coronavirus spike protein"، Journal of Virology، 88 (2): 1293–307، يناير 2014، doi:10.1128/JVI.02202-13، PMC 3911672، PMID 24227843،
The SARS-CoV can hijack two cellular proteolytic systems to ensure the adequate processing of its S protein. Cleavage of SARS-S can be facilitated by cathepsin L, a pH-dependent endo-/lysosomal host cell protease, upon uptake of virions into target cell endosomes (25). Alternatively, the type II transmembrane serine proteases (TTSPs) TMPRSS2 and HAT can activate SARS-S, presumably by cleavage of SARS-S at or close to the cell surface, and activation of SARS-S by TMPRSS2 allows for cathepsin L-independent cellular entry (26,–28).
- "Coronaviruses - drug discovery and therapeutic options"، Nature Reviews. Drug Discovery، 15 (5): 327–47، مايو 2016، doi:10.1038/nrd.2015.37، PMID 26868298،
S is activated and cleaved into the S1 and S2 subunits by other host proteases, such as transmembrane protease serine 2 (TMPRSS2) and TMPRSS11D, which enables cell surface non-endosomal virus entry at the plasma membrane.
- "The human coronavirus HCoV-229E S-protein structure and receptor binding"، eLife، 8، أكتوبر 2019، doi:10.7554/eLife.51230، PMC 6970540، PMID 31650956.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See Table 2.
- "The molecular biology of coronaviruses"، Advances in Virus Research، Academic Press، 66: 193–292، 01 يناير 2006، doi:10.1016/S0065-3527(06)66005-3، ISBN 9780120398690، PMID 16877062،
See Figure 8.
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See section: Replicase Protein Expression
- "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens"، Journal of Virology، 90 (16): 7415–28، أغسطس 2016، doi:10.1128/JVI.00080-16، PMC 4984655، PMID 27279608،
Finally, these results, combined with those from previous work (33, 44), suggest that CoVs encode at least three proteins involved in fidelity (nsp12-RdRp, nsp14-ExoN, and nsp10), supporting the assembly of a multiprotein replicase-fidelity complex, as described previously (38).
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See section: Corona Life Cycle – Replication and Transcription
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See Figure 1.
- Maier, Helena Jane؛ Bickerton, Erica؛ Britton, Paul, المحررون (2015)، "Coronaviruses: an overview of their replication and pathogenesis"، Coronaviruses، Methods in Molecular Biology، Springer، ج. 1282، ص. 1–23، doi:10.1007/978-1-4939-2438-7_1، ISBN 978-1-4939-2438-7، PMC 4369385، PMID 25720466،
See section: Coronavirus Life Cycle – Assembly and Release
قراءة متعمقة
- "Coronavirus as a possible cause of severe acute respiratory syndrome"، Lancet، 361 (9366): 1319–25، أبريل 2003، doi:10.1016/s0140-6736(03)13077-2، PMID 12711465.
- "Characterization of a novel coronavirus associated with severe acute respiratory syndrome"، Science، 300 (5624): 1394–9، مايو 2003، Bibcode:2003Sci...300.1394R، doi:10.1126/science.1085952، PMID 12730500.
- "The Genome sequence of the SARS-associated coronavirus"، Science، 300 (5624): 1399–404، مايو 2003، Bibcode:2003Sci...300.1399M، doi:10.1126/science.1085953، PMID 12730501.
- "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage"، Journal of Molecular Biology، 331 (5): 991–1004، أغسطس 2003، CiteSeerX 10.1.1.319.7007، doi:10.1016/S0022-2836(03)00865-9، PMID 12927536.
- "Rewiring the severe acute respiratory syndrome coronavirus (SARS-CoV) transcription circuit: engineering a recombination-resistant genome"، Proceedings of the National Academy of Sciences of the United States of America، 103 (33): 12546–51، أغسطس 2006، Bibcode:2006PNAS..10312546Y، doi:10.1073/pnas.0605438103، PMC 1531645، PMID 16891412.
- Coronaviruses: Molecular and Cellular Biology (ط. 1st)، Caister Academic Press، 2007، ISBN 978-1-904455-16-5.
- "Coronavirus Replication and Interaction with Host"، Animal Viruses: Molecular Biology، Caister Academic Press، 2008، ISBN 978-1-904455-22-6.
- بوابة طب
- بوابة علم الفيروسات
- بوابة كوفيد-19