Centrómero
En genética, el centrómero es la constricción primaria que, utilizando tinciones tradicionales, aparece menos teñida que el resto del cromosoma. Es la zona por la que el cromosoma interacciona con los microtúbulos del huso acromático desde profase hasta anafase, tanto en mitosis como en meiosis, y es responsable de realizar y regular los movimientos cromosómicos que tienen lugar durante estas fases. Además, el centrómero contribuye a la nucleación de la cohesión de las cromátidas hermanas para garantizar una distribución correcta del material genético a las células hijas durante el proceso de división celular, lo que los hace esencial para la estabilidad del genoma, la fertilidad y el desarrollo saludable[1]. En la estructura del centrómero intervienen tanto el ADN centromérico como proteínas centroméricas.
En la levadura de gemación (Saccharomyces cerevisiae) el ADN centromérico consta únicamente de 125 pb y está conservado entre los diferentes cromosomas.[2] Sin embargo, el ADN centromérico en metazoos puede constar de megabases, y no contiene secuencias consenso fácilmente identificables (ver la revisión de Choo en 1997[3]). Hasta hace poco, los estudios del genoma humano excluían las secuencias repetitivas dentro y cerca de los centrómeros, lo que limita el entendimiento de los mismos. Los nuevos avances nos han permitido caracterizar estas secuencias, cuales constituyen 189.9 megabases de nuestro genoma[1]. A pesar de las diferencias entre el ADN centromérico de levaduras y metazoos, el cinetocoro se ensambla en ambos casos sobre nucleosomas centroméricos que contienen una forma especializada de histona H3 (Cse4p en levaduras[4] o su homólogo CENP-A en metazoos).
El ADN centromérico se organiza en forma de heterocromatina constitutiva, que permanece condensada en casi todas las células somáticas de un organismo. Estas regiones son pobres en genes y pueden inducir la represión de la expresión génica de las regiones adyacentes de manera epigenética. Este fenómeno se denomina "variegación por efecto de posición" (PEV, por Position Effect Variegation).[5] La aparición ocasional de centrómeros de novo (neocentrómeros) sugiere que más que la secuencia del ADN per se, la característica primaria de los centrómeros es la organización estructural de los dominios centroméricos. La selección del centrómero puede ser también el resultado de un complejo número de parámetros, como el momento de su replicación, la posición dentro del núcleo celular, así como otras características heredables de la estructura de la cromatina.
El centrómero tiene un comportamiento diferente durante la anafase mitótica y la anafase-I de la meiosis, de manera que durante la anafase mitótica las cromátidas hermanas se separan a polos opuestos (segregación anfitélica) mientras que en la anafase-I de la meiosis lo que se separa a polos opuestos son los cromosomas homólogos completos, cada uno constituido por dos cromátidas (segregación sintélica).
Posición del centrómero
Cada cromosoma posee dos brazos, uno largo (llamado q) y otro corto (llamado p) separados por el centrómero, los cuales se conectan de forma metacéntrica, submetacéntrica, acrocéntrica, holocéntrica o telocéntrica.
Metacéntrico
Un cromosoma metacéntrico es un cromosoma cuyo centrómero se encuentra en la mitad del cromosoma, dando lugar a brazos de igual longitud.
Cuatro pares de los cromosomas humanos poseen una estructura metacéntrica, el 1, el 3, el 19 y el 20.
Submetacéntrico
Un cromosoma submetacéntrico es un cromosoma en el cual el centrómero se ubica de tal manera que un brazo es ligeramente más corto que el otro.
La mayor parte de los cromosomas humanos son submetacéntricos excepto los cromosomas 1, 3, 19 y el 20 que son metacéntricos; y 13, 14, 15, 21 y 22 que son acrocéntricos. Además, el cromosoma Y a veces es considerado submetacéntrico aunque otros lo describen como acrocéntrico sin satélite.
Acrocéntrico
Un cromosoma acrocéntrico es un cromosoma en el que el centrómero se encuentra más cercano a uno de los telómeros, dando como resultado un brazo muy corto (p) y el otro largo (q).
De los 23 pares de cromosomas humanos el cromosoma 13, el 14, el 15, el 21 y el 22 son acrocéntricos y actúan como organizadores nucleolares.
Telocéntrico
Aun cuando el concepto es ampliamente aceptado y distribuido entre la comunidad científica,[6] realmente, un cromosoma telocéntrico como tal no existe. Supuestamente en este tipo de cromosomas el centrómero está localizado en un extremo del mismo, pero la región telocéntrica no permite que molecularmente haya otra estructura finalizando al cromosoma. De hecho, el acortamiento del telomero o su ausencia total causa inestabilidad en los cromosomas y la consecuente Translocación robertsoniana.[7] Por tanto, el término telocéntrico es incorrecto y debe considerarse el término subtelocéntrico, el cual implica que el telómero se ubica al final así no sea visible y que el centrómero esta después invariablemente.
Ninguno de los cromosomas humanos presenta esta característica; pero, por ejemplo, los 40 cromosomas del ratón común son subtelocéntricos.
El centrómero en Saccharomyces cerevisiae
Los análisis llevados a cabo en S. cerevisiae para aislar el ADN centromérico (ADN CEN) de todos sus cromosomas, han establecido que en todos los centrómeros de levaduras estudiados existen tres regiones muy conservadas:
- La región I (CDEI), secuencia de 8-9 pb (PuTCACPuTG, donde "Pu" simboliza cualquier base púrica, o sea, adenina o guanina, "T" es timina, "C" es citosina, "A" es adenina y "G" es guanina) que se encuentra en el límite izquierdo de todos los centrómeros.
- La región II (CDEII), secuencia de 76-86 pb rica en pares AT (87-95%) que separa las regiones I y II.
- La región III (CDEIII), secuencia de 25 pb altamente conservada en el extremo derecho de los centrómeros. Presenta una secuencia palindrómica TGTTT(T/A)TGNTTTCCGAAANNNAAAA.
En la cromatina en su forma nativa, el ADN CEN es un segmento de 220-250 pb protegido de la acción de las nucleasas y flanqueado en ambos extremos por sitios hipersensibles al corte y un conjunto de nucleosomas altamente organizados, que contienen la histona especializada Cse4p (el homólogo en levaduras de CENP-A) en lugar de H3.
Las mutaciones en las regiones I y II reducen pero no inactivan la función del centrómero, mientras que las que ocurren en la región III lo inactivan completamente. En los mutantes por deleción de la región CDEII, la función centromérica puede restablecerse casi completamente insertando una secuencia de ADN compuesta solo de A-T al azar y de tamaño equivalente.[8] Por tanto, en esta región los factores críticos para una segregación cromosómica correcta son el contenido en A-T y la longitud del ADN, más que la secuencia de nucleótidos, quizás porque influyen en la conformación del ADN centromérico.
CDEIII es la zona de unión del complejo proteico CBF3 (por Centromere Binding Factor 3), compuesto por las proteínas Ndc10p, Cep3p, Ctf13p, y Skp1p[9] (esta proteína - también denominada p23SKP1- es además parte del complejo SCULCDC4 implicado en los procesos de degradación mediados por ubiquitina necesarios para la progresión a través del ciclo celular). En ausencia de CBF3, el cinetocoro no es funcional, tanto in vivo como in vitro,[10][11] y todas las proteínas del cinetocoro conocidas, incluida Cse4p, presentan alteración de la asociación con el centrómero.[12] Sin embargo, la unión de CBF3 al ADN centromérico in vivo no requiere Cse4p.[13] Por tanto, la unión específica de CBF3 a la región CDEIII participa en la definición de la localización del cinetocoro en levaduras. Otra proteína esencial para la viabilidad de las células de levaduras es CBF5p, que co-purifica con el complejo CBF3 en condiciones de baja astringencia.[14] Esta proteína también se une a microtúbulos in vitro, y parece ser importante en la transición G1/S del ciclo celular, ya que la eliminación de CBF5p bloquea la división celular antes de que ocurra la replicación del ADN.
Por otro lado, CDEI es el sitio de unión de un homodímero de Cbf1p.[15] Cbf1p no es esencial para la función del cinetocoro, pero induce el plegado del ADN[16] y por tanto puede contribuir a la estructura de nivel superior del centrómero. Cbf1p tiene similitud estructural e identidad de secuencia limitada a CENP-B, que se une al ADN centromérico de metazoos y también induce el plegado del ADN.[17]
Mif2p es una proteína esencial en levaduras, similar a CENP-C de metazoos, cuya eliminación produce fallos en la segregación cromosómica, retraso en mitosis y microtúbulos con morfología aberrante.[18] MIF2 interacciona genéticamente con tres de los genes que codifican proteínas centroméricas: CBF1, CBF3a y CBF3b. Aunque la unión de Mif2 al centrómero depende de Ndc10,[19] su localización en el ADN CEN está muy disminuida en mutantes para Cse4.[20] Mif2 presenta un dominio acídico y otro dominio rico en prolina, lo que se denomina un "gancho AT" (AT hook), un motivo que es común a las proteínas que se unen a secuencias de ADN ricas en AT (como las proteínas HMGI(Y) de mamíferos), lo que sugiere que Mif2 se une a la región CDEII.[21]
En S. cerevisiae, el ADN centromérico se replica en una etapa temprana de la fase S,[22] tal vez porque es necesario replicar el ADN centromérico para iniciar el ensamblaje de los cinetocoros hermanos.
El centrómero en la levadura de fisión
El ADN centromérico de Schizosaccharomyces pombe es considerablemente más complejo que el de S. cerevisiae y comparte algunas propiedades con los centrómeros regionales de los eucariotas superiores.[23] El centrómero de S. pombe está constituido por 40-100 kb de ADN organizado en distintos tipos de repeticiones específicas de los centrómeros (las repeticiones tipo K), que están a su vez organizadas en una gran repetición invertida.[24] Como ocurre en eucariotas superiores, la organización de los centrómeros en la levadura de fisión varía considerablemente entre diferentes cromosomas y entre cepas muy próximas.[25] El centro de la repetición invertida, el núcleo central, contiene una secuencia de 4-7 kb que es fundamental para la función centromérica. Las regiones centrales del centrómero de S. pombe se organizan en una estructura inusual, que depende de la existencia de un elemento dentro de las repeticiones tipo K (denominado enhancer centromérico) y que es fundamental para la función del centrómero.[26] Las regiones K y el núcleo central son las dianas de los mecanismos epigenéticos que afectan a la función del centrómero in vivo. Por otro lado, existe redundancia funcional tanto entre las repeticiones tipo K como en el núcleo central.
La función precisa de las secuencias repetidas de ADN en el centrómero no está muy clara, pero probablemente tienen una función estructural en el apareamiento y la segregación cromosómica (revisado por Karpen y Allshire en 1997[27]), y solo indirectamente producen silenciamiento transcripcional. Las secuencias repetidas más externas de los centrómeros de Schizosaccharomyces pombe son heterocromáticas y son necesarias para el ensamblaje de un centrómero activo. A partir de estas secuencias repetidas se generan tránscritos que son procesados por los componentes de la maquinaria de RNAi y medían el silenciamiento de la cromatina.[28] (Véase también Ensamblaje de heterocromatina mediante RNAi en S. pombe).
Centrómeros en metazoos
La determinación de los centrómeros de metazoos constituye una tarea difícil y esquiva. En animales y plantas, los centrómeros están incluidos en regiones de ADN satélite altamente repetido, que resulta difícil de analizar incluso con los métodos de mapeo más potentes. Estas regiones de ADN satélite están embebidas en regiones de heterocromatina constitutiva, que se mantiene silenciada en la mayor parte de las células somáticas de un organismo. La ausencia mayoritaria de genes activos en las regiones centroméricas es una característica que parece que se ha adquirido progresivamente a través de la evolución.[29]
ADN satélite
En Drosophila melanogaster la secuencia AATAACATAG está repetida en tándem en las regiones próximas al centrómero. Dado que estas secuencias cortas de 10 pb no son representativas del genoma de una especie, suele suceder que su contenido en G+C es diferente al contenido en G+C del resto del genoma. Esto hace que cuando el ADN de una especie eucarionte se centrifuga en gradiente de densidad de cloruro de cesio, aparezca una banda principal que contiene la mayor parte del ADN de la especie y una banda satélite (minoritaria) que está formada por una secuencia corta de ADN repetida en tándem. El ADN satélite en algunas especies tiene mayor densidad que el ADN principal (mayor contenido en G+C) y en otras especies tiene menor densidad y, por tanto, menor contenido en G+C que el ADN principal. Cuando el ADN satélite de ratón se marca radiactivamente y se realiza una hibridación in situ con el ADN de cromosomas metafásicos mitóticos, se observa que el marcaje radiactivo (hibridación) se produce en regiones próximas al centrómero. El ADN satélite también se denomina α-satélite y es uno de los componentes del genoma eucariótico que evoluciona más rápidamente.[30]
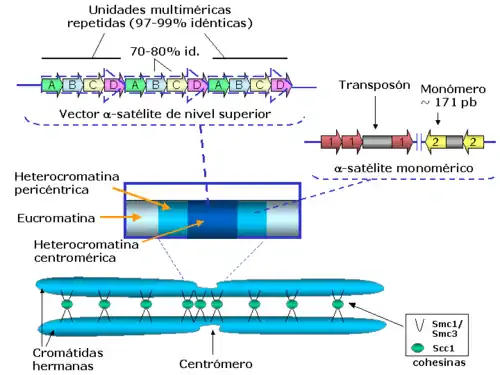
A pesar de que en la mayor parte de los casos no se han detectado motivos especialmente definidos, una secuencia candidata en el ADN satélite tendría que estar repetida en cientos de kilobases, ya que éste es el tamaño mínimo de un centrómero funcional que se ha identificado en diferentes organismos. Por ejemplo, en Drosophila la unidad mínima necesaria de repeticiones en tándem es de 420 kb, en maíz se necesitan 500 kb y en humanos la unidad mínima consiste de 100 kb. Una característica interesante de la mayor parte del ADN satélite es su unidad de longitud, ya que aunque no se han detectado secuencias con motivos conservados, la longitud de la unidad que se repite es muy parecida entre organismos. En primates, por ejemplo, la unidad básica que se repite tiene 171 pb, en el pez Sparus aurata la repetición centromérica tiene 186 pb, en el insecto Chironomus pallidivittatus tiene 155 pb, en Arabidopsis thaliana y en el maíz tiene 180 pb, y en el arroz 168 pb.[32] La estrecha variación en longitud de la unidad que se repite en el ADN satélite corresponde aproximadamente al rango de longitud del ADN que rodea a un nucleosoma, y repeticiones más largas, como las que se encuentran en los centrómeros del cerdo (340 pb) corresponden aproximadamente a la longitud de dos nucleosomas. Hay excepciones notorias, como las repeticiones pentaméricas que se encuentran en Drosophila melanogaster. En general, la selección de la longitud de un nucleosoma podría limitar la evolución del ADN centromérico, de acuerdo con su función estructural en el genoma.
Estas unidades mínimas (denominadas monómeros) se encuentran normalmente asociadas de forma cabeza-cola. En las regiones centroméricas del núcleo funcional, el ADN satélite se organiza en una unidad repetida que consta de múltiples monómeros. La unidad multimonomérica se repite a su vez muchas veces, generando un vector (array) de nivel superior. Los vectores de nivel superior de ADN satélite son la organización típica de las regiones centroméricas humanas y se extienden a través de megabases de ADN que se encuentran mayoritariamente ininterrumpidos por ningún tipo de inserción o mutación. Por tanto en animales y plantas se presentan centrómeros "regionales", frente a los centrómeros "puntuales" que se encuentran en levaduras.
Sin embargo, a pesar de que el ADN satélite se encuentra en los centrómeros humanos nativos, se han detectado centrómeros humanos generados de novo (neocentrómeros) que carecen de α-satélite u otras repeticiones en tándem,[33] lo que indica que el ADN satélite no es fundamental para definir un centrómero funcional.
Proteínas centroméricas
Una característica conservada y heredable de los centrómeros es la presencia en sus nucleosomas de una variante especial de la histona H3, que se encuentra únicamente en el núcleo de la región centromérica. Esta histona específica de los centrómeros se denomina CENP-A en mamíferos (centromeric protein A), Cid (centromere identifier) en Drosophila, Cse4 en S. cerevisiae y Cnp1 en S. pombe (revisado por Choo en 2001[34]). La presencia de esta variante de la histona H3 parece ser fundamental para el ensamblaje del cinetocoro y distingue la placa interna del cinetocoro de la heterocromatina pericéntrica, que contiene la histona H3 normal. CENP-A presenta algunas características que la diferencian de la histona H3 normal, como una cola NH2-terminal no canónica, un plegamiento divergente y una región lazo 1 más largo.[35] Aunque la histona H3 está sometida a una fuerte selección evolutiva, las histonas centroméricas son sorprendentemente divergentes. Esta diferencia podría deberse a la necesidad de H3 de interaccionar con todo el genoma, mientras que la variante centromérica solo necesita interaccionar con el ADN centromérico correspondiente. Este ADN está formado por ADN satélite, que es uno de los componentes del genoma eucariótico que evoluciona más rápidamente.[30] Se ha propuesto que la interacción entre la histona centromérica y el ADN centromérico es responsable de la longitud similar a la que rodea un nucleosoma de las repeticiones del ADN satélite.[32]
Un estudio realizado en Drosophila identificó que las regiones centrales de los centrómeros se replican como dominios aislados en estadios tempranos de la fase S, antes de la replicación de la heterocromatina pericéntrica, que se replica de forma tardía.[36] Si en el momento que se replican los centrómeros, la región del núcleo en la que se localizan los centrómeros excluye la histona H3 pero secuestra la histona centromérica, la compartimentalización aseguraría que solo CENP-A esté disponible para el ensamblaje de la cromatina centromérica.[32] Este modelo se apoya en varias líneas de evidencias en diferentes organismos.
Además de CENP-A, se han identificado otros componentes constitutivos en el centrómero. Uno de ellos es CENP-C, que está conservado evolutivamente, aunque solo comparte un motivo de 20 aminoácidos con su homólogo en S. cerevisiae, Mif2. También se han encontrado homólogos en otras especies, como HCP-4 en C. elegans. La localización centromérica de CENP-C depende de CENP-A y se ha sugerido que CENP-C podría interaccionar con la estructura de la cromatina alterada por CENP-A. Sin embargo, aunque la zona de unión de CENP-C al ADN se ha mapeado en la zona central de la proteína, no se ha identificado una secuencia específica de unión. Parece además que CENP-C presenta la capacidad de unirse a ARN de forma específica,[37] aunque la contribución de estas capacidades a la localización de CENP-C no está clara.[31]
CENP-B es la única proteína centromérica que se une a una secuencia de ADN específica de 17 pb (la "caja CENP-B"), que se encuentra en un subconjunto de monómeros de α-satélite. La función de CENP-B en los centrómeros no está clara, ya que a diferencia con CENP-A o CENP-C, CENP-B no es esencial para la función mitótica (de hecho, el ratón knockout para CENP-B es viable). CENP-B puede estar presente tanto en centrómeros activos como inactivos, lo que sugiere que no está asociada simplemente a la función centromérica.[31] Además, algunos centrómeros funcionales carecen de cajas CENP-B (como el centrómero del cromosoma Y, por ejemplo). Sin embargo, se ha demostrado que la unión de CENP-B aumenta la eficiencia de la unión de CENP-A en cromosomas humanos artificiales. A pesar de no ser esencial en humanos, CENP-B está conservada a través de varios phyla y en S. pombe se encuentran tres homólogos que sí son esenciales para la viabilidad celular.
Estas tres proteínas centroméricas están organizadas de forma diferente en el centrómero humano. CENP-B está presente en todo el vector de nivel superior, mientras que CENP-A y CENP-C se encuentran solo en algunos bloques de unidades repetidas, intercalados con bloques que contienen nucleosomas canónicos (que incluyen histona H3) y con modificaciones de histonas más características de eucromatina que de heterocromatina. Se cree que esos bloques de CENP-A se auto-organizan para presentar una "superficie" combinada que organiza el resto de las proteínas del cinetocoro, que servirá como sitio de anclaje de los microtúbulos.[31] Se considera además que los monómeros pericéntricos que flanquean las repeticiones centroméricas están generalmente desprovistos de proteínas centroméricas, y están empaquetados en nucleosomas canónicos que poseen modificaciones de histonas características de heterocromatina, y unidos a proteínas específicas de heterocromatina como HP1. Esta heterocromatina pericéntrica es importante tanto para definir los límites de los dominios centroméricos como para reclutar las cohesinas que mantendrán unidas las cromátidas hermanas hasta la anafase durante el ciclo celular.
ADN CEN y evolución
El proceso de segregación cromosómica está sometida a una fuerte presión evolutiva, dado que la pérdida o ganancia de cromosomas (una situación denominada aneuploidía) puede producir importantes alteraciones fenotípicas, como el síndrome de Down en humanos, por ejemplo. Por ello, la maquinaria encargada de distribuir los cromosomas entre las células hijas durante la división celular presenta una gran sofisticación y está sometida a un estricto control (véase checkpoint de mitosis). Los centrómeros son las regiones cromosómicas sobre las que se ensamblan los cinetocoros, que son las estructuras proteicas responsables del anclaje de los cromosomas al huso mitótico, y por ello la zona responsable del movimiento cromosómico y su regulación. Sin embargo, a pesar de ello las secuencias de ADN que definen las secuencias centroméricas están muy poco conservadas y evolucionan rápidamente incluso entre especies muy relacionadas. Esto no quiere decir que las secuencias de ADN del centrómero son hipermutables, sino que las variantes de las secuencias se fijan por expansión y contracción, y pueden aparecer de novo en sitios nuevos (neocentrómeros). Estos cambios en el ADN centromérico tienen lugar debido a la existencia de diferentes procesos mutacionales, como errores en la replicación del ADN, intercambio desequilibrado, transposición y escisión. Las proteínas centroméricas también presentan signos inesperados de una rápida evolución. Por todo ello, se ha sugerido que en el núcleo de esta rápida evolución existe un conflicto genético en funcionamiento.[32]
Parece ser que la arquitectura en un vector (array) de nivel superior que se observa en los centrómeros de humanos podría haber aparecido recientemente en un centrómero en la evolución de los primates (alrededor de la separación gorila-orangután) y se extendió a los otros cromosomas vía transposición.[38] Posteriormente, los intercambios desiguales o conversiones génicas amplificaron los vectores de nivel superior, dando lugar a la arquitectura en vectores centroméricos de nivel superior que es específica de la especie humana y que se observa en diferentes cromosomas humanos. Además, se han generado algunas variantes por mutación que se han fijado en algunos centrómeros.[31] La comparación de unidades monoméricas y unidades vectoriales de nivel superior que se encuentran en los centrómeros de cromosomas ortólogos (por ejemplo, entre chimpancés y humanos) ha llevado al descubrimiento sorprendente de que los vectores centroméricos de diferentes especies son más divergentes entre sí que las unidades pericéntricas.[39] Esta observación es anti-intuitiva, porque el vector de ADN satélite centromérico es el centrómero funcional y está sometido a una fuerte presión selectiva, mientras que las regiones de heterocromatina pericéntrica no lo están. Por tanto, la observación es paradójica: las unidades de ADN satélite que están fuertemente limitadas dentro de una especie han evolucionado rápidamente entre especies.
Esta paradoja ha llevado a pensar que alguna fuerza selectiva debe dirigir la rápida fijación de las mutaciones en los vectores centroméricos, imponiendo un sesgo para mantener las mutaciones, incrementando de esta forma las tasas de mutación del vector completo. Se ha sugerido que esta fuerza selectiva puede ser la ventaja conferida a los centrómeros durante la meiosis femenina, o "deriva-centromérica":[32] nuevas variaciones en la secuencia de α-satélite, una nueva organización o simplemente un incremento en la cantidad de α-satélite proporciona una mayor oportunidad de incorporación de CENP-A y por tanto una mayor capacidad para la unión de microtúbulos. La asimetría de la tétrada meiótica femenina proporciona una oportunidad para los cromosomas de competir por ser incluido en el núcleo del ovocito mediante una orientación favorable durante la meiosis. Los centrómeros que aprovechan esta oportunidad en meiosis I "ganan", y una ligera ventaja en cada meiosis femenina es suficiente para fijar una variación centromérica favorable.[40]
Como contrapartida, mientras que la deriva centromérica puede generar una ventaja selectiva en la meiosis femenina, puede producir defectos en la meiosis masculina, pues en este caso un centrómero mutado se apareará con otro normal, generándose una diferencia de tensión que puede activar el checkpoint de mitosis, provocando la muerte celular y con ello una disminución de la fertilidad masculina. Una forma de contrarrestar este efecto en la meiosis masculina sería la aparición de mutaciones en las proteínas centroméricas con alteración en su capacidad de unión al ADN y que equilibraran la tensión centromérica. La proteína candidata más probable es CENP-A, la cual está presente de manera constitutiva en los centrómeros y, frecuentemente, se acompaña de modificaciones asociadas a la "centrocromatina" en las histonas canónicas[1].
Si este proceso tiene lugar en dos poblaciones aisladas de la misma especie, las configuraciones del ADN satélite y CENP-A divergirán rápidamente. En cada población, CENP-A evolucionará para suprimir los efectos deletéreos de la evolución del ADN satélite. De esta forma, las nuevas variantes de CENP-A resultarán incompatibles con el ADN satélite de la otra población. Cruces entre ambas poblaciones resultarán en defectos en los híbridos. Por tanto, el proceso evolutivo entre CENP-A y el ADN satélite da lugar al inicio del aislamiento reproductivo entre las dos poblaciones (véase también Mecanismos de aislamiento reproductivo). Esto quiere decir que la evolución centromérica tiene como consecuencia inevitable la especiación.[32]
Por otra parte, gracias a comparaciones de los centrómeros en los cromosomas X, se han visto variaciones epigenéticas y estructurales en estas regiones. Además, un reciente ensamblaje del genoma humano ha detectado un posible mecanismo de como las estructuras pericentroméricas y centroméricas evolucionan, a través de un modelo de expación en capas para las secuencias αSat. Este modelo propone que diferentes repeticiones de secuencia αSat emergen periódicamente y se expanden dentro de un vector activo, desplazando a un lado las secuencias antiguas, y convirtiéndose en el sitio de ensamblaje del cinetocoro. Cabe destacar que las αSat se pueden originar del mismo, o de diferentes vectores. A medida que este proceso se repite con el tiempo, las capasque flanquean el centrómero activo se encogen y se deterioran. Este proceso, levanta interrogantes sobre la relación entre este dinámico proceso evolutivo, y la posición del centrómero[1].
Referencias
- Altemose, Nicolas; Logsdon, Glennis A.; Bzikadze, Andrey V.; Sidhwani, Pragya; Langley, Sasha A.; Caldas, Gina V.; Hoyt, Savannah J.; Uralsky, Lev et al. (2022-04). «Complete genomic and epigenetic maps of human centromeres». Science (en inglés) 376 (6588): eabl4178. ISSN 0036-8075. PMC 9233505. PMID 35357911. doi:10.1126/science.abl4178. Consultado el 15 de enero de 2023.
- Fitzgerald-hayes, M.; Clarke, L.; Carbon, J. (1982), «Nucleotide sequence comparisons and functional analysis of yeast centromere DNAs», Cell 29 (1): 235–44, doi:10.1016/0092-8674(82)90108-8.
- Choo, K.H.A. (1997), The centromere.
- Meluh, P.B.; Yang, P.; Glowczewski, L.; Koshland, D.; Smith, M.M. (1998), «Cse 4 P is a Component of the Core Centromere of Saccharomyces cerevisiae», Cell(Cambridge) 94 (5): 607–613, archivado desde el original el 1 de mayo de 2008, consultado el 28 de noviembre de 2008.
- Perrod, S.; Gasser, S.M. (2003), «Long-range silencing and position effects at telomeres and centromeres: parallels and differences», Cellular and Molecular Life Sciences (CMLS) 60 (11): 2303–2318, doi:10.1007/s00018-003-3246-x, archivado desde el original el 5 de febrero de 2009.
- "On the origin of telocentric chromosomes in mammals" Original Research Article. Hirotami T. Imai. Journal of Theoretical Biology, Volume 71, Issue 4, 20 April 1978, Pages 619-637
- "Telomere length and telomere-centromere relationships?" Original Research Article. P. Slijepcevic. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, Volume 404, Issues 1-2, 3 August 1998, Pages 215-220
- Gaudet, A.M.; Fitzgerald-hayes, M. (1990), The eukaryotic nucleus. Molecular biochemistry and macromolecular assemblies. Telford Press, Caldwell, New Jersey: 845–881.
- Lechner, J.; Carbon, J. (1991), «A 240 Kd Multisubunit Protein Complex, Cbf 3, is a Major Component of the Budding Yeast Centromere», Cell(Cambridge) 64 (4): 717–725, archivado desde el original el 1 de mayo de 2008, consultado el 28 de noviembre de 2008.
- Goh, P.Y.; Kilmartin, J.V. (1993), «NDC10: a gene involved in chromosome segregation in Saccharomyces cerevisiae», J Cell Biol 121 (3): 503–12, doi:10.1083/jcb.121.3.503.
- Sorger, P.K.; Doheny, K.F.; Hieter, P.; Kopski, K.M.; Huffaker, T.C.; Hyman, A.A. (1995), «Two Genes Required for the Binding of an Essential Saccharomyces cerevisiae Kinetochore Complex to …», Proceedings of the National Academy of Sciences 92 (26): 12026–12030.
- Ortiz, J.; Stemmann, O.; Rank, S.; Lechner, J. (1999), «A putative protein complex consisting of Ctf19, Mcm21, and Okp1 represents a missing link in the …», Genes & Development 13 (9): 1140–1155, doi:10.1101/gad.13.9.1140.
- Measday, V.; Hailey, D.W.; Pot, I.; Givan, S.A.; Hyland, K.M.; Cagney, G.; Fields, S.; Davis, T.N. et al. (2002), «Ctf3p, the Mis6 budding yeast homolog, interacts with Mcm22p and Mcm16p at the yeast outer …», Genes & Development 16 (1): 101–113, doi:10.1101/gad.949302 .
- Jiang, W.; Middleton, K.; Yoon, H.J.; Fouquet, C.; Carbon, J. (1993), «An essential yeast protein, CBF5p, binds in vitro to centromeres and microtubules», Molecular and Cellular Biology 13 (8): 4884–4893.
- Mellor, J.; Jiang, W.; Funk, M.; Rathjen, J.; Barnes, C.A.; Hinz, T.; Hegemann, J.H.; Philippsen, P. (1990), «CPF1, a yeast protein which functions in centromeres and promoters», The EMBO Journal 9 (12): 4017.
- Niedenthal, Rainer K.; Sen-gupta, Mark; Andreas, Wilmen; Hegemann, Johannes H. (1993), «Cpf1 protein induced bending of yeast centromere DNA element I», Nucleic Acids Research 21 (20): 4726–4726, PMID 8233820, doi:10.1093/nar/21.20.4726.
- Tanaka, Y.; Nureki, O.; Kurumizaka, H.; Fukai, S.; Kawaguchi, S.; Ikuta, M.; Iwahara, J.; Okazaki, T. et al. (2001), «… of the CENP-B protein–DNA complex: the DNA-binding domains of CENP-B induce kinks in the CENP-B …», The EMBO Journal 20: 6612–6618, doi:10.1093/emboj/20.23.6612, archivado desde el original el 8 de diciembre de 2008 .
- Brown, M.T. (1993), «MIF2 is required for mitotic spindle integrity during anaphase spindle elongation in Saccharomyces …», The Journal of Cell Biology 123 (2): 387–403.
- Meluh, P.B.; Koshland, D. (1997), «Budding yeast centromere composition and assembly as revealed by in vivo cross-linking», Genes & Development 11 (24): 3401–3412, doi:10.1101/gad.11.24.3401.
- Westermann, S.; Cheeseman, I.M.; Anderson, S.; Yates Iii, J.R.; Drubin, D.G.; Barnes, G. (2003), The Journal of Cell Biology 163 (2): 215 http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2173538
|url=sin título (ayuda). - Meluh, P.B.; Koshland, D. (1995), «Evidence that the MIF2 gene of Saccharomyces cerevisiae encodes a centromere protein with homology …», Molecular Biology of the Cell 6 (7): 793–807.
- McCarroll, R.M.; Fangman, W.L. (1988), «Time of replication of yeast centromeres and telomeres», Cell(Cambridge) 54 (4): 505–513, archivado desde el original el 1 de mayo de 2008, consultado el 28 de noviembre de 2008.
- Clarke, L.; Baum, M.; Marschall, L.G.; Ngan, V.K.; Steiner, N.C. (1993), «Structure and Function of Schizosaccharomyces pombe Centromeres», Cold Spring Harbor Symposia on Quantitative Biology 58: 687–687.
- Allshire, R.C. (1997), «Centromeres, checkpoints and chromatid cohesion», Current Opinion in Genetics & Development 7 (2): 264–273, doi:10.1016/S0959-437X(97)80137-2, archivado desde el original el 9 de diciembre de 2008.
- Steiner, N.C.; Hahnenberger, K.M.; Clarke, L. (1993), «Centromeres of the fission yeast Schizosaccharomyces pombe are highly variable genetic loci», Molecular and Cellular Biology 13 (8): 4578–4587.
- Ngan, V.K.; Clarke, L. (1997), «The centromere enhancer mediates centromere activation in Schizosaccharomyces pombe», Molecular and Cellular Biology 17 (6): 3305–3314.
- Karpen, G.H.; Allshire, R.C. (1997), «The case for epigenetic effects on centromere identity and function», Trends in Genetics 13 (12): 489–496, doi:10.1016/S0168-9525(97)01298-5.
- Volpe, T.; Schramke, V.; Hamilton, G.L.; White, S.A.; Teng, G.; Martienssen, R.A.; Allshire, R.C. (2003), «RNA interference is required for normal centromere function in fission yeast», Chromosome Research 11 (2): 137–146, doi:10.1023/A:1022815931524, archivado desde el original el 20 de abril de 2004.
- Nagaki, K.; Cheng, Z.; Ouyang, S.; Talbert, P.B.; Kim, M.; Jones, K.M.; Henikoff, S.; Buell, C.R. et al. (2004), «Sequencing of a rice centromere uncovers active genes», Nature Genetics 36: 138–145, doi:10.1038/ng1289 .
- Csink, A.K.; Henikoff, S. (1998), «Something from nothing: the evolution and utility of satellite repeats», Trends in Genetics 14 (5): 200–204, doi:10.1016/S0168-9525(98)01444-9.
- Bayes, Joshua J.; Malik, Harmit S. (Abril de 2008). «The Evolution of Centromeric DNA Sequences». Encyclopedia of Life Sciences (ELS). John Wiley & Sons, Ltd: Chichester. Archivado el 9 de diciembre de 2008 en Wayback Machine. DOI: 10.1002/9780470015902.a0020827
- Henikoff, Steven; Ahmad, Kami; Malik, Harmit S. (2001), «The Centromere Paradox: Stable Inheritance with Rapidly Evolving DNA», Science 293 (5532): 1098–1102, PMID 11498581, doi:10.1126/science.1062939.
- Lo, A.W.I.; Craig, J.M.; Saffery, R.; Kalitsis, P.; Irvine, D.V.; Earle, E.; Magliano, D.J.; Choo, K.H.A. (2001), «A 330 kb CENP-A binding domain and altered replication timing at a human neocentromere», The EMBO Journal 20: 2087–2096, doi:10.1093/emboj/20.8.2087.
- Choo, K.H.A. (2001), «Domain Organization at the Centromere and Neocentromere», Developmental Cell 1 (2): 165–177, doi:10.1016/S1534-5807(01)00028-4.
- Malik, H.S.; Henikoff, S. (2001), «Adaptive Evolution of Cid, a Centromere-Specific Histone in Drosophila», Genetics 157 (3): 1293–1298.
- Ahmad, K.; Henikoff, S. (2001), «Centromeres Are Specialized Replication Domains in Heterochromatin», The Journal of Cell Biology 153 (1): 101–110.
- Wong, L.H.; Brettingham-moore, K.H.; Chan, L.; Quach, J.M.; Anderson, M.A.; Northrop, E.L.; Hannan, R.; Saffery, R.; Shaw, M.L.; Williams, E.; Others (2007), «Centromere RNA is a key component for the assembly of nucleoproteins at the nucleolus and centromere», Genome Research 17 (8): 1146, doi:10.1101/gr.6022807.
- Schueler, M.G.; Sullivan, B.A. (2006), «Structural and Functional Dynamics of Human Centromeric Chromatin», Annual Reviews of Genomics and Human Genetics 77: 301-313, archivado desde el original el 13 de junio de 2010.
- Rudd, M.K.; Wray, G.A.; Willard, H.F. (2006), «The evolutionary dynamics of α-satellite», Genome Res 16: 88–96, doi:10.1101/gr.3810906.
- Fishman L., Saunders A. (2008). «Centromere-Associated Female Meiotic Drive Entails Male Fitness Costs in Monkeyflowers». Science 322 (5907). 1559 - 1562.
Bibliografía
- Alberts, Bruce, et al. Molecular Biology of the Cell, 4th ed. New York: Garland Publishing, 2000.
- Atherly, Alan G., Jack R. Girton, and John F. McDonald. The Science of Genetics. Philadelphia, PA: Saunders College Publishing, 1998.
- Cooper, D. N. Human Gene Mutation. Bios Scientific Publishers Ltd., 1997.