Embriogénesis en Drosophila
La embriogénesis en Drosophila es el conjunto de procesos biológicos que controlan la transformación de una única célula, el cigoto, en un individuo maduro de Drosophila melanogaster, animal modelo conocido popularmente como «mosca de la fruta». Puesto que se conocen mejor los detalles moleculares de este fenómeno en D. melanogaster que en ningún otro animal,[1] se ha conseguido una comprensión científica no desdeñable del proceso en conjunto, proceso que abarca las disciplinas de la genética molecular y del desarrollo y la biología molecular y celular.

Visión global
.jpg.webp)
Como en humanos, cada nuevo individuo de mosca de la fruta es producto de la fecundación por parte de un espermatozoide procedente de un macho al óvulo maduro de una hembra. Ambos gametos, haploides, entremezclan su material genético dando lugar a un cigoto diploide que contiene la información genética característica del nuevo ser. Tras la fecundación, el cigoto inicia una sucesión de divisiones casi sincrónicas en las regiones centrales del huevo que dan lugar a la aparición de un sincitio; después, tras nuevas divisiones, se establece una monocapa de células en la periferia del agregado que albergan al vitelo central, pero aún careciendo de membranas plasmáticas que definan las células individualmente; esta estructura, típica de insectos, se denomina blastodermo sincitial.[2] Antes de que surja dicha separación, se habla de que las células poseen un carácter totipotente, esto es, que cada futura célula posee la potencialidad de generar cualquier tipo celular; después de que se produzca la transición a un estado no sincitial, en cambio, sus núcleos se han determinado, lo cual quiere decir que cada una se diferenciará en un determinado tipo de tejido de la mosca adulta. La naturaleza de dicha capa celular, el blastodermo, ya determinado, produce que el desarrollo temprano sea esencialmente bidimensional; no obstante, el carácter tridimensional de la larva requiere un incremento en complejidad volumétrico, que sucede posteriormente, en la gastrulación.[2]
La simetría bilateral de Drosophila supone la aparición de dos ejes, anteroposterior y dorsoventral, ortogonales entre ellos, que han de ser definidos molecularmente. A lo largo del eje anteroposterior la larva posee una segmentación regular: anteriormente aparece la cabeza, luego tres segmentos torácicos y finalmente ocho segmentos abdominales, cada uno de los cuales tiene su propia identidad tanto por aspecto externo como interno. En los extremos de este eje aparecen dos estructuras especializadas, el acron y el telson. En cuanto al eje dorsoventral, este define cuatro regiones en el cuerpo larval.[2]
Establecimiento de los ejes
El establecimiento de los ejes en Drosophila está mediado por genes maternos, esto es, ARN mensajeros y proteínas preformados y procedentes de la madre pero también por genes zigóticos, es decir, aquellos producidos por expresión del genoma del embrión en desarrollo. Unos y otros intervienen tanto en la delimitación del eje dorsoventral como en el anteroposterior.
La expresión diferencial de genes cuyo productos son elementos reguladores del desarrollo repercuten en la morfología celular, morfología que queda modificada dando lugar, en conjunto, a uno o más ejes de simetría. Por ejemplo, se requiere de una red de microtúbulos intacta y polarizada dentro del ovocito a fin de establecer los ejes del huevo; de este modo, las señales entre las células foliculares y las del oocito (como los factores semejantes al TGF-alfa) provocan la reorganización de los microtúbulos situando su extremo menos en la zona anterior del oocito, lo que polariza la estructura y conlleva la aparición de un eje anterior-dorsal.[3]
Eje dorsoventral
El eje dorsoventral se establece en el embrión de Drosophila mediante la proteína Dorsal, que, al inicio de la embriogénesis, se encuentra dispersa por todo el embrión. No obstante, tras la migración de los núcleos a la periferia y la constitución del blastodermo, la proteína Dorsal penetra en los núcleos celulares de las regiones ventral y laterales, si bien permanece en el citoplasma en la región dorsal. Este mecanismo de transporte de Dorsal está regulado por una proteína señal denominada Spätzle, que, a su vez, se distribuye de forma heterogénea por el embrión: concretamente, posee un gradiente ventral a dorsal entre la matriz extracelular presenten entre la membrana plasmática del huevo sin fertilizar y la cubierta de este. Spätzle es un ligando de receptores Toll, y, puesto que se activa dependiendo de la abundancia de su ligando, está en plena actividad en la zona ventral del embrión, y decrece progresivamente en las regiones más laterales. Toll, cuando inicia su cascada de señalización, ocasiona la degradación de un inhibidor citoplasmático denominado Cactus (que afecta a Dorsal), lo que provoca la activación de este último. De este modo, se instaura el gradiente de Dorsal, especialmente abundante en la zona ventral y más diluido en las regiones laterales.[2]
Dorsal afecta a la actividad de otras proteínas dependiendo del umbral de sensibilidad de éstas: para algunas, es activo como estimulante a bajas concentraciones; para otras, requiere de altas dosis. De este modo, puede definirse una retahíla de umbrales en el eje dorsoventral que permitan una fina regulación a través de este. De este modo, tras la celularización del embrión, que sucede unas dos horas tras la fertilización, se instauran uno sumbrales que iniciarán la diferenciación de tres tejidos distintos: mesodermo, endodermo y ectodermo, que, como en todo animal triblástico, definirán la histología y anatomía del individuo adulto.[1]
El establecimiento del citado gradiente de Dorsal repercute en la regulación de tres genes diana distintos, activados por altas, intermedias y bajas concentraciones de la proteína Dorsal: estas son twist, rhomboid y sog. Esta regulación se produce: mediante la afinidad diferencial de enhancers en la secuencia de DNA aledaña a cada gen; debido a la presencia represores; y debido a la intervención de activadores.[1]
Twist
Los niveles más altos de Dorsal, propios de la zona ventral del embrión, estimulan la expresión de twist en las 18 células más ventrales que forman el mesodermo. Inactivo en las regiones laterales y ectodermo neurogénico pese a las concentraciones intermedias de Dorsal, twist no es estimulado debido a que en su región reguladora 5' posee dos lugares de unión de baja afinidad para Dorsal; de este modo, ambos deben enlazar a Dorsal, para lo cual su concentración debe ser muy alta, lo cual no sucede en las regiones laterales.[1] Posee un papel crucial en el desarrollo del mesodermo, y, por ello, del miotomo, uno de cuyos derivados, el músculo, ha sido muy estudiado.[4]
Rhomboid
Rhomboid es activado, en cambio, por niveles intermedios de la proteína Dorsal, concretamente en el ectodermo neurogénico ventral. Su región reguladora en el 5' contiene un enhancer que contiene un cluster de lugares de uión a Dorsal, la mayoría de los cuales son semejantes a los descritos pra twist, esto es, de baja afinidad. No obstante, uno de ellos es de alta afinidad, lo cual permite la unión eficaz aun a concentraciones intermedias de Dorsal, las presentes en las regiones laterales. De este modo, el enhancer de rhomboid' se vería activado tanto por los altos niveles de la proteína Dorsal en el mesodermo como mediante las intermedias de las zonas laterales; no obstante, esto no sucede debido a que en el mesodermo existe un represor transcripcional denominado Snail, que no existe en el ectodermo neurogénico.[1][5]
Sog
Sog, el tercer elemento sensible a Dorsal, esta vez a baja concentración, posee, para ello, cuatro lugares de unión a Dorsal con una afinidad óptima; concretamente, estos se sitúan en un enhancer interno al gen, cerca del primer intrón. De este modo, se expresa más ampliamente que los dos genes antes citados, debido a que, como en el caso de rhomboid, la presencia del represor Snail descarta su expresión en los lugares donde la concentración de Dorsal es máxima; de este modo, se establece un equilibrio dinámico entre la presencia de Snail y la alta afinidad de Dorsal por la región reguladora de sog.[1]
Eje anteroposterior
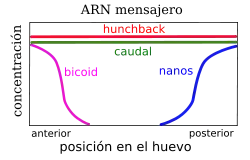
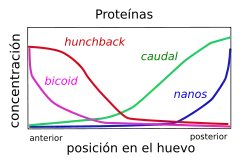
Durante los primeros estadios del desarrollo, son los genes maternos los que definen un eje anteroposterior, antes de la fertilización del huevo; dichas diferencias se acentúan delimitando una futura cabeza y un futuro elemento distal. Por conveniencia, se agrupan estos genes maternos en tres grupos: primero, aquellos que afectan a la región anterior; segundo, los equivalentes para la región posterior; y, finalmente, aquellos que se ven representados en ambas regiones terminales. Por ejemplo, bicoid pertenece al primer grupo, nanos al segundo y torso al tercero.[2]
Bicoid
Bicoid es un gen materno acumulado específicamente en la región cefálica, esto es, anterior del embrión, de tal modo que genera un gradiente en la larva, hasta no estar representado en el extremo posterior. Su ARN mensajero ya muestra dicha distribución diferencial en el huevo antes de la fecundación. Una vez se produce ésta, el RNA mensajero se traduce a proteína, la cual forma un gradiente con la máxima concentración en la zona más anterior del embrión, en el lugar de síntesis; además, su relativamente corta vida media, de treinta minutos, contribuye a la permanencia de dicho gradiente, puesto que, de ser más estable, por simple difusión el mencionado gradiente desaparecería. Dicha proteína posee actividad de factor de transcripción.[2][6]
Nanos
Nanos es otro gen materno que muestra una pauta de distribución recíproca a la de bicoid: en este caso, se acumula especialmente en el extremo posterior, y su concentración va disminuyendo conforme se avanza al futuro extremo cefálico. Dicha distribución peculiar se encuentra regulada por un grupo de genes posteriores, como oskar, que localiza el ARN mensajero de nanos en su posición cuando el huevo está sin fecundar. Más adelante, la traducción de nanos genera, como en el caso de bicoid, una proteína que también responde al gradiente definido por su ARNm. No obstante, existe una diferencia con respecto a bicoid, y es que nanos no es un morfógeno per se, puesto que actúa por inhibición de la traducción del ARNm de otro gen, este sí con actividad de morfógeno, denominado hunchback.[2][7]
Hunchback
Hunchback es un gen materno que, en el huevo sin fertilizar, se distribuye como ARNm a lo largo de todo el huevo. No obstante "nanos" actúa como represor que impide la traducción de dicho mRNA allá donde el propio nanos es más abundante, esto es, en el extremo posterior, lo cual repercute en una menor concentración de hunchback en este lugar.[2]
Por otra parte, el producte cigótico hunchback está regulado por el mRNA materno de bicoid, de forma que cuanta mayor sea la cantidad de bicoid mayor será la promoción de su transcripción.[8]
Así obtenemos un gradiente genético muy marcado donde la expresión de "hunchback" está muy promovida en la parte anterior del embrión y muy reprimida en la parte posterior.
Caudal
Caudal es otro elemento materno que define el eje anteroposterior en el embrión de Drosophila. Su ARNm de origen materno está distribuido a lo largo de todo el huevo sin fertilizar, pero, a lo largo de la embriogénesis, se establece un gradiente debido a que bicoid inhibe la síntesis de su proteína allá donde este, bicoid, es más abundante, esto es, en el extremo anterior; de este modo, caudal es menos abundante en dicha zona, la que en el futuro corresponderá a la cabeza.[2][9]
Sistema terminal
La configuración de los dos extremos del embrión, el acron, en la cabeza, y el telson, en la zona caudal, se deben a componentes maternos. Un gen, torso, que posiblemente se trata de un receptor tirosín kinasa de la membrana plasmática, se considera responsable de la recepción de una señal, un ligando, que lo activa especialmente en ambos extremos del huevo, de forma conjunta. Por tanto, el receptor, torso, se encuentra distribuido homogéneamente por la membrana plasmática, lo cual traslada el papel de señalizador topográfico al mencionado ligando, que, se postula, se trata de trunk, el cual poseería una distribución irregular en el vitelo en torno al huevo.[10]
Segmentación
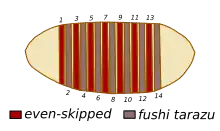
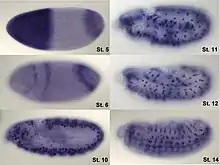
Dentro de la clase de los insectos, una característica muy representativa es la presencia de una serie de segmentos corporales con una entidad clara y una diferenciación variable dependiendo de su posición. Dicha metamería, regular respecto del eje anteroposterior, se presenta también en estadios anteriores al adulto, y debe establecerse durante el desarrollo embrionario.
No obstante, los elementos repetidos visibles en las larvas maduras no son estrictamente las primeras unidades de segmentación del individuo. Por el contrario, la segmentación empieza con el establecimiento de cuatro regiones anchas por acción de los genes gap.
Posteriormente, tras la gastrulación, aparecen en la superficie de la larva una serie de surcos que definen las áreas corporales que se denominan parasegmentos.[10] La identidad de cada parasegmento se define mediante la acción de genes de la regla de los pares[11] cada uno de los cuales se expresa en series de siete elementos transversales al eje anteroposterior, de forma alternada a otro u otros genes que lo hacen con un desfase de un parasegmento. Es decir: existen genes expresados en los parasegmentos pares (el 2, 4, 6, 8, 10, 12 y 14) y otros que lo hacen en los impares (esto eso, el 1, 3, 5, 7, 9 10, 11 y 13).[2] De este modo, la pauta de desarrollo de la larva se refina enormemente, pues se define un patrón consistente en el número correcto de segmentos pero, también, otro grupo de genes modula qué identidad posee cada uno.
Finalmente, actúan también los genes de la polaridad de segmento, que determinan la conversión de los parasegmentos en segmentos definitivos.
Genes homeóticos
El grupo de genes que controlan el establecimiento de la identidad de segmento se denominan complejos de genes homeóticos, presentes también en vertebrados.[10] Drosophila posee dos de estos complejos, que reciben el nombre de Antennapedia y bithorax.[12]
El complejo Antennapedia modula en buena parte la identidad de los segmentos cefálicos y del tórax anterior. Comprende cinco genes homeobox: labial (lab), proboscidea (pb), Deformed (Dfd), Sex combs reduced (Scr)y Antennapedia (Antp). Su disposición en el genoma mantiene una correlación con la pauta de expresión espacial y temporal: los elementos más cercanos al 3' son expresados de forma más temprana y en el extremo más cefálico. Deformed afecta a las estructuras de origen ectodérmico de los parasegmentos 0 y 1, Sex combs reduced lo hace en los parasegmentos 2 y 3, y Antennapedia, a los 4 y 5.[2]
Por otro lado, bithorax está relacionado con la identidad de los segmentos del tórax posterior y el abdomen. Agrupa a tres genes con homeobox: Ultrabithorax (Ubx), abdominal-A (abd_A) y Abdominal-B (Abd-B). Se expresan de forma combinatoria en los distintos parasegmentos para determinar su identidad: Ultrabithorax lo hace del 5 al 12; abdominal-A, del 7 al 13; y Abdominal-B, del parasegmento 10 hacia el extremo posterior. Sus actividades, no obstante, pueden combinarse para dotar de una especificidad biológica al parasegmento, siguiendo las reglas de los genes gap y de la regla de los pares.[10]

Debido a su elevada posición en la jerarquía de genes reguladores del patrón corporal, mutaciones en estos dos grupos de genes pueden provocar cambios drásticos en el fenotipo de las larvas; este hecho, denominado homeosis, se define como la conversión de una parte del cuerpo en otra. Tres ejemplos de cambios homeóticos, que afectan a la identidad del segmento pero al número total de éstos es:[11]
- Mutaciones de pérdida de función en bithorax: producen la conversión del tercer segmento torácico en el segundo segmento torácico, obteniéndose moscas con cuatro alas, en lugar de las dos normales (descontando los halterios).
- Mutaciones de ganancia de función Tab: provocan la transformación del segundo segmento torácico en el sexto abdominal, en el adulto.
- Mutaciones de ganancia de función Antennapedia: convierten las antenas en patas.
Apéndice
Lista de algunos genes implicados en el desarrollo de D. melanogaster:[11]
| Símbolo del gen | Nombre del gen | Función de la proteína | Implicación |
|---|---|---|---|
| hb-z | hunchback-zygotic | Factor de transcripción tipo dedo de cinc | Gen gap |
| Kr | Krüpel | Factor de transcripción tipo dedo de cinc | Gen gap |
| kni | knirps | Factor de transcripción tipo dedo de cinc | Gen gap |
| eve | even-skipped | Factor de transcripción; con homeodominio | Regla de los pares |
| ftz | futshi tarazu | Factor de transcripción; con homeodominio | Regla de los pares |
| opa | odd-paired | Factor de transcripción tipo dedo de cinc | Regla de los pares |
| prd | paired | Factor de transcripción | Regla de los pares |
| en | engrailed | Factor de transcripción; con homeodominio | Polaridad de segmento |
| ci | cubitus interruptus | Factor de transcripción tipo dedo de cinc | Polaridad de segmento |
| wg | wingless | Proteína de señalización WG | Polaridad de segmento |
| hh | hedgehog | Proteína de señalización HH | Polaridad de segmento |
| fu | fused | Serín-treonín kinasa citoplasmática | Polaridad de segmento |
| ptc | patched | Proteína transmembrana | Polaridad de segmento |
| arm | armadillo | Proteína de adhesión célula-célula | Polaridad de segmento |
| lab | labial | Factor de transcripción; con homeodominio | Identidad de segmento |
| Dfd | Deformed | Factor de transcripción; con homeodominio | Identidad de segmento |
| Antp | Antennapedia | Factor de transcripción; con homeodominio | Identidad de segmento |
| Ubx | Ultrabithorax | Factor de transcripción; con homeodominio | Identidad de segmento |
| dl | dorsal | Factor de transcripción | Eje dorsoventral |
| cact | cactus | Unión a la proteína Dorsal | Eje dorsoventral |
| spz | spaetzle | Molécula señal | Eje dorsoventral |
| Tl | Toll | Receptor del ligando de Spaetzle | Regulación de la expresión de Dorsal |
| snk | snake | Serín proteasa | Activación de spaetzle |
| dpp | decantaplegic | Molécula señal | Gen cardinal para el ectodermo dorsal |
| sax | saxophone | Receptor seríntreonínquinasa | Patrón de ectodermo dorsal |
| tkv | thick veins | Receptor seríntreonínquinasa | Patrón de ectodermo dorsal |
| pnt | punt | Receptor seríntreonínquinasa | Patrón de ectodermo dorsal |
| sog | short gastrulation | Señal, ligando SOG | Patrones generales de ectodermo |
| spi | spitz | Señal, ligando SPI | Patrón de ectodermo ventral |
Referencias
- Watson, J. D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. y Losick, R. (2004). Molecular Biology of the Gene (Fifth edition edición). San Francisco: Benjamin Cummings. ISBN 0-321-22368-3.
- Wolpert, Lewis; Beddington, R.; Jessell, T.; Lawrence, P.; Meyerowitz, E. y Smith, W. (2002). Principles of Development (Second edition edición). Oxford: Oxford University Press. ISBN 0-19-879291-3.
- Van Eeden, F.; Stjohnston, D. (1999), «The polarisation of the anterior-posterior and dorsal-ventral axes during Drosophila oogenesis», Current Opinion in Genetics & Development 9 (4): 396-404, doi:10.1016/S0959-437X(99)80060-4.
- Mary K. Baylies y Michael Bate (7 de junio de 1996). «twist: A Myogenic Switch in Drosophila». Science 272 (5267): 1481-1484. DOI: 10.1126/science.272.5267.1481.
- Brentrup, D.; H. Lerch, H. Jackle y M. Noll (2000). «Regulation of Drosophila wing vein patterning: net encodes a bHLH protein repressing rhomboid and is repressed by rhomboid-dependent Egfr signalling». Development 127 (21): 4729-4741.
- Berleth, T.; M. Burri, G. Thoma, D. Bopp, S. Richstein, G. Frigerio, M. Noll y C. Nüsslein-Volhard (1998). «The role of localization of bicoid RNA in organizing the anterior pattern of the Drosophila embryo». EMBO J. 7 (6): 1749-1756. PMCID: PMC457163.
- Wharton, Robin P. y Gary Struhl (1991). «RNA regulatory elements mediate control of Drosophila body pattern by the posterior morphogen nanos». Cell, Vol. 67 67: 955-967.
- Driever, Wolfgang & Christiane Nüsslein-Volhard (1989). «The bicoid protein is a positive regulator of hunchback transcription in the early Drosophila embryo». Nature 337: 138-143. doi:10.1038/337138a0.
- Mlodzik, M., Fjose, A., Gehring, W. J. (1985). «Isolation of caudal, a Drosophila homeo box-containing gene with maternal expression, whose transcripts form a concentration gradient at the pre-blastoderm stage». EMBO J. 4 (11): 2961-2969.
- Lawrence, Peter A. (1992). The Making of a Fly. Blackwell Science Ltd. ISBN 0-632-03048-8.
- Griffiths, J. F. A. et al. (2002). Genética. McGraw-Hill Interamericana. ISBN 84-486-0368-0.
- Michael Regulski, Katherine Harding, Richard Kostriken, Francois Karch, Michael Levine y William McGinnis (1985). «Homeo box genes of the Antennapedia and bithorax complexes of Drosophila». Cell 43 (1-80): 71-80.
Enlaces externos
- Patrones de expresión de algunos genes implicados en la embriogénesis de Drosophila, mediante hibridación in situ (en inglés).
- Animación de la embriogénesis de Drosophila