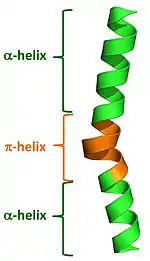
Hélice pi
Une hélice π est un type de structure secondaire des protéines. Cette structure secondaire, supposée rare, est en réalité observée dans 15 % des structures de protéines sous forme d'hélices π courtes. Elles sont formées par l'insertion d'un seul résidu d'acide aminé dans une hélice α et correspondrait à une adaptation évolutive. L'insertion d'un acide aminé au sein d'une hélice est énergétiquement déstabilisante ; les hélices π sont conservées lorsqu'elles apportent un avantage fonctionnel à la protéine, ce qui explique également leur position généralement à proximité du site actif des protéines.
Historique
Les hélices sont les principales structures secondaires trouvées dans les protéines. Elles sont décrites par la nomenclature Xy proposée par Donohue[1] où X correspond au nombre d'acides aminés par tour et y au nombre d'atomes dans la boucle formée par la liaison hydrogène. En plus des hélices α (3.613) et gamma (5.117) proposées par Pauling, Corey et Branson[2], d'autres hélices ont été proposées par une étude théorique des hélices 2.27, 310, 4.317 et 4.416[1]. De toutes ces hélices, seules les hélices α, les hélices 310 et les hélices π ont été observées dans les structures des protéines.
Caractéristiques
Structure
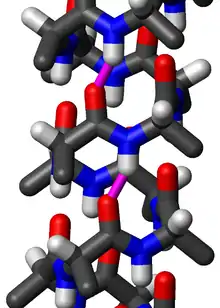
Les acides aminés sont organisés en une structure hélicoïdale droite dans une hélice π classique. Chaque acide aminé forme un angle de 87° dans l'hélice et entraîne une translation de 1,15 Å (0,115 nm) le long de l'axe hélicoïdal. Le pas de l'hélice π est formé de 4,4 résidus d'acides aminés[3]. Le groupe NH de la chaîne principale d'un acide aminé forme une liaison hydrogène avec le groupe C=O de la chaîne principale du cinquième acide aminé le précédant ; cette répétition i + 5 → i de liaisons hydrogène définit une hélice π, il faut un minimum de deux liaisons de ce type pour définir une hélice π.
La majorité des hélices π sont formées de sept acides aminés dont les angles dièdres (φ, ψ) ne prennent pas de valeurs moyennes comme dans le cas des hélices α et des feuillets β. Pour cette raison, les bases de données des angles dièdres peuvent donner des résultats trompeurs dans l'identification des hélices π, il est possible d'améliorer ces résultats si les angles dièdres des acides aminés des extrémités de l'hélice π sont exclus. Lorsque ces deux acides aminés sont exclus, la moyenne de la somme des angles dièdres φ et ψ est de -125°. La moyenne de la somme des angles dièdres du premier acide aminé est d'environ -95° et de -105°.
Composition en acides aminés
L'analyse de nombreuses hélices π montre une présence privilégiée des acides aminés aromatiques et à longues chaînes aliphatiques aux extrémités de ce type de structure : phénylalanine, tryptophane, tyrosine, isoleucine, leucine et méthionine. Les autres positions sont généralement occupées par des acides aminés polaires : asparagine, acide glutamique, thréonine et sérine. La position centrale des hélices π est occupée par un résidu d'asparagine[3].
Les acides aminés placés avant ou après l'hélice π ne sont pas spécifiques, hormis pour la position +1 après l'hélice π en direction de l'extrémité C-terminale, occupée par un résidu de proline. Cet acide aminé semble impliqué dans une liaison hydrogène avec l'acide aminé en position π3, et contribuerait à induire la formation de l'hélice π.
Stabilité
Les premières hypothèses présentent l'hélice π comme une structure secondaire instable pour des raisons de répulsions stériques[4]. Les interactions entre les chaînes latérales de types Van der Waals, les cycles aromatiques et des interactions électrostatiques sont des facteurs augmentant la stabilité de cette structure secondaire. De plus, l'alignement des acides aminés formant l'hélice π montré sur la figure entraîne un raccourcissement de la liaison hydrogène de la chaîne principale des acides aminés stabilisant davantage la structure.
Conformation gauche
Théoriquement, il serait possible d'obtenir une hélice π à pas de rotation gauche en inversant le signe des angles dièdres (φ, ψ) devenant alors (55°, 70°). Cette pseudo « image miroir » d'hélice π a le même nombre de résidus par tour (4,4) et le même pas (1,5 Å ou 150 pm) que l'hélice π classique. Cette structure n'est pas une véritable image miroir de l'hélice π classique car les acides aminés biologiques conservent leur chiralité initiale. Il est pratiquement impossible d'obtenir une hélice π à pas de rotation gauche, seule la glycine peut en effet adopter les angles dièdres requis tels que la valeur de 55° pour l'angle φ.
Les hélices π dans les protéines
Les premiers programmes d'identification de structures secondaires, comme DSSP, ont montré une présence des hélices π dans moins de 1 % des protéines criblées. Cette mauvaise caractérisation vient de la difficulté à les identifier. Les hélices π naturelles sont généralement courtes, formées de 7 à 10 résidus, presque toujours entourées par des hélices α, elles étaient attribuées comme partie intégrante d'hélices α tordues. Les nouveaux programmes d'identification utilisent les liaisons hydrogène et des restrictions pour les angles dièdres φ et ψ pour caractériser les hélices π, ils ont permis d'identifier la présence de ce type d'hélices dans 15 % des protéines[5].
Les hélices π naturelles peuvent être facilement identifiées dans une structure protéique comme un « gonflement » dans une hélice α longue. Ce renflement dans les hélices α a parfois été nommé renflement α, renflement π, boucle ou coude π, mais l'étude des liaisons hydrogène a permis d'identifier une répétition i + 5 → i caractéristique de l'hélice π. La formation d'une hélice π est liée à l'insertion d'un acide aminé supplémentaire au sein d'une hélice α existante. Il est possible de passer d'hélices α à des hélices π et réciproquement par l'insertion ou la suppression d'un seul acide aminé. L'ajout d'un acide aminé provoque l'apparition d'une hélice π, structure secondaire stable, qui entraîne cependant une déstabilisation de la structure globale de la protéine de l'ordre de 3 à 6 kcal/mol. Cette déstabilisation, la découverte d'un taux de 15 % de présence d'hélices π dans les protéines, principalement au niveau des sites actifs, suggèrent que la conversion des hélices α en hélices π est un mécanisme important de modification et de diversification de la fonctionnalité des protéines[6].
Ces différentes évolutions peuvent être observées dans la superfamille des ferritines qui inclut la ferritine, la bactérioferritine (en), la rubrérythrine (en), les ribonucléotide réductases de classe I ou les méthane monooxygénases (en) solubles. Cette dernière molécule contient plus de 13 hélices π[7]. La chaîne polypeptidique simple contenant le plus d'hélices π est un homologue bactérien d'un transporteur de neurotransmetteur Na+/Cl− dépendant qui en contient 8[8]. Il a également été observé un déplacement d'une hélice π au sein d'un site actif qui s'est déplacée de six acides aminés vers l'extrémité C-terminale dans la toluène 4-monooxygénase hydroxylase, ce mouvement péristaltique a pour conséquence d'augmenter le volume du site actif mais aussi de réguler l'activité de la protéine.
Notes et références
- (en) J Donohue, « Hydrogen Bonded Helical Configurations of the Polypeptide Chain », Proceedings of the National Academy of Sciences of the United States of America, vol. 39, no 6, , p. 470-478 (PMID 16589292, PMCID PMC1063809)
- (en) L Pauling, RB Corey et HR Branson, « The Structure of Proteins: Two Hydrogen-Bonded Helical Configurations of the Polypeptide Chain », Proceedings of the National Academy of Sciences of the United States of America, vol. 37, no 4, , p. 205-211 (PMID 14816373, PMCID 1063337, DOI 10.1073/pnas.37.4.205)
- (en) MN Fodje et S Al-Karadaghi, « Occurrence, conformational features and amino acid propensities for the pi-helix », Protein Engineering Design & Selection, vol. 15, no 5, , p. 353–358 (PMID 12034854, DOI 10.1093/protein/15.5.353)
- (en) BW Low et HJ Grenville-Wells, « Generalized Mathematical Relationships for Polypeptide Chain Helices. The Coordinates of the II Hélix », Proceedings of the National Academy of Sciences of the United States of America, vol. 39, no 8, , p. 785-801 (PMID 16589334, PMCID PMC1063858)
- (en) RB Cooley, TW Rhoads, DJ Arp et PA Karplus, « A diiron protein autogenerates a valine-phenylalanine cross-link. », Science, vol. 332, no 6032, , p. 629 (PMID 21596985, PMCID PMC3736988, DOI 10.1126/science.1205687)
- (en) LJ Keefe, J Sondek, D Shortle et EE Lattman, « The alpha aneurism : a structural motif revealed in an insertion mutant of staphylococcal nuclease », Proceedings of the National Academy of Sciences of the United States of America, vol. 90, no 8, , p. 3275–3279 (PMID 8475069, PMCID 46282, DOI 10.1073/pnas.90.8.3275)
- (en) AC Rosenzweig, H Brandstetter, DA Whittington, P Nordlund, SJ Lippard et CA Frederick, « Crystal structures of the methane monooxygenase hydroxylase from Methylococcus capsulatus (Bath): implications for substrate gating and component interactions. », Proteins, vol. 29, no 2, , p. 141-152 (PMID 9329079, DOI 10.2210/pdb1mty/pdb)
- (en) A Yamashita, SK Singh, T Kawate, Y Jin et E Gouaux, « Crystal structure of a bacterial homologue of Na+/Cl--dependent neurotransmitter transporters. », Nature, vol. 437, no 7056, , p. 215-223 (PMID 16041361, DOI 10.1038/nature03978)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Pi helix » (voir la liste des auteurs).
Voir aussi
Bibliographie
Articles connexes
- Portail de la biochimie
- Portail de la biologie cellulaire et moléculaire