Prédiction de la structure des protéines
La prédiction de la structure des protéines est l'inférence de la structure tridimensionnelle des protéines à partir de leur séquences d'acides aminés, c'est-à-dire la prédiction de leur pliage et de leur structures secondaire et tertiaire à partir de leur structure primaire.
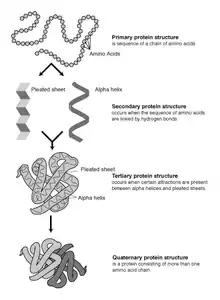
La prédiction de la structure est fondamentalement différente du problème inverse de la conception des protéines. Elle est l'un des objectifs les plus importants poursuivis par la bioinformatique et la chimie théorique. Elle est très importante en médecine (par exemple, dans la conception de médicaments) et en biotechnologie (par exemple, dans la conception de nouvelles enzymes).
Tous les deux ans, la performance des méthodes utilisées est évaluée dans l'expérience CASP (en) (en anglais : Critical Assessment of protein Structure Prediction, Évaluation critique des techniques de prédiction des protéines). Une évaluation continue des serveurs Web de prédiction de la structure des protéines est réalisée par le projet communautaire CAMEO3D (en).
Structure des protéines et terminologie
Les protéines sont des chaînes d'acides aminés réunis par des liaisons peptidiques. De nombreuses conformations de cette chaîne sont possibles du fait de la rotation de la chaîne autour de chaque atome de carbone (Cα). Ces changements de conformation sont responsables de différences dans la structure tridimensionnelle des protéines. Chaque acide aminé de la chaîne est polaire, c'est-à-dire qu'il a séparé des régions chargées positives et négatives avec un groupe C=O libre, qui peut agir comme accepteur de liaison hydrogène et un groupe NH, qui peut agir comme donneur de liaison hydrogène. Ces groupes peuvent donc interagir dans la structure protéique. Les 20 acides aminés peuvent être classés selon la chimie de leur chaîne latérale, qui joue également un rôle structurel important. La glycine occupe une position particulière, car elle possède la plus petite chaîne latérale, un seul atome d'hydrogène, et donc peut augmenter la flexibilité locale dans la structure protéique. La cystéine, pour sa part, peut réagir avec un autre résidu cystéine et ainsi former une liaison croisée stabilisant la structure entière.
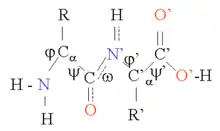
La structure protéique peut être considérée comme une séquence d'éléments structurels secondaires, tels que des hélices α et des feuilles β, qui constituent ensemble la configuration tridimensionnelle globale de la chaîne protéique. Dans ces structures secondaires, des motifs réguliers de liaisons H sont formés entre des acides aminés voisins, et les acides aminés ont des angles Φ et ω similaires.
La formation de ces structures neutralise les groupes polaires sur chaque acide aminé. Les structures secondaires sont étroitement emballées dans le cœur de protéine dans un environnement hydrophobe. Chaque groupe latéral d'acides aminés a un volume limité à occuper et un nombre limité d'interactions possibles avec d'autres chaînes latérales proches, une situation qui doit être prise en compte dans la modélisation moléculaire et les alignements[1].
Hélice α
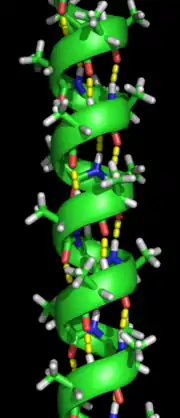
L'hélice α est le type de structure secondaire le plus abondant dans les protéines.
Elle est constituée de 3,6 acides aminés par tour, avec une liaison H formée entre chaque quatrième résidu ; la longueur moyenne est de 10 acides aminés (3 tours) ou 10 Å, mais peut varier de 5 à 40 (1,5 à 11 tours). L'alignement des liaisons H crée un moment dipolaire pour l'hélice, avec une charge positive partielle résultante à l'extrémité aminée de l'hélice. Comme cette région a des groupes NH2 libres, elle interagira avec des groupes chargés négativement tels que des phosphates.
La localisation la plus courante des hélices α est la surface des cœurs de protéines, où elles fournissent une interface avec le milieu aqueux. Le côté tourné vers l'intérieur de l'hélice a tendance à avoir des acides aminés hydrophobes et le côté extérieurs des acides aminés hydrophiles latéraux. Ainsi, chaque troisième de quatre acides aminés le long de la chaîne aura tendance à être hydrophobe, un motif qui peut être facilement détecté. Dans le motif à leucine, un motif récurrent de leucines sur les côtés opposés de deux hélices adjacentes est très prédictif du motif. Un tracé de roue hélicoïdale peut être utilisé pour montrer ce motif répété[pas clair]. D'autres hélices α enfouies dans le cœur de la protéine ou dans des membranes cellulaires ont une distribution plus élevée et plus régulière d'acides aminés hydrophobes et sont fortement prédictives de telles structures. Les hélices exposées à la surface ont une proportion plus faible d'acides aminés hydrophobes.
La teneur en acides aminés peut être prédictive d'une région α-hélicoïdale. Les régions riches en alanine (A), acide glutamique (E), leucine (L) et méthionine (M) et plus pauvres en proline (P), Glycine (G), tyrosine (Y) et serine (S) tendent à former des hélices α. La proline déstabilise ou casse les hélices α mais peut être présente dans des hélices plus longues, formant un virage.
Feuillets β

Les feuillets β sont formés par des liaisons H entre une moyenne de 5-10 acides aminés consécutifs dans une partie de la chaîne avec 5 à 10 autres acides aminés plus loin dans la chaîne. Les régions d'interaction peuvent être adjacentes, avec une courte boucle entre elles, ou éloignée, séparées par d'autres structures. Chaque chaîne peut s'associer avec des chaînes dans le même sens pour former un feuillet parallèle, ou dans le sens inverse pour former une feuillet anti-parallèle ; les feuillets mélangées comprennent des chaînes parallèles et des chaînes anti-parallèles. Le collage est différent dans les configurations parallèles et anti-parallèles.
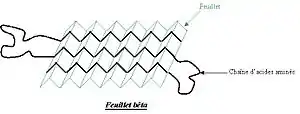
Chaque acide aminé des brins intérieurs du feuillet forme deux liaisons H avec des acides aminés voisins, alors que chaque acide aminé des brins extérieurs ne forme qu'une liaison avec un brin intérieur. En regardant à travers le feuillet perpendiculairement aux brins, les brins plus éloignés sont tournés légèrement dans le sens contraire des aiguilles d'une montre pour former une torsion à gauche. Les atomes de Carbone alternent au-dessus et au-dessous du feuillet dans une structure plissée, et les groupes latéraux R des acides aminés alternent au-dessus et au-dessous des plis. Les angles Φ et Ψ des acides aminés en feuillets varient considérablement dans le diagramme de Ramachandran. Il est donc plus difficile de prédire l'emplacement des feuilles β que des hélices α. La situation s'améliore quelque peu lorsqu'on prend en compte la variation des acides aminés dans les alignements de séquences multiples.
Coudes
Les coudes sont des régions d'une chaîne protéique qui sont (1) entre des hélices α et des feuillets β, (2) de longueurs et de configurations tridimensionnelles variables, et (3) à la surface de la structure. Les boucles en épingle à cheveux qui inversent complètement le sens de la chaîne polypeptidique pour joindre deux brins ß antiparallèles peuvent ne pas dépasser deux acides aminés de longueur.
Les coudes interagissent avec l'environnement aqueux environnant et d'autres protéines. Puisque les acides aminés dans les coudes ne sont pas contraints par l'espace et l'environnement comme ceux de la région centrale, et n'ont pas d'effet sur l'agencement des structures secondaires dans le cœur protéique, plus de substitutions, d'insertions et de délétions peuvent s'y produire. Ainsi, dans un alignement de séquences, la présence de ces caractéristiques peut être l'indication d'un coude. Les coudes ont également tendance à avoir des acides aminés chargés et polaires et sont fréquemment une composante des sites actifs.
Enroulements
Une région de structure secondaire qui n'est pas une hélice α, une feuille β ou une spire reconnaissable est communément appelée enroulement.
Classification des protéines
Les protéines peuvent être classées en fonction de la structure et de la similitude des séquences. Pour la classification structurale, les tailles et les agencements spatiaux des structures secondaires décrites dans le paragraphe ci-dessus sont comparés dans des structures tridimensionnelles connues.
La classification basée sur la similitude de séquence est la première à avoir été utilisée. Elle était initialement basée sur des alignements de séquences entières. Plus tard, les protéines ont été classées sur la base de l'apparition de motifs d'acides aminés conservés. Des bases de données qui classent les protéines selon un ou plusieurs de ces schémas sont disponibles.
En considérant les schémas de classification des protéines, il est important de garder plusieurs éléments à l'esprit :
- Premièrement, deux séquences de protéines entièrement différentes provenant d'origines évolutives différentes peuvent se plier dans une structure similaire. Inversement, la séquence d'un gène ancien pour une structure donnée peut avoir divergé considérablement dans différentes espèces, tout en conservant les mêmes caractéristiques structurelles de base. Reconnaître toute similarité de séquence restante dans de tels cas peut être très difficile.
- Deuxièmement, deux protéines qui partagent un degré significatif de similarité de séquence, soit l'une avec l'autre, soit avec une troisième séquence, partagent aussi une origine évolutive et devraient également partager certaines caractéristiques structurelles. Toutefois, la duplication des gènes et les réarrangements génétiques au cours de l'évolution peuvent donner lieu à de nouvelles copies de gènes, qui peuvent ensuite évoluer en protéines dotées de nouvelles fonctions et de nouvelles structures[1].
Termes les plus couramment utilisés
Les termes les plus couramment utilisés pour les relations évolutives et structurelles entre les protéines sont énumérés ci-dessous. De nombreux termes supplémentaires sont utilisés pour divers types de caractéristiques structurelles des protéines. Des descriptions de ces termes peuvent être trouvées sur les sites Web de la CATH (en) et de la Classification structurelle des protéines (SCOP) et un tutoriel Glaxo-Wellcome sur le site Web de Swiss Bioinformatics Expasy.
Architecture
Les orientations relatives des structures secondaires dans une structure tridimensionnelle, sans considérer si elles partagent ou non une structure de boucle similaire.
Pliage
Un type d'architecture qui a également une structure de boucle conservée[pas clair].
Blocs
Un modèle de séquence d'acides aminés conservé dans une famille de protéines. Le motif comprend une série de correspondances possibles à chaque position dans les séquences représentées, mais il n'y a pas de positions insérées ou supprimées dans le motif ou dans les séquences. En revanche, les profils de séquence sont un type de matrice de notation qui représente un ensemble de motifs semblables qui comprend des insertions et des suppressions.
Classe
Terme utilisé pour classer les domaines protéiques selon leur contenu structurel secondaire et leur organisation. Quatre classes ont été initialement reconnues par Levitt et Chothia (1976), et plusieurs autres ont été ajoutées dans la base de données SCOP. Trois classes sont données dans la base de données CATH (en) : principalement-α, principalement-β, et α-β, classe comprenant les structures alternantes α / β et α + β.
Cœur
La partie d'une molécule protéique repliée qui comprend l'intérieur hydrophobe des hélices α et des feuillets β. Cette structure compacte rassemble les groupes latéraux d'acides aminés dans une proximité suffisamment proche pour qu'ils puissent interagir. Lors de la comparaison des structures protéiques, comme dans la base de données SCOP, le cœur est la région commune à la plupart des structures qui partagent un pli commun ou appartiennent à la même superfamille. Dans la prédiction de structure, le cœur est parfois défini comme l'arrangement de structures secondaires susceptible d'être conservé pendant le changement évolutif.
Domaine (contexte de séquence)

Un segment d'une chaîne polypeptidique qui peut se replier dans une structure tridimensionnelle indépendamment de la présence d'autres segments de la chaîne. Les domaines distincts d'une protéine donnée peuvent interagir de manière extensive ou peuvent être joints seulement par une longueur de chaîne polypeptidique. Une protéine avec plusieurs domaines peut utiliser ces domaines pour des interactions fonctionnelles avec différentes molécules.
Famille (contexte de séquence)
Un groupe de protéines de même fonction biochimique qui sont identiques à plus de 50 % lorsqu'elles sont alignées. Cette définition est encore utilisée par la base de données Protein Information Resource (PIR). Une famille de protéines comprend des protéines ayant la même fonction dans différents organismes (séquences orthologues) mais peut également inclure des protéines du même organisme (séquences paralogues) dérivées de duplication de gènes et de réarrangements. Si un alignement de séquences multiples d'une famille de protéines révèle un niveau commun de similarité sur toute la longueur des protéines, PIR se réfère à la famille comme une famille homéomorphique. La région alignée est appelée domaine homéomorphe, et cette région peut comprendre plusieurs domaines d'homologie plus petits qui sont partagés avec d'autres familles. Les familles peuvent être subdivisées en sous-familles ou regroupées en superfamilles en fonction des niveaux respectifs de similitude des séquences. La base de données SCOP comporte 1296 familles et la base de données CATH (en) (version 1.7 bêta) 1846 familles.
Lorsqu'on examine plus en détail les séquences de protéines ayant la même fonction, on constate qu'elles partagent une grande similitude de séquence. Elles sont évidemment membres de la même famille selon les critères ci-dessus. D'autres ont cependant très peu de similitude de séquence avec d'autres membres de la famille. Dans de tels cas, la relation familiale entre deux membres de la famille distants A et C peut souvent être démontrée en trouvant un membre de famille supplémentaire B qui partage une similarité significative avec A et C. B fournit ainsi un lien de connexion entre A et C. Une autre approche est d'examiner les alignements éloignés pour les similitudes très conservées.
À un niveau d'identité de 50 %, les protéines sont susceptibles d'avoir la même structure tridimensionnelle, et les atomes identiques dans l'alignement de séquences seront également superposés dans environ 1 Å dans le modèle structurel. Ainsi, si la structure d'un membre d'une famille est connue, une prédiction fiable peut être faite pour un deuxième membre de la famille, et plus le niveau d'identité est élevé, plus la prédiction est fiable. La modélisation structurale des protéines peut être réalisée en examinant à quel point les substitutions d'acides aminés s'intègrent dans le cœur de la structure tridimensionnelle.
Pli
Similaire à un motif structurel, un pli comprend une plus grande combinaison d'unités structurelles secondaires dans la même configuration. Ainsi, les protéines partageant le même pli ont la même combinaison de structures secondaires reliées par des boucles similaires. Un exemple est le pli Rossmann, qui comprend plusieurs hélices α alternées et brins β parallèles. Dans les bases de données SCOP, CATH et FSSP, les structures protéiques connues ont été classées en niveaux hiérarchiques de complexité structurelle, avec le pli comme niveau de classification de base.
Domaine homologue (contexte de séquence)
C'est un modèle de séquence étendu, généralement trouvé par des méthodes d'alignement de séquences, qui indique une origine évolutive commune parmi les séquences alignées. Un domaine d'homologie est généralement plus long que des motifs. Le domaine peut inclure toute une séquence de protéine donnée ou seulement une partie de la séquence. Certains domaines sont complexes et composés de plusieurs domaines d'homologie plus petits qui se sont joints pour former un plus grand au cours de l'évolution. Un domaine qui couvre une séquence entière est appelé domaine homéomorphe dans la base PIR (Protein Information Resource).
Module
C'est une région de motifs d'acides aminés conservés comprenant un ou plusieurs motifs et considérée comme une unité fondamentale de structure ou de fonction. La présence d'un module a également été utilisée pour classer les protéines en familles.
Motif protéique (contexte de séquence)
C'est un modèle d'acides aminés conservé dans deux ou plusieurs protéines. Dans le catalogue PROSITE, un motif est un motif d'acides aminés qui se trouve dans un groupe de protéines qui ont une activité biochimique similaire, et qui est souvent près du site actif de la protéine. Des exemples de base de données de motifs de séquence sont le catalogue PROSITE et la base de données Stanford Motifs[2].
Motif (contexte structurel)
C'est une combinaison de plusieurs éléments structurels secondaires produits par pliage de sections adjacentes de la chaîne polypeptidique en une configuration tridimensionnelle spécifique. Un exemple est le motif hélice-boucle-hélice. Les motifs structuraux sont également appelés structures super-secondaires et plis.
Structure primaire
C'est la séquence des acides aminés constituant la protéine, sans aucune considération de la structure de celle-ci.
Profil (contexte de séquence)
C'est une matrice de notation qui représente l'alignement de séquences multiples d'une famille de protéines. Le profil est habituellement obtenu à partir d'une région bien conservée dans un alignement de séquences multiples.
Il se présente sous la forme d'une matrice, chaque colonne représentant une position dans l'alignement et chaque ligne l'un des acides aminés. Les valeurs de la matrice donnent la probabilité de chaque acide aminé à la position correspondante dans l'alignement. Le profil est déplacé le long de la séquence cible pour localiser les meilleures régions de notation par un algorithme de programmation dynamique. Les écarts sont autorisés pendant l'appariement et une pénalité d'écart est incluse dans ce cas comme score négatif quand aucun acide aminé n'est apparié. Un profil de séquence peut également être représenté par un modèle de Markov caché, appelé profil HMM (en anglais hidden Markov model).
Profil (contexte structurel)
Une matrice de notation qui représente quels acides aminés doivent bien s'adapter et lesquels ne devraient pas convenir à quelle position dans une structure protéique connue. Les colonnes du profil représentent des positions séquentielles dans la structure et ses rangées représentent les 20 acides aminés. Comme pour un profil de séquence, le profil structurel est déplacé le long d'une séquence cible pour trouver le score d'alignement le plus élevé possible par un algorithme de programmation dynamique. Les écarts peuvent être inclus et recevoir une pénalité. Le score obtenu fournit une indication pour savoir si la protéine pourrait adopter la structure considérée.
Site actif
Une combinaison localisée de groupes latéraux d'acides aminés au sein de la structure tertiaire (tridimensionnelle) ou quaternaire (sous-unité de protéine) qui peut interagir avec un substrat chimiquement spécifique et qui fournit à la protéine une activité biologique. Les protéines de séquences d'acides aminés très différentes peuvent se plier dans une structure qui produit le même site actif.
Structure tridimensionnelle
C'est la structure tridimensionnelle d'une molécule protéique comprenant plusieurs chaînes polypeptidiques indépendantes. Les interactions qui se produisent entre les groupes C, O et NH sur les acides aminés dans les chaînes polypeptidiques forment des hélices α, des feuillets ß, des spires, des boucles et d'autres formes qui facilitent le pliage dans une structure tridimensionnelle.
Super-famille
C'est un groupe de familles de protéines de même longueur ou de différentes longueurs qui sont liées par une similarité de séquence distante, mais détectable. Les membres d'une super-famille donnée ont donc une origine évolutive commune.
À l'origine, Margaret Oakley Dayhoff a défini le seuil pour le statut de super-famille comme étant la chance que les séquences ne soient pas liées de 10 6[pas clair], sur la base d'un score d'alignement (Dayhoff et al., 1978). Les protéines ayant peu d'identités dans un alignement de séquences, mais avec un nombre convaincant commun de caractéristiques structurelles et fonctionnelles, sont placées dans la même super-famille.
Au niveau de la structure tridimensionnelle, les protéines de la super-famille partagent des caractéristiques structurelles communes telles qu'un pli commun, mais peuvent également présenter des différences dans le nombre et la disposition des structures secondaires. La ressource PIR utilise le terme « superfamille homéomorphes » pour désigner des super-familles composées de séquences qui peuvent être alignées de bout en bout, représentant le partage d'un domaine d'homologie de séquences unique, une région de similarité qui s'étend tout au long de l'alignement. Ce domaine peut également comprendre des domaines d'homologie plus petits qui sont partagés avec d'autres familles et super-familles de protéines.
Bien qu'une séquence de protéine donnée puisse contenir des domaines trouvés dans plusieurs super-familles, indiquant ainsi une histoire évolutive complexe, les séquences seront assignées à une seule super-famille homéomorphe selon la présence de similarité dans un alignement de séquences multiples. L'alignement de la super-famille peut également comprendre des régions qui ne s'alignent ni à l'intérieur, ni aux extrémités de l'alignement. En revanche, les séquences d'une même famille s'alignent bien tout au long de l'alignement.
Structure supersecondaire
Terme ayant une signification similaire pour un motif structurel. La structure tertiaire est la structure tridimensionnelle ou globulaire formée par l'assemblage ou le pliage des structures secondaires d'une chaîne polypeptidique.
Structure secondaire
La prédiction de la structure secondaire est un ensemble de techniques de bio-informatique qui visent à prédire la structure locale de la protéine en se basant seulement sur la connaissance de leur séquence d'acides aminés. La prédiction consiste à désigner des régions de la séquence d'acides aminés comme hélices alpha, feuillets bêta (souvent notées comme des conformations « étendues ») ou coudes probables. Le succès de la prédiction est déterminé en le comparant aux résultats de l'algorithme DSSP (en) (ou similaire, par exemple STRIDE (en)) appliqué à la structure cristalline de la protéine. Des algorithmes spécialisés ont été développés pour détecter des motifs protéiques bien définis comme les hélices transmembranaires (en) et les superhélices[1].
Les meilleures méthodes modernes de prédiction de la structure secondaire dans les protéines atteignent une précision d'environ 80 %[3]. Cette précision élevée permet d'utiliser ces prédictions pour améliorer la reconnaissance des repliements et la prédiction de la structure des protéines ab initio, la classification des motifs structurels et la finesse des alignement de séquences. L'exactitude des méthodes actuelles de prédiction de la structure secondaire des protéines est évaluée par des tests de performance hebdomadaires comme LiveBench (en) et EVA (en).
Structure tertiaire
Le rôle pratique de la prédiction de la structure des protéines est maintenant plus important que jamais[4]. Des quantités massives de données de séquence de protéines sont produites par des efforts de séquençage de l'ADN à grande échelle comme le projet génome humain. En dépit des efforts communautaires de génomique structurale, la production de structures protéiques déterminées expérimentalement - typiquement par une cristallographie aux rayons X ou une spectroscopie RMN relativement coûteuse - est très en retard par rapport à la production de séquences de protéines.
La prédiction de la structure des protéines demeure une entreprise extrêmement difficile et non résolue. Les deux principaux problèmes sont le calcul de l'énergie libre des protéines et la recherche de son minimum global. Une méthode de prédiction de structure de protéine doit explorer l'ensemble des structures protéiques possibles, qui est gigantesque. Ces problèmes peuvent être partiellement contournés par des procédés de modélisation « comparative », par homologie ou par reconnaissance de pli, dans lesquels l'espace de recherche est élagué en supposant que la protéine en question adopte une structure proche de la structure (déterminée expérimentalement) d'une protéine homologue. D'un autre côté, les méthodes de prédiction de structure de protéine de novo (ou ab initio) doivent résoudre explicitement ces problèmes. Les progrès et les défis de la prédiction de la structure des protéines ont été examinés par Zhang en 2008[5].
Méthodes basées sur l'énergie de fragments
Les méthodes de modélisation des protéines ab initio (ou de novo) cherchent à construire des modèles protéiques tridimensionnels « à partir de zéro », c'est-à-dire basés sur des principes physiques plutôt que (directement) sur des structures préalablement résolues. Il existe de nombreuses procédures possibles qui tentent d'imiter le repliement des protéines ou d'appliquer une méthode stochastique pour rechercher des solutions possibles (c'est-à-dire l'optimisation globale d'une fonction énergétique appropriée). Ces procédures ont tendance à nécessiter de vastes ressources informatiques et n'ont donc été réalisées que pour des protéines minuscules. Pour prédire la structure de la protéine de novo pour de plus grandes protéines, il faudra de meilleurs algorithmes et de plus grandes ressources informatiques, comme celles fournies par de puissants supercalculateurs (comme Blue Gene ou MDGRAPE-3) ou par informatique distribuée (comme Folding@home, Human Proteome Folding Project et Rosetta@home). Bien que ces barrières de calcul soient élevéess, les avantages potentiels de la génomique structurale (par des méthodes prédites ou expérimentales) font de la prédiction de la structure ab initio un domaine de recherche actif[5].
En 2009, une protéine de 50 résidus[Quoi ?] pouvait être simulée atome par atome sur un supercalculateur pendant 1 milliseconde[6]. En 2012, un échantillonnage comparable à l'état stable pourrait être effectué sur un ordinateur bureau standard avec une carte graphique récente et des algorithmes plus sophistiqués[7]. La simulation d'une durée beaucoup plus importante peut être obtenue en utilisant une modélisation moléculaire à grains grossiers[8],[9].
Covariation évolutive pour prédire les contacts 3D
À mesure que le séquençage devenait plus courant dans les années 1990, plusieurs équipes ont utilisé des alignements de séquences de protéines pour prédire des mutations corrélées et on espérait que ces résidus coevolués pourraient être utilisés pour prédire la structure tertiaire (en utilisant l'analogie aux contraintes de distance des procédures expérimentales comme la RMN). L'hypothèse est que lorsque les mutations de résidus simples sont légèrement délétères, des mutations compensatoires peuvent se produire pour rétablir les interactions résidus-résidus. Ces premiers travaux utilisaient ce que l'on appelle des méthodes locales pour calculer des mutations corrélées à partir de séquences protéiques, mais ils ont souffert de fausses corrélations indirectes résultant du traitement de chaque paire de résidus comme indépendant de toutes les autres paires[10],[11],[12].
En 2011, une approche statistique différente, cette fois globale, a démontré que les résidus prévus coévolués étaient suffisants pour prédire le pli 3D d'une protéine, à condition de fournir assez de séquences (plus de 1 000 séquences homologues sont nécessaires)[13]. Cette méthode (EVfold) n'utilise pas de modélisation d'homologie, de segmentation ou de fragments de structure 3D et peut être exécutée sur un ordinateur personnel standard même pour des protéines possédant des centaines de résidus. La précision des contacts prévus par cette approche et les approches connexes a maintenant été démontrée sur de nombreuses structures et cartes de contact connues[14],[15],[16], y compris la prédiction de protéines transmembranaires non résolues expérimentalement.
Modélisation comparée des protéines
La modélisation comparée des protéines utilise des structures préalablement résolues comme points de départ ou modèles. Ceci est efficace car bien que le nombre de protéines réelles soit important, il n'existe qu'un ensemble limité de motifs structurels tertiaires auxquels appartiennent la plupart d'entre elles. Il a été suggéré qu'il n'y aurait qu'environ 2 000 plis protéiques distincts dans la nature, contre plusieurs millions de protéines différentes.
Ces méthodes peuvent également être divisées en deux groupes[5] :
Elle est basée sur l'hypothèse raisonnable que deux protéines homologues partageront des structures très semblables. Puisque le pli d'une protéine est plus conservé évolutivement que sa séquence d'acides aminés, une séquence cible peut être modélisée avec une précision raisonnable sur un modèle très éloigné, à condition que la relation entre la cible et le modèle puisse être discernée par alignement de séquences. Il a été suggéré que le principal goulet d'étranglement dans la modélisation comparée découle des difficultés d'alignement plutôt que des erreurs dans la prédiction de la structure, étant donné un alignement bien connu[17]. Sans surprise, la modélisation par homologie est plus précise lorsque la cible et le modèle ont des séquences similaires.
La modélisation par enfilage[18] compare la séquence d'acides aminés d'une structure inconnue à une base de données de structures résolues. Dans chaque cas, une fonction de notation est utilisée pour évaluer la compatibilité de la séquence avec la structure, sélectionnant ainsi des modèles tridimensionnels possibles. Ce type de procédé est également connu sous le nom de reconnaissance de pliage 3D-1D en raison de son analyse de compatibilité entre des structures tridimensionnelles et des séquences de protéines linéaires. Ce procédé a également donné lieu à des procédés effectuant une recherche de repliement inverse en évaluant la compatibilité d'une structure donnée avec une grande base de données de séquences, prédisant ainsi quelles séquences ont le potentiel de produire un pli donné.
Structure quaternaire
L'amarrage macromoléculaire est la modélisation informatique de la structure quaternaire de complexes formés par plusieurs macromolécules biologiques en interaction. Les modélisations les plus courantes sont celles des complexes protéine-protéine et protéine-acide nucléique.
Notes et références
- (en) Mount DM, Bioinformatics : Sequence and Genome Analysis, vol. 2, Cold Spring Harbor Laboratory Press, (ISBN 0-87969-712-1).
- (en) JY. Huang et DL. Brutlag, « The emotif database », Nucleic Acids Res, vol. 29, no 1, , p. 202-04 (PMID 11125091, DOI 10.1093 / nar / 29.1.202).
- (en) W. Pirovano et J. Heringa, Data Mining Techniques for the Life Sciences, vol. 609, coll. « Methods in Molecular Biology », , 327-48 p. (PMID 20221928, DOI 10.1007 / 978-1-60327-241-4_19), « Protein secondary structure prediction ».
- (en) Márcio Dorn, Mariel Barbachan e Silva, Luciana S. Buriol et Luis C. Lamb, « Three-dimensional protein structure prediction: Methods and computational strategies », Computational Biology and Chemistry, vol. 53, , p. 251–276 (ISSN 1476-9271, PMID 25462334, DOI 10.1016/j.compbiolchem.2014.10.001, lire en ligne)
- (en) Zhang Y., « Progress and challenges in protein structure prediction », Current Opinion in Structural Biology, vol. 18, no 3, , p. 342–8 (PMID 18436442, PMCID 2680823, DOI 10.1016/j.sbi.2008.02.004).
- (en) Shaw DE, Dror RO, Salmon JK, Grossman JP, Mackenzie KM, Bank JA, Young C, Deneroff MM, Batson B, Bowers KJ, Chow E, Millisecond-scale molecular dynamics simulations on Anton, , 1 p. (ISBN 9781605587448, DOI 10.1145/1654059.1654126)
- (en) Pierce LC, Salomon-Ferrer R, de Oliveira CA, McCammon JA, Walker RC, « Routine Access to Millisecond Time Scale Events with Accelerated Molecular Dynamics », Journal of Chemical Theory and Computation, vol. 8, no 9, , p. 2997–3002 (PMID 22984356, PMCID 3438784, DOI 10.1021/ct300284c)
- (en) Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A, « Coarse-Grained Protein Models and Their Applications », Chemical Reviews, vol. 116, no 14, , p. 7898–936 (PMID 27333362, DOI 10.1021/acs.chemrev.6b00163)
- (en) Cheung NJ, Yu W, « De novo protein structure prediction using ultra-fast molecular dynamics simulation », PLOS ONE, vol. 13, no 11, , e0205819 (PMID 30458007, PMCID 6245515, DOI 10.1371/journal.pone.0205819, Bibcode 2018PLoSO..1305819C)
- (en) Göbel U, Sander C, Schneider R, Valencia A, « Correlated mutations and residue contacts in proteins », Proteins, vol. 18, no 4, , p. 309–17 (PMID 8208723, DOI 10.1002/prot.340180402, S2CID 14978727)
- (en) Taylor WR, Hatrick K, « Compensating changes in protein multiple sequence alignments », Protein Engineering, vol. 7, no 3, , p. 341–8 (PMID 8177883, DOI 10.1093/protein/7.3.341)
- (en) Neher E, « How frequent are correlated changes in families of protein sequences? », Proceedings of the National Academy of Sciences of the United States of America, vol. 91, no 1, , p. 98–102 (PMID 8278414, PMCID 42893, DOI 10.1073/pnas.91.1.98, Bibcode 1994PNAS...91...98N)
- (en) Marks DS, Colwell LJ, Sheridan R, Hopf TA, Pagnani A, Zecchina R, Sander C, « Protein 3D structure computed from evolutionary sequence variation », PLOS ONE, vol. 6, no 12, , e28766 (PMID 22163331, PMCID 3233603, DOI 10.1371/journal.pone.0028766, Bibcode 2011PLoSO...628766M)
- (en) Burger L, van Nimwegen E, « Disentangling direct from indirect co-evolution of residues in protein alignments », PLOS Computational Biology, vol. 6, no 1, , e1000633 (PMID 20052271, PMCID 2793430, DOI 10.1371/journal.pcbi.1000633, Bibcode 2010PLSCB...6E0633B)
- (en) Morcos F, Pagnani A, Lunt B, Bertolino A, Marks DS, Sander C, Zecchina R, Onuchic JN, Hwa T, Weigt M, « Direct-coupling analysis of residue coevolution captures native contacts across many protein families », Proceedings of the National Academy of Sciences of the United States of America, vol. 108, no 49, , E1293-301 (PMID 22106262, PMCID 3241805, DOI 10.1073/pnas.1111471108
 , Bibcode 2011PNAS..108E1293M, arXiv 1110.5223)
, Bibcode 2011PNAS..108E1293M, arXiv 1110.5223) - (en) Nugent T, Jones DT, « Accurate de novo structure prediction of large transmembrane protein domains using fragment-assembly and correlated mutation analysis », Proceedings of the National Academy of Sciences of the United States of America, vol. 109, no 24, , E1540-7 (PMID 22645369, PMCID 3386101, DOI 10.1073/pnas.1120036109
 , Bibcode 2012PNAS..109E1540N)
, Bibcode 2012PNAS..109E1540N) - (en) Zhang Y, Skolnick J, « The protein structure prediction problem could be solved using the current PDB library », Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no 4, , p. 1029–34 (PMID 15653774, PMCID 545829, DOI 10.1073/pnas.0407152101
 , Bibcode 2005PNAS..102.1029Z)
, Bibcode 2005PNAS..102.1029Z) - (en) Bowie JU, Lüthy R, Eisenberg D, « A method to identify protein sequences that fold into a known three-dimensional structure », Science, vol. 253, no 5016, , p. 164–70 (PMID 1853201, DOI 10.1126/science.1853201, Bibcode 1991Sci...253..164B)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Protein structure prediction » (voir la liste des auteurs).
Bibliographie
- (en) K. Majorek, L. Kozlowski, M. Jakalski, J. M. Bujnicki et J. Bujnicki (dir.), Prediction of Protein Structures, Functions, and Interactions, Chichester, John Wiley & Sons, Ltd., , 39–62 p. (ISBN 978-0-470-51767-3, DOI 10.1002/9780470741894.ch2, lire en ligne), « Chapter 2: First Steps of Protein Structure Prediction »
- D. Baker et A. Sali, « Protein structure prediction and structural genomics », Science, vol. 294, no 5540, , p. 93–96 (PMID 11588250, DOI 10.1126/science.1065659)
- LA. Kelley et MJ. Sternberg, « Protein structure prediction on the Web: a case study using the Phyre server », Nat Protoc, vol. 4, no 3, , p. 363–71 (PMID 19247286, DOI 10.1038/nprot.2009.2)
- A. Kryshtafovych et K. Fidelis, « Protein structure prediction and model quality assessment », Drug Discov Today, vol. 14, nos 7–8, , p. 386–93 (PMID 19100336, DOI 10.1016/j.drudis.2008.11.010)
- X. Qu, R. Swanson, R. Day et J. Tsai, « A guide to template based structure prediction », Curr Protein Pept Sci, vol. 10, no 3, , p. 270–85 (PMID 19519455)
- PR. Daga, RY. Patel et RJ. Doerksen, « Template-based protein modeling: recent methodological advances », Curr Top Med Chem, vol. 10, no 1, , p. 84–94 (PMID 19929829, DOI 10.2174/156802610790232314)
- A. Fiser, « Template-based protein structure modeling », Methods Mol Biol, vol. 673, , p. 73–94 (ISBN 978-1-60761-841-6, PMID 20835794, PMCID 4108304, DOI 10.1007/978-1-60761-842-3_6)
- D. Cozzetto et A. Tramontano, « Advances and pitfalls in protein structure prediction », Curr Protein Pept Sci, vol. 9, no 6, , p. 567–77 (PMID 19075747, DOI 10.2174/138920308786733958)
- Nayeem A, Sitkoff D, Krystek S Jr, Sitkoff et Krystek Jr, « A comparative study of available software for high-accuracy homology modeling: From sequence alignments to structural models », Protein Sci, vol. 15, no 4, , p. 808–24 (PMID 16600967, PMCID 2242473, DOI 10.1110/ps.051892906)
Voir aussi
- Complexe biomoléculaire, tout complexe biologique de protéines, d'ARN, d'ADN (contenant parfois des lipides et des glucides)
- Docking (moléculaire), petite molécule d'amarrage aux protéines
- Enfilage, l'une des techniques de modélisation de la structure des protéines, par analogie avec une structure existante
- AlphaFold
Liens externes
- CASP experiments home page
- ExPASy Proteomics tools — list of prediction tools and servers
- Portail de la biologie cellulaire et moléculaire