Ruta de señalización Notch
La Ruta de Señalización Notch es un sistema de señalización celular altamente conservado en los animales, con el cometido principal de controlar los destinos celulares mediante la amplificación y consolidación de diferencias entre células adyacentes.[1][2]
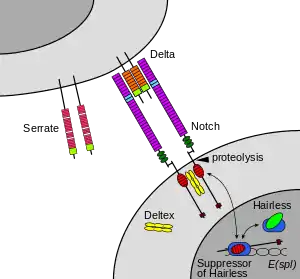
La Ruta se basa en la proteína funcional NOTCH, proteína transmembranal que sirve como receptor de señales extracelulares. Esta proteína es un hetero-oligomero compuesto por dos porciones extracelulares, una larga y una corta, asociadas por una interacción no covalente dependiente de calcio, y una porción intracelular corta.[3] Los mamíferos cuentan con cuatro receptores NOTCH diferentes.[4]
La ruta de señalización Notch participa en procesos proliferativos en la neurogénesis y mantenimiento de nichos de célula madre. Su actividad es inhibida por Numb.
Historia
En 1914, Jhon S. Dexter detectó el gen Notch en las alas de la mosca de la fruta Drosophila melanogaster. Los alelos del gen se describieron en 1917 por Thomas Hunt Morgan,[5] pero la secuenciación de estos se logró durante los 80's por los equipos de Spyros Artavanis-Tsakonas[6] & Michael W. Young.[7]
Posteriormente dos paralogos redundantes del gen Notch en C. elegans se identificaron basado en los fenotipos lin-12[8] y glp-1.[9][10] La clonación y secuenciación parcial de lin-12 también se reportó para Drosophila por Iva Greenwald en 1985.[11]
Proteínas Notch
| Dominio de Repetición Lin12/Notch (LNR) | ||
|---|---|---|
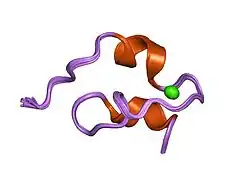 Prototipo de estructura del módulo LNR de la proteína Notch1 Humana [12] | ||
| Identificadores | ||
| Símbolo | Notch | |
| Pfam | PF00066 | |
| InterPro | IPR000800 | |
| SMART | SM00004 | |
| Familia OPM | 462 | |
| Proteína OPM | 5kzo | |
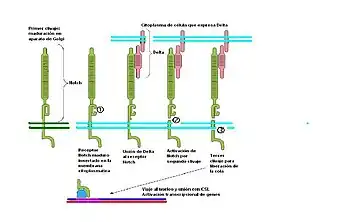
Las proteína Notch pertenecen a la familia de proteínas transmembranales tipo 1. Compuesta por dos dominios principales, el dominio Notch extracelular (en inglés NECD) que interactúa directamente con la familia de ligandos DSL (Delta, Serrate, Lag2) y el dominio Notch intracelular (NICD) conectados por un dominio transmembranal de un solo paso (TM). La proteína se sintetiza y ensambla en el aparato de golgi mediante proteasas furinas.[13]
El NECD está compuesto por repeticiones múltiples de similes de los Factores de Crecimiento Epidermal (EGF). La cantidad de repeticiones varía según el organismo: 36 para Drosophila,[14] 28-36 en humanos, en C.elegans encontramos 13 repeticiones para Lin-12 y 10 para GLP-1.[15] Estas repeticiones son fuertemente modificadas por o-glicosilación y se ha demostrado que estas modificaciones son importantes para su funcionamiento.[16]
Estas repeticiones de EGF son seguidas por repeticiones ricas en cisteína de la Repetición Lin-12/Notch (LNR) y un dominio de heterodimerización (HD).Tanto la LNR como la HD componen la región regulatoria negativa adyacente a la membrana celular que previene la señalización en ausencia del ligando.
El NICD actúa como un Factor de Transcripción que es liberado una vez que el ligando active su clivaje. Contiene una secuencia de localización nuclear (NLS) que permite su translocación al núcleo. También posee repeticiones de Akirina (ANK) e interacciones de dominio RAM (módulo asociado RBP-Kappa) que ayudan en la activación génica.
Mecanismo
El receptor es activado por el contacto directo célula-célula, donde la proteína Notch entra en contacto con los ligandos de la otra célula. La unión del ligando al NECD induce el clivaje proteolítico que libera el NICD y se transloca al núcleo donde modifica la expresión génica.[2][17]
El modelo de clivaje de Notch fue propuesto en 1993 basado en trabajos en lin-12.[18][19] Este se confirmó en 1998 con análisis in-vivo en Drosophila por Gary Struhl[20] y de manera in-vitro por Raphael Kopan.[21]
Después del clivaje, una vez en el núcleo, las repeticiones de akirina (ANK) y las interacciones por dominio RAM presentes en el NCID interactúa con la Familia de Factores de Transcripción CSL (CBF1/RBP-JKappa[22] Supresor de Hairless & Lag-1) y también Mastermind,[23] formando un complejo de transcripción.[24][25] Se ha visto que en humanos el domino PEST ayuda en la degradación del NICD.[26]
Función
Neurogénesis
La señalización por medio de NOTCH está involucrada en el desarrollo de la mayoría de los tejidos, pero ha sido mayormente estudiada en la producción de células nerviosas en Drosophila, en un proceso conocido como inhibición lateral, en el que células con expresión NOTCH y Delta de un clúster empiezan a inhibirse unas a otras cuando Delta activa a NOTCH en algún lugar, y se empiezan a enviar señales inhibitorias a todas las células que estén expresando NOTCH. Se da así una competencia que consecuentemente disminuye la habilidad de las células para responder con la señal inhibitoria de Delta. Queda por último una única célula que envía una fuerte señal inhibitoria a sus vecinas, y no recibe señal a cambio. Esa será entonces una célula neural rodeada de células epiteliales.[27][28]
Los procesos de inhibición lateral y diversificación celular que son iniciados por la expresión de genes proneurales y mediados por NOTCH han demostrado ser cruciales en la formación de patrones finos en una gran variedad de tejidos, tejidos en los que se requieren mezclas equilibradas de diferentes tipos celulares, y NOTCH permite en ese proceso que células individuales expresen un grupo de genes que indiquen a las células adyacentes que expresen un grupo diferente.[29]
Otros procesos
Mediante el mecanismo de inhibición lateral la ruta notch influye fuertemente en la morfogénesis de diferentes tejidos como:
- La estabilización de los destinos endoteliales en la angiogénesis.[30] Y la expansión de la células hemogénicas endoteliales junto a señales SHH y Scl.[31]
- Regulación de eventos de comunicación celular entre el endocardio y miocardio durante la formación de los primordios de la válvula cardiaca y la diferenciación ventriculares.[32] La homeostasis de la válvula cardiaca y la implicación en desórdenes cardiovasculares.
- Especificación de linajes celulares en páncreas endocrino y exocrino.[33]
- Influencia del destino entre linajes absorbente o secretores del intestino.[34]
- Expansión del compartimiento de células madres hematopoyéticas durante el desarrollo óseo. También la participación en el linaje osteoblástico.[35]
- Compromiso del linaje de células T a partir de un precursor linfoide único.[36]
- Regulación del destino de glándulas mamarias a través de diferentes etapas del desarrollo.[37]
- Regulación de la línea germinal de C.elegans y los destinos mitóticos/meióticos.[38]
En la enfermedad
La señal de notch se ha visto des-regulada en diferentes cánceres, incluyendo Leucemia linfoblástica aguda de células T.[39] Tampien la deficiencia de la ruta Notch se relaciona con otras enfermedades como CADASIL ( Arteriopatía cerebral autosómica dominante con infartos subcorticales y leucoencefalopatía), esclerosis múltiple, Tetralogía de Fallot, Síndrome de Alagille, entre otras enfermedades.
Referencias
- "Molecular Cell Biology". 4th edition. Lodish H, Berk A, Zipursky SL, et al. New York: W. H. Freeman; 2000.
- Gilbert, Scott F., 1949-. Developmental biology (Eleventh edition edición). ISBN 978-1-60535-470-5. OCLC 945169933. Consultado el 19 de mayo de 2020.
- Brou, Christel; Logeat, Frédérique; Gupta, Neetu; Bessia, Christine; LeBail, Odile; Doedens, John R; Cumano, Ana; Roux, Pascal et al. (2000-02). «A Novel Proteolytic Cleavage Involved in Notch Signaling». Molecular Cell (en inglés) 5 (2): 207-216. doi:10.1016/S1097-2765(00)80417-7. Consultado el 19 de mayo de 2020.
- Kumar, Rajesh; Juillerat-Jeanneret, Lucienne; Golshayan, Dela (8 de septiembre de 2016). «Notch Antagonists: Potential Modulators of Cancer and Inflammatory Diseases». Journal of Medicinal Chemistry (en inglés) 59 (17): 7719-7737. ISSN 0022-2623. doi:10.1021/acs.jmedchem.5b01516. Consultado el 19 de mayo de 2020.
- Morgan, T. H. (1917-09). «The Theory of the Gene». The American Naturalist (en inglés) 51 (609): 513-544. ISSN 0003-0147. doi:10.1086/279629. Consultado el 19 de mayo de 2020.
- Wharton, Kristi A.; Johansen, Kristen M.; Xu, Tian; Artavanis-Tsakonas, Spyros (1985-12). «Nucleotide sequence from the neurogenic locus Notch implies a gene product that shares homology with proteins containing EGF-like repeats». Cell (en inglés) 43 (3): 567-581. doi:10.1016/0092-8674(85)90229-6. Consultado el 19 de mayo de 2020.
- Kidd, S; Kelley, M R; Young, M W (1986-09). «Sequence of the notch locus of Drosophila melanogaster: relationship of the encoded protein to mammalian clotting and growth factors.». Molecular and Cellular Biology (en inglés) 6 (9): 3094-3108. ISSN 0270-7306. doi:10.1128/MCB.6.9.3094. Consultado el 19 de mayo de 2020.
- Greenwald, Iva S.; Sternberg, Paul W.; Robert Horvitz, H. (1983-09). «The lin-12 locus specifies cell fates in caenorhabditis elegans». Cell (en inglés) 34 (2): 435-444. doi:10.1016/0092-8674(83)90377-X. Consultado el 19 de mayo de 2020.
- Austin, Judithe; Kimble, Judith (1987-11). «glp-1 Is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans». Cell (en inglés) 51 (4): 589-599. doi:10.1016/0092-8674(87)90128-0. Consultado el 19 de mayo de 2020.
- Austin, Judith; Kimble, Judith (1989-08). «Transcript analysis of glp-1 and lin-12, homologous genes required for cell interactions during development of C. elegans». Cell (en inglés) 58 (3): 565-571. doi:10.1016/0092-8674(89)90437-6. Consultado el 20 de mayo de 2020.
- Greenwald, Iva (1985-12). «lin-12, a nematode homeotic gene, is homologous to a set of mammalian proteins that includes epidermal growth factor». Cell (en inglés) 43 (3): 583-590. doi:10.1016/0092-8674(85)90230-2. Consultado el 19 de mayo de 2020.
- «Nuclear magnetic resonance structure of a prototype Lin12-Notch repeat module from human Notch1». Biochemistry 42 (23): 7061-7. June 2003. PMID 12795601. doi:10.1021/bi034156y. Parámetro desconocido
|vauthors=ignorado (ayuda) - Munro, Sean; Freeman, Matthew (2000-07). «The Notch signalling regulator Fringe acts in the Golgi apparatus and requires the glycosyltransferase signature motif DxD». Current Biology (en inglés) 10 (14): 813-820. doi:10.1016/S0960-9822(00)00578-9. Consultado el 21 de mayo de 2020.
- Wharton, Kristi A.; Johansen, Kristen M.; Xu, Tian; Artavanis-Tsakonas, Spyros (1985-12). «Nucleotide sequence from the neurogenic locus Notch implies a gene product that shares homology with proteins containing EGF-like repeats». Cell (en inglés) 43 (3): 567-581. doi:10.1016/0092-8674(85)90229-6. Consultado el 20 de mayo de 2020.
- Greenwald, Iva (1985-12). «lin-12, a nematode homeotic gene, is homologous to a set of mammalian proteins that includes epidermal growth factor». Cell (en inglés) 43 (3): 583-590. doi:10.1016/0092-8674(85)90230-2. Consultado el 20 de mayo de 2020.
- Shao, L.; Luo, Y.; Moloney, D. J.; Haltiwanger, R. S. (1 de noviembre de 2002). «O-Glycosylation of EGF repeats: identification and initial characterization of a UDP-glucose: protein O-glucosyltransferase». Glycobiology (en inglés) 12 (11): 763-770. ISSN 0959-6658. doi:10.1093/glycob/cwf085. Consultado el 20 de mayo de 2020.
- Oswald, Franz; Täuber, Birgitt; Dobner, Thomas; Bourteele, Soizic; Kostezka, Ulrike; Adler, Guido; Liptay, Susanne; Schmid, Roland M. (15 de noviembre de 2001). «p300 Acts as a Transcriptional Coactivator for Mammalian Notch-1». Molecular and Cellular Biology (en inglés) 21 (22): 7761-7774. ISSN 1098-5549. doi:10.1128/MCB.21.22.7761-7774.2001. Consultado el 20 de mayo de 2020.
- Lieber, T; Kidd, S; Alcamo, E; Corbin, V; Young, M W (1 de octubre de 1993). «Antineurogenic phenotypes induced by truncated Notch proteins indicate a role in signal transduction and may point to a novel function for Notch in nuclei.». Genes & Development (en inglés) 7 (10): 1949-1965. ISSN 0890-9369. doi:10.1101/gad.7.10.1949. Consultado el 20 de mayo de 2020.
- Struhl, Gary; Fitzgerald, Kevin; Greenwald, Iva (1993-07). «Intrinsic activity of the lin-12 and Notch intracellular domains in vivo». Cell (en inglés) 74 (2): 331-345. doi:10.1016/0092-8674(93)90424-O. Consultado el 20 de mayo de 2020.
- Struhl, Gary; Adachi, Atsuko (1998-05). «Nuclear Access and Action of Notch In Vivo». Cell (en inglés) 93 (4): 649-660. doi:10.1016/S0092-8674(00)81193-9. Consultado el 20 de mayo de 2020.
- Schroeter, Eric H.; Kisslinger, Jeffrey A.; Kopan, Raphael (1998-05). «Notch-1 signalling requires ligand-induced proteolytic release of intracellular domain». Nature (en inglés) 393 (6683): 382-386. ISSN 0028-0836. doi:10.1038/30756. Consultado el 20 de mayo de 2020.
- Wilson, Jeffrey J.; Kovall, Rhett A. (2006-03). «Crystal Structure of the CSL-Notch-Mastermind Ternary Complex Bound to DNA». Cell (en inglés) 124 (5): 985-996. doi:10.1016/j.cell.2006.01.035. Consultado el 21 de mayo de 2020.
- Nam, Yunsun; Sliz, Piotr; Song, Luyan; Aster, Jon C.; Blacklow, Stephen C. (2006-03). «Structural Basis for Cooperativity in Recruitment of MAML Coactivators to Notch Transcription Complexes». Cell (en inglés) 124 (5): 973-983. doi:10.1016/j.cell.2005.12.037. Consultado el 21 de mayo de 2020.
- Deregowski, Valerie; Gazzerro, Elisabetta; Priest, Leah; Rydziel, Sheila; Canalis, Ernesto (22 de mayo de 2006). «Role of the Ram Domain and Ankyrin Repeats on Notch Signaling and Activity in Cells of Osteoblastic Lineage». Journal of Bone and Mineral Research (en inglés) 21 (8): 1317-1326. doi:10.1359/jbmr.060505. Consultado el 20 de mayo de 2020.
- Tamura, Kumiko; Taniguchi, Yoshihito; Minoguchi, Shigeru; Sakai, Takashi; Tun, Tin; Furukawa, Takahisa; Honjo, Tasuku (1995-12). «Physical interaction between a novel domain of the receptor Notch and the transcription factor RBP-Jκ/Su(H)». Current Biology (en inglés) 5 (12): 1416-1423. doi:10.1016/S0960-9822(95)00279-X. Consultado el 20 de mayo de 2020.
- Weng, A. P. (8 de octubre de 2004). «Activating Mutations of NOTCH1 in Human T Cell Acute Lymphoblastic Leukemia». Science (en inglés) 306 (5694): 269-271. ISSN 0036-8075. doi:10.1126/science.1102160. Consultado el 20 de mayo de 2020.
- Madame Curie Bioscience Database [Internet]. Austin (TX): Landes Bioscience; 2000-.
- Greenwald, Iva (2012-07). «Notch and the Awesome Power of Genetics». Genetics (en inglés) 191 (3): 655-669. ISSN 0016-6731. doi:10.1534/genetics.112.141812. Consultado el 21 de mayo de 2020.
- "Molecular Biology of the Cell". 4th edition. Alberts B, Johnson A, Lewis J, et al. New York: Garland Science; 2002.
- Liu, Zhao-Jun; Shirakawa, Takashi; Li, Yan; Soma, Akinobu; Oka, Masahiro; Dotto, G. Paolo; Fairman, Ronald M.; Velazquez, Omaida C. et al. (1 de enero de 2003). «Regulation of Notch1 and Dll4 by Vascular Endothelial Growth Factor in Arterial Endothelial Cells: Implications for Modulating Arteriogenesis and Angiogenesis». Molecular and Cellular Biology (en inglés) 23 (1): 14-25. ISSN 0270-7306. PMC 140667. PMID 12482957. doi:10.1128/MCB.23.1.14-25.2003. Consultado el 21 de mayo de 2020.
- Kim, P. G.; Albacker, C. E.; Lu, Y.-f.; Jang, I.-h.; Lim, Y.; Heffner, G. C.; Arora, N.; Bowman, T. V. et al. (8 de enero de 2013). «Signaling axis involving Hedgehog, Notch, and Scl promotes the embryonic endothelial-to-hematopoietic transition». Proceedings of the National Academy of Sciences (en inglés) 110 (2): E141-E150. ISSN 0027-8424. PMC 3545793. PMID 23236128. doi:10.1073/pnas.1214361110. Consultado el 21 de mayo de 2020.
- Grego-Bessa, Joaquín; Luna-Zurita, Luis; del Monte, Gonzalo; Bolós, Victoria; Melgar, Pedro; Arandilla, Alejandro; Garratt, Alistair N.; Zang, Heesuk et al. (2007-03). «Notch Signaling Is Essential for Ventricular Chamber Development». Developmental Cell (en inglés) 12 (3): 415-429. doi:10.1016/j.devcel.2006.12.011. Consultado el 21 de mayo de 2020.
- Murtaugh, L. C.; Stanger, B. Z.; Kwan, K. M.; Melton, D. A. (9 de diciembre de 2003). «Notch signaling controls multiple steps of pancreatic differentiation». Proceedings of the National Academy of Sciences (en inglés) 100 (25): 14920-14925. ISSN 0027-8424. doi:10.1073/pnas.2436557100. Consultado el 21 de mayo de 2020.
- Sander, Guy R.; Powell, Barry C. (2004-04). «Expression of Notch Receptors and Ligands in the Adult Gut». Journal of Histochemistry & Cytochemistry (en inglés) 52 (4): 509-516. ISSN 0022-1554. doi:10.1177/002215540405200409. Consultado el 21 de mayo de 2020.
- Nobta, Masuhiro; Tsukazaki, Tomoo; Shibata, Yasuaki; Xin, Chang; Moriishi, Takeshi; Sakano, Seiji; Shindo, Hiroyuki; Yamaguchi, Akira (22 de abril de 2005). «Critical Regulation of Bone Morphogenetic Protein-induced Osteoblastic Differentiation by Delta1/Jagged1-activated Notch1 Signaling». Journal of Biological Chemistry (en inglés) 280 (16): 15842-15848. ISSN 0021-9258. doi:10.1074/jbc.M412891200. Consultado el 21 de mayo de 2020.
- Laky, Karen; Fowlkes, B.J. (2008-04). «Notch signaling in CD4 and CD8 T cell development». Current Opinion in Immunology (en inglés) 20 (2): 197-202. doi:10.1016/j.coi.2008.03.004. Consultado el 21 de mayo de 2020.
- Dontu, Gabriela; Jackson, Kyle W; McNicholas, Erin; Kawamura, Mari J; Abdallah, Wissam M; Wicha, Max S (2004-12). «Role of Notch signaling in cell-fate determination of human mammary stem/progenitor cells». Breast Cancer Research (en inglés) 6 (6): R605. ISSN 1465-542X. doi:10.1186/bcr920. Consultado el 21 de mayo de 2020.
- Austin, Judithe; Kimble, Judith (1987-11). «glp-1 Is required in the germ line for regulation of the decision between mitosis and meiosis in C. elegans». Cell (en inglés) 51 (4): 589-599. doi:10.1016/0092-8674(87)90128-0. Consultado el 21 de mayo de 2020.
- Sharma, Vishva Mitra; Draheim, Kyle M.; Kelliher, Michelle A. (15 de abril de 2007). «The Notch1/c-Myc Pathway in T Cell Leukemia». Cell Cycle (en inglés) 6 (8): 927-930. ISSN 1538-4101. doi:10.4161/cc.6.8.4134. Consultado el 21 de mayo de 2020.