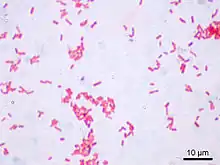
Escherichia coli
Escherichia coli, également appelée colibacille et abrégée en E. coli, est une bactérie intestinale (Gram négatif) des mammifères, en forme de bâtonnet, très commune chez l'être humain. E. coli est une bactérie anaérobie facultative que l'on trouve dans l'intestin des vertébrés. En effet, elle compose environ 80 % de notre flore intestinale aérobie. Découverte en 1885 par Theodor Escherich, dans des selles de chèvres, c'est un coliforme fécal généralement commensal. Cependant, certaines souches d’E. coli peuvent être pathogènes, entraînant alors des gastro-entérites, infections urinaires, méningites ou sepsis. Sous certaines conditions, E. coli serait capable de produire du méthane.

Grossissement × 15 000
| Règne | Bacteria |
|---|---|
| Embranchement | Proteobacteria |
| Classe | Gammaproteobacteria |
| Ordre | Enterobacterales |
| Famille | Enterobacteriaceae |
| Genre | Escherichia |
Description
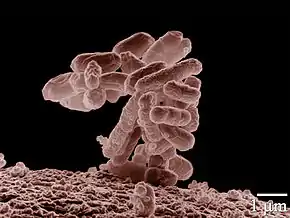
Escherichia coli est un bacille gram négatif radiorésistant de la famille des Enterobacteriaceae[1]. Sa taille varie en fonction des conditions de croissance (entre 0,5 à 3 µm), pesant de 0,5 à 5 picogrammes[2] les bactéries en croissance rapide étant plus allongées et donc plus grandes que les bactéries quiescentes. Le diamètre d'E. Coli est de 0,5 µm en moyenne[1].
C’est un hôte commun du microbiote intestinal (anciennement appelé microflore commensale intestinale) de l’humain et des animaux homéothermes[3]. Son établissement dans le tractus digestif s’effectue durant les premières heures ou journées qui suivent l’accouchement [4]. Escherichia coli constitue alors tout au long de la vie de l’hôte l’espèce bactérienne dominante de la flore aérobie facultative intestinale. E. coli est sans doute l’organisme vivant le plus étudié à ce jour : en effet, l'ancienneté de sa découverte et sa culture aisée (division cellulaire toutes les vingt minutes à 37 °C dans un milieu riche) en font un outil d'étude de choix. La profusion de publications scientifiques qui la mentionnent en témoigne, et elle joue le rôle de « cheval de labour » dans tous les laboratoires de biologie moléculaire.
La métagénétique a également permis d'apprendre qu'Escherichia coli fait partie de la flore bactérienne commensale de l’oreille des chiens et que sa présence aurait un rôle d’agent protecteur contre d’autres pathogènes[5].
On a récemment montré qu'en présence de fer libre, elle peut produire du méthane[6].
Historique
Theodor Escherich, en observant la fréquence des diarrhées néonatales, avait déjà posé la question de l’implication du colibacille dans les entérites. Après la Seconde Guerre mondiale, les connaissances ont convergé pour établir le concept de virulence de certaines souches de E. coli. Dans les années 1950, de nombreuses souches d’E. coli ont été incriminées en tant qu’agent étiologique de diarrhées infantiles[7].
- On sait maintenant que certaines souches « spécialisées » d’E. coli sont associées à des pathologies très diverses (y compris extra-intestinales), tant chez l’humain que chez l’animal ; diarrhées, gastro-entérites, infections urinaires, méningites, septicémies, syndrome hémolytique et urémique (SHU ou maladie des hamburgers), etc.
En prévention, une surveillance des SHU a lieu au Centre National de Référence des E. coli[8], situé dans l’unité de Biodiversité des Bactéries Pathogènes Émergentes à l’Institut Pasteur (France), qui est chargé d’étudier les souches pathogènes.
Depuis les années 1950, les bactériologistes ont essayé, grâce aux différences antigéniques de E. coli, de subdiviser l’espèce en sérotypes en immunisant des lapins avec des antigènes somatiques et flagellaires. Le sérogroupage reste la méthode la plus utilisée actuellement.
Le sérotype est la combinaison des deux antigènes, somatique O et flagellaire H, (exemples : O157:H7 et O111:H8), alors que le sérogroupe n'est déterminé que par l’antigène O (exemple : O157, O111). Cependant le sérotype n’est pas suffisant pour caractériser les E. coli pathogènes. Chaque sérotype n’est pas nécessairement corrélé à la pathogénicité.
Depuis 2010, un laboratoire de Californie (LS9[9]) a expliqué qu'il utilise cette bactérie pour produire des hydrocarbures, technique qui sera peut-être à l'origine de la synthèse du pétrole[réf. nécessaire]. Cette bactérie est encore depuis 2010 l'objet d'études en xénobiologie.
Cycle de vie

Antigènes et sérogroupage
L’antigène somatique O, définissant le sérogroupe, est contenu dans les lipopolysaccharides présents sur la paroi bactérienne des souches à Gram négatif. L’antigène flagellaire H est de nature protéique entrant dans la structure du flagelle (cilliature péritriche) permettant la mobilité de la bactérie. L'antigène K de surface n'est pas toujours présent mais s'il est présent, il bloque l'agglutinabilité de l'antigène O.
Antigènes somatiques O
Il en existe plus de 150. Les antigènes somatiques sont composés de lipopolysaccharides complexes. Actuellement certains laboratoires d'analyses médicales utilisent l'agglutination avec des sérums pour déterminer le sérogroupe, mais cette technique est limitée par le nombre de plus en plus élevé de sérums à fabriquer, par la présence d'agglutinations croisées entre les antigènes O de E. coli, Shigella et ceux de Salmonella, et par le passage de la consistance crémeuse de la colonie à une consistance rugueuse ayant pour conséquence l’absence de synthèse de l'antigène O. C'est pour cette raison qu'une technique de sérotypage moléculaire a été développée.
L'antigène O fait partie du lipopolysaccharide (LPS) de la membrane externe (faisant partie intégrante de la paroi cellulaire fine) des bactéries à Gram négatif. Il contient un grand nombre d’unités répétées d’oligosaccharides de trois à six sucres dont la combinaison détermine la diversité des antigènes O. Les gènes codant les enzymes impliquées dans la synthèse de l’antigène O sont regroupés dans le groupe de gènes rfb.
Ce groupe rfb peut être amplifié spécifiquement grâce à un système d’amorces puis, après restriction par l’endonucléase MboII, un profil noté « R » peut être obtenu par électrophorèse, correspondant à un sérogroupe de E. coli[11]. Un profil d'électrophorèse est fonction de l’emplacement des sites de restriction propre à MboII. Ainsi tous les groupes de gènes correspondant à un antigène somatique auront un profil de restriction qui lui est propre. Ce profil R sera ensuite analysé avec le logiciel Taxotron puis comparé à une base de données, en perpétuel développement. Par exemple, le profil R aura un numéro R111, correspondant au sérogroupe O111 obtenu avec le sérum.
Antigènes flagellaires H
Les antigènes H ne servent pas à l'identification des E. coli pathogènes mais présentent un grand intérêt au point de vue épidémiologique : l'identité de l'antigène H constitue un élément pour assurer qu'il s'agit d'une même souche.
La diversité des antigènes H est due aux différents types de flagelline composant la structure du flagelle. C'est le flagelle qui permet la mobilité bactérienne. Le typage s'effectue également par séroagglutination, mais n’est développé que dans de très rares laboratoires dans le monde. Cependant, certaines souches perdent leur mobilité et sont classées comme non mobiles (NM ou H-). Une technique de sérotypage moléculaire a donc été également développée pour déterminer l'antigène H.
L'antigène H est codé par le gène fliC. Les parties N et C terminales de la flagelline sont très conservés et c'est la partie médiane, plus variable, qui donne la spécificité de l'antigène H. Les E. coli immobiles possèdent également le gène fliC mais sont incapables de synthétiser un flagelle fonctionnel. Après amplification et restriction du gène fliC, il est possible de typer l'antigène H en comparant le profil obtenu à une base de données de profil-type[12]. Par exemple, le profil fliC (noté F) aura un numéro F8, correspondant au type H8 obtenu avec le sérum.
Antigènes de surface ou d'enveloppe K
Il existe trois types d'antigène K désignés par les lettres L, A ou B :
- l'antigène L est le plus fréquent mais est thermolabile (il est détruit en une demi-heure à 100 °C) ; donc le chauffage provoque une perte du pouvoir antigénique, du pouvoir de fixer les anticorps et du pouvoir de masquer l'antigène O ;
- l'antigène A est rare ; c'est un antigène capsulaire (les E. coli encapsulés sont relativement fréquents dans les infections urinaires). L'Ag A est très thermostable (il faut un autoclavage pour le détruire) ;
- l'antigène B est toujours présent chez les E. coli enthéropathogènes de gastro-entérite infantile. Il a une thermolabilité intermédiaire : après une demi-heure. à 100 °C, il reste toujours de l'antigène B mais l'antigène O peut entrer en contact avec le sérum par « trouage » de l'enveloppe, la fixation de l'anticorps est toujours positive mais le pouvoir antigénique se perd progressivement (en fonction de la durée de chauffage).
Différence entre l'antigène B et les antigènes A ou L, dans une population homogène sur boîte de Petri :
- 80 % de colonies + et 20 % de colonies - pour A ou L ;
- répartition homogène dans toute la population pour B.
Critères d'identification d'E. coli (démarche)
C'est une bactérie de la famille des Enterobacteriaceae ne possédant pas de désaminase, ce qui exclut les genres Proteus, Morganella et Providencia (typiquement ex-tribu des Proteae).
Elle fermente le glucose par la voie des acides mixtes (rouge de méthyle +, VP -) ce qui exclut les genres Klebsiella, Enterobacter, Hafnia et Serratia (typiquement groupe des KEHS, ex-tribu des Klebsielleae).
De plus,
- Fermentation du glucose en voie fermentative (Création de gaz dans un tube Durham).
- Production d'indole à partir du tryptophane.
- Ne produit pas d'H2S.
- Incapable d'assimiler le citrate comme seule source de carbone en aérobiose.
- Uréase -
- Indole +++
- Devient vert métallique sur gélose EMB (Eosin Methylene Blue Agar)
- Devient rose sur milieu MacConkey
Génome
Le patrimoine génétique de la souche E. coli de laboratoire non pathogène a été entièrement séquencé en 1997. Son génome comprend 4,6 millions de paires de bases codant environ 4 200 protéines[13].
En 2001, le génome d'une souche d’E. coli entérohémorragique[14] (provoquant la maladie du hamburger) a été séquencé. Il comprend 5,5 millions de paires de bases codant 5 400 protéines. L'année suivante, le génome d'une souche d'E. coli provoquant des infections urinaires (cystite, pyélonéphrite) et des méningites néonatales, a été séquencé. Il comprend 5,2 millions de paires de bases codant 5 300 protéines.
La comparaison des génomes de ces trois souches d’E. coli révèle que seulement 40 % de leurs gènes sont communs — à titre de comparaison, 99 % des gènes de l'humain et des grands singes sont communs. Ceci témoigne du remarquable potentiel évolutif et de la versatilité de ce taxon bactérien. En effet, les souches d’E. coli pathogènes ont acquis au cours de l’évolution un répertoire de gènes de virulence, qui leur permettent de coloniser de nouvelles niches écologiques en contournant les mécanismes de défense de l’hôte. L’expression d’un répertoire spécifique de facteurs de virulence est corrélée à une pathologie particulière et permet de définir différents pathovars (voir infra).
Plasticité du génome d’E. coli et pathogénicité
Le concept de la pathogénicité bactérienne résultant d’un processus multifactoriel, impliquant une myriade de gènes, dont l’expression est chorégraphiée par des processus de régulation est maintenant bien accepté. L’expression de ces gènes permet une adhésion plus efficace, ou l’invasion des tissus de l’hôte, et permet ainsi la colonisation de niches inaccessibles ou inhospitalières pour les E. coli commensaux. En ce sens, la pathogénicité peut être considérée comme un avantage sélectif, et le succès d’une souche d'E. coli en tant que pathogène requiert probablement l’acquisition et la sélection de gènes de virulence, envers des recombinaisons et des transferts génétiques non spécifiques. La plasticité du génome d’E. coli est à la base de ce processus. La séquence complète du génome de plusieurs souches d’E. coli montre la présence de nombreuses Séquence d'insertion (IS), de séquences bactériophagiques, ainsi que d'autres plages de séquences inusuelles qui témoignent de l’extraordinaire plasticité du génome de ce genre bactérien. Ce sont les isolats cliniques d’E. coli qui possèdent les plus grands génomes, alors que celui de l’E. coli de laboratoire, non pathogène, fait 4,63 Mb. Il apparaît ainsi que le fossé qui sépare les E. coli commensales des E. coli pathogènes est dû à l'acquisition de répertoires de gènes de virulence. Il se pourrait que l’acquisition de ces gènes soit facilitée par une importante aptitude à muter. En effet, plus d'1 % des isolats d’E. coli ou de Salmonella impliqués dans des infections alimentaires sont des « mutateurs » qui présentent une forte tendance à muter, un phénomène corrélé à une déficience dans certains systèmes de réparation de l’ADN. Les gènes de virulence sont le plus souvent localisés sur des éléments génétiques transmissibles comme des transposons, des plasmides ou des bactériophages. De plus, ils peuvent être regroupés sur de grands blocs d’ADN chromosomique appelés « îlots de virulence » ou «îlots de pathogénicité».
Les recherches génétiques sur la bactérie ont permis la découverte de l'ARNI ou ARN I[15] (à ne pas confondre avec l'ARN interférent, ARNi). C'est un ARN non codant qui est un répresseur antisens de la réplication de certains plasmides d'Escherichia coli, dont ColEl[16]. Celui-ci explique la neutralisation de certains gènes.
Escherichia coli, une bactérie commensale et un agent pathogène
Certaines souches spécialisées d’E. coli sont associées à des pathologies très diverses tant chez l’être humain que chez l’animal ; diarrhées, gastro-entérites, infections urinaires, méningites, septicémies, etc. Les techniques modernes de la biochimie, de la génétique, de la biologie moléculaire et de la microbiologie cellulaire ont permis d’identifier et d’analyser les mécanismes impliqués dans l’interaction des E. coli pathogènes avec leur hôte. Malgré la diversité des affections provoquées par les souches d’E. coli pathogènes, toutes ces souches utilisent une stratégie classique d’infection, commune à de nombreux autres agents pathogènes.
Comme la plupart des pathogènes des muqueuses, les souches d’E. coli responsables de diarrhées et d'infections extra-intestinales utilisent une stratégie d'infection dont les points clés sont les suivants: colonisation des muqueuses, éventuellement invasion des cellules, multiplication, évasion des défenses de l’hôte, dommages à l’hôte.
Pour survivre et se multiplier dans le tractus intestinal, les colibacilles doivent surmonter les premières lignes de défense de l’organisme hôte, à savoir le péristaltisme et l’effet de barrière de la microflore commensale. Cette microflore accapare les nutriments, produit des inhibiteurs et occupe les surfaces des muqueuses. L’effet de barrière est surmonté par les E. coli pathogènes grâce à des mécanismes qui sont connus en termes généraux : la compétition pour les sources de carbone, de fer, d’énergie sous des conditions anaérobies, la production de bactériocines, ainsi qu’un fort taux de croissance. L’étape de colonisation implique aussi la capacité à adhérer à la surface de la muqueuse intestinale. Virtuellement toutes les souches d’entérobactéries pathogènes ou non possèdent des systèmes d’adhésion, et il est bien établi que ce pouvoir d’adhésion est la caractéristique la plus conservée chez les E. coli pathogènes. Les structures bactériennes responsables de l’adhésion aux cellules épithéliales sont des adhésines fimbriaires (fimbriae ou pili) ou afimbriaires. Exposées à la surface des bactéries, ces adhésines interagissent avec des récepteurs de la membrane des cellules cibles. C’est ainsi que des souches d'E. coli pathogènes sont capables en partie grâce à leurs adhésines de coloniser des biotopes qui ne sont normalement peu ou pas colonisés par les E. coli commensales. Par exemple, les E. coli responsables d'infections urinaires déploient des pili « P » (pili associés aux pyélonéphrites) qui reconnaissent des glycolipides à la surface des cellules épithéliales du tractus urinaire.
La multiplication est essentielle dans le processus de pathogénicité ; on conçoit en effet qu’une multiplication rapide est un avantage pour la colonisation, ainsi que pour causer des dommages avant que le système immunitaire n'entre en action. Une multiplication lente, voire son arrêt, peut aussi constituer un avantage dans la persistance des pathogènes qui causent des maladies chroniques.
Un autre point essentiel dans le processus de pathogénicité est l’interférence des E. coli pathogènes avec le système immunitaire de l’hôte. On sait par exemple que certains types de lipo-polysaccharides (LPS ; antigène « O ») présents à la surface des bactéries les protègent de l’action lytique du complément, de la fixation des anticorps et de la phagocytose. Les capsules polysaccharidiques (antigènes « K ») qui sont sécrétées à la surface de certaines souches d’E. coli pathogènes (principalement celles causant des affections extra-intestinales) peuvent participer à l’évasion des défenses de l’hôte. Les capsules K1 et K5, qui comportent des homologies avec des molécules eucaryotes (les adhésines n-CAM et les héparanes), présentent ainsi une faible immunogénicité. Les variations antigéniques de certaines molécules protéiques de surface (comme les pili), peuvent également participer à l’évitement des défenses immunitaires.
La première étape de colonisation effectuée, certaines souches pathogènes produisent de puissantes toxines, ces dernières pouvant être responsables à elles seules des dommages infligés à l’hôte. D’autres souches pathogènes détournent à leur avantage des fonctions cellulaires essentielles, afin de survivre et persister. Ainsi, en altérant le cytosquelette cellulaire, elles peuvent adhérer très fortement à la surface cellulaire (on parle d’adhésion « intime »), voire pénétrer dans les cellules des muqueuses et s’y multiplier, telles Shigella flexneri ou Salmonella typhimurium.
Sur la base de ces modes d’interaction et des signes cliniques de l’infection, les souches d’E. coli inductrices de diarrhées peuvent être actuellement classées en cinq pathovars : E. coli entérotoxigéniques (ETEC), E. coli entéroinvasives (EIEC), E. coli entéropathogènes (EPEC), E. coli entérohémorragiques[14] (EHEC) et E. coli entéroaggrégatives (EAggEC). Outre les E. coli induisant des diarrhées, on distingue aussi le pathovar des E. coli pathogènes extraintestinales (ExPEC) impliquées dans des affections non-intestinales: infections urinaires, méningites, septicémies, mammites...
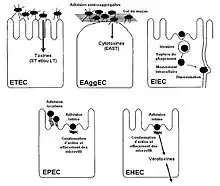
E. coli de gastro-entérites infantiles
Une dizaine de types particuliers d'E. coli, caractérisés par leurs antigènes, peuvent causer un syndrome entéritique grave avec toxicose et déshydratation chez les jeunes enfants. On admet généralement que ces colibacilles ne sont pathogènes qu'en dessous de l'âge de 2 ans mais c'est surtout chez les nouveau-nés et plus particulièrement chez les prématurés que ces germes se manifestent de la façon la plus sévère. L'allaitement maternel confère une certaine protection contre ces infections[réf. souhaitée] (rôle des bifides de la flore intestinale).
Dans les collectivités de nourrissons (maternités, services pédiatriques), la maladie prend généralement une allure épidémique : il s'agit dans ces cas de souches à la fois virulentes et multirésistantes réalisant le tableau d'une véritable infection nosocomiale.
E. coli entérotoxigéniques (ECET)
Les ECET sont une cause majeure de diarrhée aqueuse chronique avec déshydratation chez les enfants de bas âge (moins de 3 ans) dans les pays en voie de développement, et sont aussi responsables de la « diarrhée des voyageurs » (ou « turista »). Des ECET sont également une cause fréquente de diarrhées néonatales souvent fatales chez les animaux d’élevage (veau, agneau, porcelet).
Les ECET colonisent essentiellement la partie proximale de l’intestin grêle, grâce à leurs « facteurs de colonisation » (CFAx et CSx) qui sont des adhésines fimbriaires. Les ECET n’induisent pas d’altérations histologiques marquées de la muqueuse. Le pouvoir pathogène des ECET s’explique principalement par la sécrétion des toxines thermostables (ST) et/ou thermolabiles (LT). La toxine LT, après endocytose, ADP-ribosyle la sous-unité alpha de la protéine hétérotrimérique Gs. Il s’ensuit l’hyper-activation de l’adénylate cyclase, l’augmentation de la concentration du second messager AMPc, et la phosphorylation de transporteurs membranaires - particulièrement le « CFTR », le régulateur de la conductance membranaire impliqué dans la mucoviscidose. Cette action se traduit par une sécrétion d’ions chlorure et une inhibition de l’absorption de chlorure de sodium par les cellules intestinales, ce qui provoque la diffusion osmotique d’eau vers la lumière intestinale. L’action des toxines ST est moins connue. En se fixant à leur récepteur à la surface des cellules intestinales (une guanylate cyclase), elles induisent des concentrations accrues en GMPc, ce qui résulte également en l’activation du CFTR, l’altération de l’homéostasie intestinale, et une diarrhée osmotique. Ainsi, c’est l’action des toxines ST et LT qui explique le tableau clinique de l’infection : diarrhée aqueuse peu fébrile, nausées et crampes abdominales.
E. coli entéroinvasives (ECEI)
Les ECEI sont responsables de syndromes dysentériques caractérisés par une forte fièvre, des crampes abdominales et des nausées, accompagnés d’une diarrhée aqueuse qui évolue rapidement en une dysenterie (selles contenant du sang et du mucus). Les ECEI ont des caractères biochimiques, antigéniques, génétiques et fonctionnels très proches de ceux des Shigella, et mettent en œuvre un mécanisme de pathogénicité similaire. Les ECEI et les Shigella envahissent la muqueuse intestinale au niveau du côlon, s’y multiplient, provoquent la mort cellulaire et déclenchent une intense réaction inflammatoire. Le processus d’invasion est complexe et multifactoriel, sous la dépendance de loci chromosomiques et d’un plasmide de virulence (pInv ; ~220 kb). En ce qui concerne Shigella, le processus d’invasion peut être résumé ainsi : lors du contact avec les cellules épithéliales, les bactéries sécrètent des « invasines » (Ipa), qui interagissent avec la surface cellulaire et provoquent un réarrangement localisé du cytosquelette aboutissant à la pénétration de la bactérie dans la cellule. Une fois en position intracellulaire, la membrane de la vacuole est rapidement lysée grâce à une hémolysine de contact, libérant les bactéries dans le cytoplasme où elles peuvent se multiplier. Puis, les bactéries induisent la polymérisation de l’actine cellulaire à un de leurs pôles (grâce à IcsA) pour se mouvoir et se disséminer de cellule en cellule. Le processus d’invasion mis en jeu par les EIEC reste à être élucidé, mais les données actuelles indiquent qu’il est probablement identique à celui de Shigella. Les ECEI et les Shigella élaborent également une ou plusieurs entérotoxines qui seraient impliquées dans l'épisode de diarrhée aqueuse qui précède la dysenterie.
E. coli entéropathogènes (ECEP)
Les ECEP sont responsables de gastro-entérites infantiles (GEI). On admet généralement que ces colibacilles ne sont pathogènes qu'en dessous de l'âge de deux ans. Les principaux sérotypes impliqués sont O111 B4 et O119 B14. Le plus fréquent dans l'UE ces dernières années est l'O111 B4 mais on commence à parler d'autres types (O55 et O26). Chez l'adulte, en principe, les E. coli de GEI ne sont pas pathogènes. Certains avancent que certaines diarrhées du voyageur seraient dues à des types d'E. coli particuliers inconnus dans la région où vit le voyageur.

Ce n'est que chez les nourrissons en bas âge que la maladie prend une allure grave et épidémique (surtout et presque uniquement en milieu hospitalier). Il s'agit de diarrhées avec déséquilibre de la balance ionique ; d'où le plus important est de rétablir la balance ionique ; l'antibiothérapie interviendra secondairement.
E. coli entérohémorragiques (ECEH)
Les ECEH sont responsables de colites hémorragiques[17]. Le principal réservoir de ces bactéries est le tube digestif des bovins ; la contamination humaine se fait par l'intermédiaire d'aliments, principalement la viande de bœuf hachée et le lait cru. Le sérotype O157 est le plus fréquent (ex : sérotype O157:H7 pour la « maladie du hamburger »[18]). Il est responsable d'épidémies. Les ECEH produisent une verotoxine (ou Shiga-toxine) qui peut entraîner un syndrome hémolytique et urémique (SHU). Des intoxications à ECEH se sont déclarées à la suite de l'ingestion de viande contaminée et insuffisamment cuite (hamburger). Une intoxication a eu lieu en France en 2005.
Les cytotoxines (vérotoxines) sont à l'origine de la destruction des cellules intestinales. Les symptômes peuvent aller de la diarrhée simple à une diarrhée sanglante et abondante. Les manifestations sont plus graves chez les enfants de moins de 8 ans et chez les personnes de plus de 65 ans.
Le syndrome hémolytique et urémique (SHU) se manifeste entre autres par une anémie hémolytique, une thrombopénie et une insuffisance rénale aiguë.
E. coli entéroaggrégatives (ECEAgg)
Jusqu’au début des années 1980, les souches d’E. coli inductrices de diarrhées étaient classées en trois catégories ; les ECET, les ECEI et les ECEP, ces dernières étant alors caractérisées essentiellement par leur appartenance à des sérotypes distinctifs. Au début des années 1980, il a été constaté que la plupart des souches classées dans les ECEP adhèrent sur des cellules de lignée. Par la suite, trois modes distincts d’adhésion aux cellules ont été décrits : l’adhésion « localisée », l’adhésion « diffuse », et l’adhésion « agrégative », ce qui a permis de différencier un nouveau pathovar d’E. coli diarrhégéniques, les EAggEC. Les ECEAgg sont actuellement définies comme des souches qui ne sécrètent pas les entérotoxines LT ou ST, et qui adhèrent aux cellules de culture en formant des images « d’amas de briques » (adhésion agrégative). Il est probable que cette définition inclut des souches non pathogènes ; l’hétérogénéité de ce groupe a d’ailleurs été confirmée par des études épidémiologiques et des infections expérimentales d’adultes volontaires. Néanmoins, les ECEAgg sont de plus en plus reconnues comme étant responsables de retards de croissance et de diarrhées persistantes dans les pays en voie de développement ainsi que dans les pays industrialisés. Des fimbriae (AAF/I et AAF/II) responsables de l’adhésion agrégative ont été décrits, mais ils sont présents dans une minorité d’isolats d’ECEAgg. Ceci suggère que la colonisation du tractus digestif serait due à un ou plusieurs fimbriae, comme dans le cas des ECET. Les ECEAgg adhèrent sur la muqueuse intestinale, enchâssées dans un biofilm de mucus produit par les cellules en gobelets de la muqueuse. Environ 40 % des souches d’ECEAgg produisent l’entérotoxine EAST1 (pour « EAggEC ST-like toxin »), qui présente environ 50 % d’homologie avec la toxine ST des ECET et 30 % avec les toxines amnésiantes de guanylate cyclase de coquilles STJ. La contribution de ces toxines au pouvoir pathogène n’est pas encore établie. Le gène codant la toxine EAST1 ou ses variants a été trouvé dans des souches ECEP et ECET, ainsi que dans des souches d’E. coli pathogènes ou non, qui ne possèdent pas les gènes marqueurs des pathovars classiques.
E. coli à adhésion diffuse (ECDA)
Les ECDA ont initialement été classées avec les E.Coli entéro-pathogènes du fait de leur adhésion aux cellules Hep-2 (lignée cellulaire d'origine tumorale) mais forment actuellement un groupe à part. En effet, avec la découverte des ECEAgg (E.Coli entéroaggrégatives) les ECDA ont été différenciées par leur phénotype d'adhésion n'impliquant pas d'agrégats microbiens et formant des lésions diffuses sur les cellules Hep-2. Les ECDA sont essentiellement responsables de pathologies extra-intestinales (infections urinaires : cystites, pyélonephrites). Cependant, les études menées ont démontré que selon la tranche d'âge les ECDA pourraient également induire des signes cliniques intestinaux (diarrhée) - la sensibilité aux ECDA serait étroite, située entre un et cinq ans.
E. coli pathogènes extra-intestinales
Les E. coli sont pathogènes dans des localisations extra-intestinales : méningites, abcès, péritonites, septicémies et surtout infections urinaires (UPEC).
Origine hématogène
Les germes charriés par la circulation (bactériémie) viennent se fixer au niveau de tractus urinaire si une cause favorisante permet leur implantation : rétrécissement, malformation, calcul. Ces causes entraînent une stagnation.
Origine ascendante
Cette origine est la plus fréquente. Des germes d'origine fécale en provenance de la région péri-anale remontent dans la vessie, surtout chez les femmes. L'origine explique la fréquence des infections urinaires par germes fécaux surtout les entérobactéries : colibacilles, klebsiella, proteus mais aussi les entérocoques.
Origine ascendante iatrogène
Sondage, cathétérisme. Ces manœuvres peuvent introduire des germes à partir de l'extérieur, en particulier le pyocyanique.
Diagnostic bactériologique des infections urinaires
Le prélèvement devrait idéalement se faire par ponction sus-pubienne ou par sondage. Comme la première méthode n'est pas toujours réalisable en pratique et qu'il est déconseillé d'effectuer systématiquement la seconde (risque d'infections iatrogènes), on se contente généralement d'utiliser un échantillon récolté à la miction (partie moyenne du jet) après toilette des organes génitaux externes. Cette façon de procéder n'empêche cependant pas l'urine d'être souillée par des germes des orifices externes, gênants pour l'interprétation des résultats. De ce fait, ce prélèvement n'est valable que moyennant une analyse bactériologique quantitative. On admet généralement que la présence de moins de 10 000 germes par ml d'urine correspond à une contamination externe, alors que plus de 100 000 germes traduisent une bactériurie significative. Entre ces deux nombres, le résultat est plus difficile à interpréter. Pour que cette analyse quantitative soit faite dans de bonnes conditions, il faut que l'échantillon soit frais. Une méthode simple de triage quantitatif est la culture sur lames gélosées (lame de verre tapissée de gélose) plongées dans l'urine : le nombre de colonies apparaissant après incubation reflète le nombre de germes par ml d'urine. Cette méthode doit être complétée par l'identification des germes et la réalisation de l'antibiogramme. Des méthodes plus rigoureuses peuvent être appliquées au laboratoire par l'ensemencement d'un volume déterminé d'urine diluée sur une boîte de Petri et par la numération des colonies obtenues.
- Chez les nouveau-nés, particulièrement sensibles à E. coli, on observe des manifestations graves (méningites, septicémies).
- Certains types de colibacilles sont spécifiquement entéro-pathogènes pour les nourrissons.
- Les E. coli étant d'origine fécale peuvent servir de révélateurs dans certaines analyses de bactériologie alimentaire : dans le contrôle d'eau potable, la présence d'E. coli fait supposer une contamination fécale.
- La présence d'E. coli dans un lait pasteurisé peut indiquer un degré insuffisant de pasteurisation.
Prophylaxie
Les EHEC résistent au froid, mais sont détruites par la cuisson.
Il faut donc rappeler, notamment pour les populations sensibles :
- les enfants de moins de 5 ans (et par mesure de précaution, les enfants jusqu’à 15 ans) ;
- les personnes âgées de plus de 65 ans ;
- les personnes immunitairement déficientes.
Il est impératif :
- de consommer la viande hachée dans les 3 heures qui suivent le hachage ou de la congeler immédiatement ;
- de la conserver à -18 °C maximum ou de la faire attendre à 4 °C maximum ;
- de bien cuire la viande hachée (au moins > 65 °C à cœur)[19], ce qui correspond à un cœur du steak haché qui ne doit être ni rouge, ni rosé, mais cuit à point ;
- selon l’Agence nationale de sécurité sanitaire de l'alimentation, de l'environnement et du travail, « Il est nécessaire de bien cuire à cœur les viandes hachées ou produits à base de viande hachée consommés par les jeunes enfants et les personnes âgées. Une température à cœur de 70 °C pendant deux minutes pour la cuisson des steaks hachés de bœuf est recommandée »[20].
Dans certains pays, et en particulier aux États-Unis ou au Royaume-Uni, les autorités sanitaires préconisent une cuisson des viandes hachées à au moins 70 °C à cœur maintenus pendant deux minutes.
Pour une pièce de viande non hachée, les bactéries, présentes uniquement à la surface, sont facilement détruites lors de la cuisson par un passage, même rapide, à la poêle. Mais, dans le cas de la viande hachée, des bactéries sont susceptibles d’être présentes en profondeur et ces dernières ne seraient pas détruites par une cuisson rapide (viande bleue ou saignante) : une cuisson plus longue est indispensable.
Pour que la cuisson des steaks hachés surgelés destinés aux enfants permette d’atteindre en quelques minutes une température à cœur suffisante pour détruire les bactéries, il est recommandé de décongeler les steaks avant leur cuisson. La décongélation doit alors être faite soit en conservant les steaks le temps nécessaire dans la partie la plus froide du réfrigérateur dans leur conditionnement protecteur d’origine, soit dans un four à micro-ondes. La décongélation ne doit jamais être faite à température ambiante.
De plus, les légumes, mais aussi les fruits et les herbes aromatiques, en particulier ceux qui vont être consommés crus, doivent être soigneusement lavés puis épluchés si nécessaire avant leur préparation et leur consommation.
Les aliments crus doivent être conservés séparément des aliments cuits ou prêts à être consommés pour éviter les contaminations croisées (bonne séparation de la viande crue des autres aliments dans le réfrigérateur[21]).
Enfin, les recommandations d’hygiène courantes doivent également être appliquées afin d’éviter la transmission de personne à personne et la contamination croisée des aliments : lavage soigneux des mains et séchage avec un torchon propre avant de préparer le repas, plus particulièrement pour les jeunes enfants ; lavage soigneux des couteaux, planches à découper et autres ustensiles avant de cuisiner ; et lavage soigneux des mains après être allé aux toilettes[22].
Traitement
Antibiotique
Résistances naturelles : Escherichia coli est sensible à toutes les bêta-lactamines malgré la production d'une céphalosporinase chromosomique non inductible de type AmpC qui peut entraîner chez certaines souches une réduction de la sensibilité aux aminopénicillines, à leurs associations au clavulanate et/ou au C1G.
Phagothérapie
La phagothérapie est utilisée chez l'humain dans certains pays comme la Russie et la Géorgie, mais la société ne communique ni sur la composition de ses préparations ni sur ses résultats. Des données regroupant différents petits tests cliniques montreraient une éradication du pathogène dans 40 % des cas, avec un taux de récupération des patients de 18 %. Globalement, ce secteur de la recherche souffre d'un manque d'essais contrôlés et du désintérêt de l'industrie pharmaceutique, seule l'industrie alimentaire ayant déposé des brevets de phages qualifiés d'additifs alimentaires, sans doute pour contourner une réglementation contraignante. L'autre contrainte est le manque de connaissances sur les mécanismes d'action[23].
Espoir de vaccin
Des chercheurs de l'université du Michigan (États-Unis) ont publié le dans la revue PLoS Pathogens les résultats prometteurs obtenus sur des souris qui créent une forte résistance immunitaire contre la bactérie Escherichia coli.
L'équipe dirigée par le Dr Mobley a analysé 5 379 protéines bactériennes susceptibles d'induire une réponse immunitaire. Sur les six substances retenues comme candidats vaccins et injectées par voie nasale à des souris, trois ont permis de ne pas contracter d'infection.
Les chercheurs américains ont établi que ces protéines ciblent des récepteurs du fer situés à la surface de la bactérie et qui jouent un rôle crucial dans la propagation de l'infection. Surtout, la réponse immunitaire est induite aussi bien dans la muqueuse nasale, où l'injection est pratiquée, que dans celle de l'appareil urinaire.
L'équipe du Dr Mobley a également montré que ses candidats vaccins sont efficaces sur plusieurs souches d'E. coli et envisage de procéder à des essais cliniques de phase 1 sur des volontaires humains.
Résistances acquises
- Pénicillinase de haut niveau chez 50 % des E. coli.
- Hyperproduction de céphalosporinase chez 5 % des E. coli.
- Bêta-lactamase à spectre étendu chez 1 % des E. coli.
La souche E. coli ST131, multirésistante aux antibiotiques, est impliquée dans des infections urinaires. Le gène qui leur confère cette grande résistance se transmet facilement entre individus E. coli par plasmides[24],[25]. Il code l'enzyme aminoglycoside-(6')-N-acetyltransferase (aac(6’)-Ib-cr) qui inactive la ciprofloxacine, un antibiotique à large spectre de la classe des fluoroquinolone (FQ).
Prédation
Escherichia coli étant une bactérie, elle est sensible aux bactériophages comme les phages T4 et lambda.
Production et utilisation commerciale
Escherichia coli est très utilisée pour sa capacité à produire l'enzyme de restriction EcoRI.
En 2008, après un premier pilote industriel (installé en 2007 dans la raffinerie de Pomacle-Bazancourt près de Reims), E. coli est également utilisée pour produire de l'acide succinique à partir de sucres et résidus lignocellulosiques fermentés en atmosphère enrichie en CO2[26]. Cette raffinerie dispose d'une capacité de production de 2 000 t/an.
L'acide succinique est notamment utilisé comme excipient dans le vaccin Prevenar13 de Wyeth Lederle Vaccines SA (Pfizer)[27].
Escherichia coli est aussi utilisé comme hôte hétérologue pour produire de nombreuses protéines[28].
Notes et références
- (en) « Bacteria - Diversity of structure of bacteria », sur Encyclopedia Britannica (consulté le )
- (en) H. Bremer et P.P. Dennis, « Modulation of chemical composition and other parameters of the cell by growth rate », dans F.C Neidhardt, R Curtiss, III, J.L Ingraham, E.C.C Lin, K.B Low, B Magasanik, W.S Reznikoff, M Riley, M Schaechter et H.E Umbarger, Escherichia coli and Salmonella typhimurium Cellular and Molecular Biology, Washington, DC, ASM Press, (ISBN 0-914826-89-1, lire en ligne), p. 1553-1569.
- (en) Olivier Tenaillon, David Skurnik, Bertrand Picard et Erick Denamur, « The population genetics of commensal Escherichia coli », Nature Reviews Microbiology, vol. 8, no 3, , p. 207–217 (ISSN 1740-1534, DOI 10.1038/nrmicro2298, lire en ligne, consulté le )
- P. Raibaud et R. DUCLUZEAU, « Etude de la colonisation bactérienne du tractus gastro-intestinal à l'aide de modèles expérimentaux », Rev.sci.tech. Off. int. Ep, .
- Kathrine Trivière, Modifications de l'écosystème microbien de l'oreille du chien en cas d'otite externe et enseignements pour le traitement, Liège, Belgique, Université de Liège, (lire en ligne)
- (en) V. I. Bruskov, Zh. K. Masalimov et A. V. Chernikov, « [No title found] », Doklady Biochemistry and Biophysics, vol. 384, nos 1/6, , p. 181–184 (DOI 10.1023/A:1016036617585, lire en ligne, consulté le ).
- (en) L. Le Minor, P. Nicolle, R. Buttiaux, Y. Chabbert et S. Le Minor, « Studies on Escherichia coli Isolated in Infantile Gastroenteritis », dans Ann. Inst. Pasteur, vol. 86, 1954, pp. 204-226 et vol. 87, 1954, pp. 175-184, 1954 ; (en) L. Le Minor, R. Buttiaux, B. Gaudier, S. Le Minor et P. Nicolle, « Epidemiologic Research on Gastroenteritis Due to Escherichia coli in a Hospital in Northern France », dans Arch. Mal. Appar. Dig. Mal. Nutr., vol. 45, 1956, pp. 225-247.
- Centre National de Référence des E. coli, www.pasteur.fr.
- LS9.
- E. Stewart et al. PLoS Biology February 2005
- Coimbra et al., 2000.
- Machado et al, 1998.
- (en) FR Blattner, G Plunkett, CA Bloch, NT Perna, V Burland, M Riley, J Collado-Vides, JD Glasner, CK Rode, GF Mayhew, J Gregor, NW Davis, HA Kirkpatrick, MA Goeden, DJ Rose, B Mau et Y Shao, « The complete genome sequence of Escherichia coli K-12. », Science, vol. 277, , p. 1453-1462 (PMID 9278503).
- [vidéo] [audio] Une bactérie meurtrière se propage en Allemagne, tsrinfo.ch, .
- Hisao Masukata et Jun-ichi Tomizawa, « Control of primer formation for ColE1 plasmid replication: Conformational change of the primer transcript », Cell, vol. 44, no 1, , p. 125–136 (ISSN 0092-8674, DOI 10.1016/0092-8674(86)90491-5, lire en ligne, consulté le )
- Lin He, Fredrik Söderbom, E. Gerhart H. Wagner et Uta Binnie, « PcnB is required for the rapid degradation of RNAI, the antisense RNA that controls the copy number of CoIE1-related plasmids », Molecular Microbiology, vol. 9, no 6, , p. 1131–1142 (ISSN 0950-382X et 1365-2958, DOI 10.1111/j.1365-2958.1993.tb01243.x, lire en ligne, consulté le )
- [PDF] Surveillance des infections à E. coli entérohémorragiques (EHEC) et du syndrome hémolytique et urémique (SHU) en Europe, Euro Surveillane, Bulletin européen sur les maladies transmissibles, vol. 2, no 12, décembre 1997.
- (en) [PDF] Hamburger Disease (E. coli O157:H7).
- Comment se protéger d'Ecoli? - Consommation du steak haché sur le site La-viande.fr.
- Anses.
- Les consommateurs et la qualité sanitaire, La-viande.fr.
- Centre d'Information des Viandes, www.civ-viande.org.
- (en) Harald Brüssow, « What is needed for phage therapy to become a reality in Western medicine? », Virology, special issue: viruses of microbes, vol. 434, no 2, , p. 138–142 (ISSN 0042-6822, DOI 10.1016/j.virol.2012.09.015, lire en ligne, consulté le )
- https://www.pourquoidocteur.fr/Articles/Question-d-actu/38356-E-coli-gene-rend-bacterie-insensible-antibiotiques-identifie
- (en) Minh-Duy Phan, (…) Mark A. Schembri, « Plasmid-Mediated Ciprofloxacin Resistance Imparts a Selective Advantage on Escherichia coli ST131 », ASM Journals Antimicrobial Agents and Chemotherapy, vol. 66, no 1, (DOI 10.1128/AAC.02146-21, lire en ligne, consulté le ).
- Démarrage de la première usine commerciale d'acide succinique d'origine vététale, Biofutur, mars 2010, p. 21.
- [PDF] RCP du Prévenar13, Agence européenne des médicaments.
- « Genosphere : Expression protéique chez E. coli », sur www.genosphere-biotech.com (consulté le ).
Voir aussi
Articles connexes
Bibliographie
- James B. Kaper, James P. Nataro et Harry L. T. Mobley, « Pathogenic Escherichia coli », Nature Reviews Microbiology, vol. 2, p. 123-140, 2004 (ISSN 1740-1534) [(en) texte intégral].
- Richard d'Ari et Guennadi Sezonov, Biologie et génétique d'Escherichia coli, Paris, éditions Belin, 2008 (ISBN 978-2-7011-3642-4).
Liens externes
- Consommation de viande - Comment se protéger d'E.Coli, La-Viande.fr.
- Portail de la microbiologie