Protéase 3C-like
Protéase de type 3C
La protéase 3C-like[alpha 1] (en français « de type 3C ») ou 3CLpro ou Mpro (pour Main protease, en français « protéase principale ») ou endopeptidase C30 est une enzyme codée par le génome des coronavirus. La fonction de cette protéase est de cliver des polyprotéines produites dans les cellules infectées par le virus, au cours de la phase de réplication de ce virus[1]. Cette enzyme peut couper à l'intérieur des chaînes peptidiques (et pas seulement cliver les résidus terminaux), c'est donc une endopeptidase (ou protéinase). Il s'agit également d'une protéase à cystéine[alpha 2].
Dénomination
Le nom 3C-like (en français « de type 3C ») provient des similitudes de cette molécule avec la picornaïne 3C (en), protéase codée dans le génome des picornavirus. La dénomination « 3C » des picornavirus a été actée dans une convention établie à la suite d'une décision prise lors de la 3e réunion du Groupe d'étude européen sur la biologie moléculaire des picornavirus[alpha 3] (EUROPIC) tenue du 5 au 10 septembre 1983 à Urbin en Italie. Cette dénomination est liée à la position de la protéase dans la polyprotéine des picornavirus selon un schéma idéalisé de cette polyprotéine, appelé diagramme L434, selon lequel cette polyprotéine est constituée d'une séquence « leader » (L) suivie de trois régions (P1, P2 et P3) comportant respectivement 4, 3 et 4 protéines. La protéase 3C est située dans la troisième région (d'où le chiffre 3), en troisième position (d'où la lettre C)[2].
Dans la nomenclature EC, la protéase 3C-like porte la référence EC .
Dans la littérature scientifique on trouve parfois l'appellation 3-chymotrypsin-like, 3CLpro présente en effet quelques similitudes avec la chymotrypsine, une peptidase du pancréas.
Historique
En 1989, des chercheurs américains et soviétiques décodent le gène 1 du coronavirus murin[alpha 4]. Ce travail montre la présence d'une séquence codant potentiellement une protéase du même type que les protéases 3C codées dans le génome des picornavirus[3]. En 1995, une équipe de chercheurs américains confirme la production d'une protéase de type 3C à partir du génome du virus MHV-A59 (une souche du coronavirus murin), elle confirme également l'existence d'au moins un site de clivage dans la polyprotéine virale. L'étude a aussi démontré que les résidus d'acides aminés His41 et Cys145 étaient essentiels pour l'activité catalytique de la protéase[4].
Génomique
Le génome des coronavirus est formé d'un simple brin d'ARN positif qui contient, à son extrémité 5’, un cadre de lecture ouvert codant les protéines non structurales du complexe de réplicase-transcriptase, dénommé ORF1, qui contient lui-même deux cadres de lecture ouverts qui se chevauchent (nommés ORF1a et ORF1b) et qui codent deux polyprotéines appelées pp1a (codée par ORF1a) et pp1ab (codée par ORF1a et ORF1b). ORF1a se termine par un codon-stop et la traduction de ORF1b ne peut donc se faire que lorsque se produit un décalage du cadre de lecture (d'un nucléotide) causé par la séquence glissante U_UU.A_AA.C[alpha 5] suivie quelques nucléotides plus loin par un pseudonœud, sur le brin d'ARN. Ce décalage permet de ne pas interpréter le codon-stop et donc de continuer la traduction du brin d'ARN. Le décalage se produit en moyenne dans environ 30 % des traductions de l'ARN, les protéines codées par ORF1a seront donc produites en beaucoup plus grand nombre que celles codées par ORF1b[5]. Les polyprotéines contiennent chacune deux protéases : la protéase principale 3CLpro (en 5e position[alpha 6]) et la protéase papain-like[alpha 7] PLpro (en 3e position).
Structure
| Pfam | PF05409 |
|---|---|
| InterPro | IPR008740 |
| PROSITE | PS51442 |
| MEROPS | C30 |
| SCOP | d1q2wb1 |
| SUPERFAMILY | d1q2wb1 |
3CLpro est une protéine qui a été fortement conservée au cours de l'évolution, elle est très semblable d'un coronavirus à l'autre.
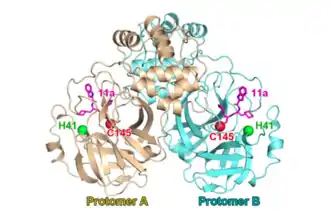
3CLpro n'est active que sous la forme d'un homodimère, c'est-à-dire constituée de deux sous-unités protéiques (monomères) identiques. Ces deux unités sont disposées perpendiculairement l'une par rapport à l'autre. Chaque sous-unité, d'environ 300 acides aminés, contient trois domaines différents[6], dénommés I, II et III.
Le domaine III, qui contient des structures en hélice α, intervient dans la régulation de la dimérisation de 3CLpro. Une chaîne longue relie le domaine III au domaine II. Les extrémités N et C de chacun des monomères sont situées à l'interface entre les deux monomères.
Le site de liaison au substrat (la molécule qui va être clivée) de chaque monomère est situé dans une cavité profonde, formée par deux tonneaux β comme dans la chymotrypsine, entre les domaines II et III. Le site actif catalytique est situé au centre de cette cavité[7], il est formé par un résidu d'acide aminé histidine situé en 41e position (His41) de la chaîne protéique et un résidu de cystéine situé en 145e position (Cys145)[8]. Contrairement à la chymotrypsine, il n'a pas été découvert de troisième résidu intervenant dans l'activité catalytique, il s'agit donc d'une dyade et non d'une triade catalytique. Une molécule d'eau est liée par une liaison hydrogène au résidu His41, cette molécule d'eau pourrait jouer le rôle habituellement tenu par le troisième résidu catalytique (acide aspartique) de la triade des protéases à sérine[9].
Activité
| N° EC | EC |
|---|
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
Zones clivées
Après la pénétration du coronavirus dans une cellule, ORF1 est traduit par un ribosome de la cellule hôte, qui produit alors les deux polyprotéines pp1a et pp1ab. Les polyprotéines sont ensuite clivées par les deux protéases qui sont elles-mêmes produites à partir de ces polyprotéines par auto-clivage. Ces clivages, qui ont lieu au niveau de liaisons peptidiques, produisent en tout 16 protéines virales fonctionnelles. La polyprotéine pp1a sera divisée en 11 protéines produites à partir de zones dénommées ns1 (côté N-terminal) à ns11 (« ns » pour non-structural), tandis que pp1ab sera divisée en 15 protéines (les dix mêmes premières protéines de pp1a ainsi que cinq autres supplémentaires) à partir de zones dénommées ns1 à ns10 et ns12 à ns16. À noter que la protéine de ns11 est la seule à n'être produite qu'á partir de pp1a.
3CLpro clive la polyprotéine pp1a en 7 endroits et pp1ab en 11 endroits, dont son propre clivage[10], tandis que PLpro clive la polyprotéine en 3 endroits. Les clivages de PLpro se produisent au niveau des sites ns1-ns2, ns2-ns3 et ns3-ns4 et ceux de 3CLpro au niveau des autres sites.
Catalyse
Les substrats spécifiques à 3CLpro possèdent tous un résidu de glutamine en position P1 (acide aminé en 1re position par rapport à la liaison peptidique qui va être clivée, côté extrémité N-terminale) et un résidu de petite taille (sérine, alanine, asparagine) en position P1’ (1re position de l'autre côté de la liaison peptidique qui va être clivée)[11]. Le clivage aura donc lieu entre ces deux résidus. En position P2, il y a souvent un résidu de leucine.
La première étape de la catalyse est la liaison du substrat avec le site actif de la protéase pour former un complexe de Michaelis. Le substrat est alors clivé à la liaison Gln–Ala (ou Gln–Ser ou autre…), la partie N-terminale du peptide clivé est libérée comme premier produit de la réaction, tandis que l'autre partie C-terminale réalise l'acylation (ajout d'un groupe acyle RCO—) du résidu Cys145 du site catalytique. La désacylation de Cys145 est effectuée dans une catalyse basique générale effectuée par l'autre résidu catalytique His41, ce qui permet de libérer la seconde partie du peptide, terminant le cycle catalytique[12].
Rôle dans l'inhibition de l'inflammation
Les protéases 3CLpro de certains coronavirus animaux sont impliquées dans la suppression de la production d'interféron de type I (protéines qui participent à la régulation de l'activité du système immunitaire). Des travaux réalisés en 2021 ont montré que les protéases 3CLpro des coronavirus humains SARS-CoV et SARS-CoV-2 suppriment elles aussi la production d'interféron de type I[13].
En 2020, des travaux sur le SARS-CoV-2, ont montré que 3CLpro peut également cliver des protéines de la cellule hôte qui jouent un rôle dans les processus inflammatoires. Ces protéines sont NLRP12 (en) et TAB1 (en)[14]. Les protéines NLRP (en) (protéines contenant les domaines NACHT (en)[alpha 8], LRR et PYD (en)[alpha 9]) font partie du système immunitaire inné, elles peuvent détecter des caractéristiques spécifiques des pathogènes comme les peptidoglycanes. TAB1 (Mitogen-activated protein kinase kinase kinase) est une protéine adaptatrice qui conduit à l’activation de la kinase TAK1 (Transformating growth factor-beta-Activated Kinase 1), qui fait partie de la voie NF-κB (Nuclear Factor-kappa B).
Cible thérapeutique
3CLpro est indispensable à la réplication des coronavirus et elle n'est pas présente naturellement dans les cellules humaines non infectées, de ce fait les inhibiteurs de cette protéase sont des candidats très intéressants pour combattre les infections de coronavirus pathogènes[15], comme le SARS-CoV ou le SARS-CoV-2.
Le 6 avril 2021 le groupe pharmaceutique américain Pfizer a dévoilé la structure d'un inhibiteur enzymatique de la protéase 3CLpro, le nirmatrelvir, anciennement PF-07321332, montrant une affinité nanomolaire avec la protéase et capable de supprimer la réplication du virus SARS-CoV-2 dans les cellules humaines à des concentrations sous-micromolaires. Des simulations de dynamique moléculaire semblent indiquer un mécanisme d'inhibition en deux étapes : d'abord une liaison non covalente avec la dyade catalytique de la protéase dans un état neutre, puis la formation d'un couple ionique thiolate-imidazolium et la relocalisation du ligand pour finaliser l'attaque électrophile[16].
Pfizer a développé un traitement contre la Covid-19, dénommé Paxlovid, mettant en œuvre cet inhibiteur en combinaison avec le ritonavir. En novembre 2021, Pfizer annonce que des résultats intermédiaires d'un essai clinique de ce traitement montre une efficacité de 89% sur la réduction du risque d'hospitalisation et de décès, lorsque le traitement débute moins de 5 jours après l'apparition des symptômes[17]. Le 22 décembre 2021, la FDA annonce l'autorisation d'utilisation en urgence (Emergency Use Authorization) du Paxlovid pour le traitement de la Covid-19 aux États-Unis, pour certaines catégories de patients[18]. L'agence européenne des médicaments avait fait de même le 16 décembre sur le territoire de l'Union européenne.
Notes
- Une protéase (ou peptidase) est une enzyme qui peut couper une chaîne peptidique entre deux résidus d'acides aminés, pour cela elle hydrolyse la liaison peptidique concernée.
- Une protéase à cystéine, parfois appelée protéase à thiol, est une peptidase dont le résidu d'acide aminé nucléophile de la triade (ou dyade) catalytique est une cystéine.
- Dénomination officielle en anglais : European Study Group on the Molecular Biology of Picornaviruses.
- À l'époque, connu sous le nom de virus de l’hépatite murine (MHV pour murine hepatitis virus en anglais).
- Les lettres U A C désignent respectivement les bases nucléiques uracile, adénine et cytosine de l'ARN viral, les espaces soulignés « _ » séparent les codons tels qu'ils sont lus par le ribosome lorsqu'il n'y a pas de décalage et les points « . » séparent les codons tels qu'ils sont lus en cas de décalage.
- En débutant depuis l'extrémité N-terminale du polypeptide.
- En français « de type papaïne ».
- NACHT est un domaine nucléoside-triphosphatase.
- PYD est un domaine pyrine.
Références
-
(en) Xiao Tao Lu, Amy C. Sims et Mark R. Denison, « Mouse Hepatitis Virus 3C-Like Protease Cleaves a 22-Kilodalton Protein from the Open Reading Frame 1a Polyprotein in Virus-Infected Cells and In Vitro », Journal of Virology, vol. 72, no 3, , p. 2265-2271 (PMID 9499085, PMCID 109524, DOI 10.1128/JVI.72.3.2265-2271.1998, lire en ligne
 )
) -
(en) Roland R. Rueckert et Eckart Wimmer, « Systematic Nomenclature of Picornavirus Proteins », Journal of Virology, vol. 50, no 3, , p. 957-959 (PMID 6726891, PMCID 255765, DOI 10.1128/jvi.50.3.957-959.1984, lire en ligne
 )
) -
(en) Han-Jung Lee, Chien-Kou Shieh, Alexander E. Gorbalenya, Eugene V. Koonin, Nicola La Monica, Jeremy Tuler, Anush Bagdzhadzhyan et Michael M. C. Lai, « The complete sequence (22 kilobases) of murine coronavirus gene 1 encoding the putative proteases and RNA polymerase », Virology, vol. 180, no 2, , p. 567-582 (PMID 1846489, PMCID 7131164, DOI 10.1016/0042-6822(91)90071-I, lire en ligne
 )
) -
(en) Yiqi Lu, Xiaotao Lu et Mark R. Denison, « Identification and characterization of a serine-like proteinase of the murine coronavirus MHV-A59 », Journal of Virology, vol. 69, no 6, , p. 3554-3559 (PMID 7745703, PMCID 189070, DOI 10.1128/JVI.69.6.3554-3559.1995, lire en ligne
 )
) - Sunil K. Lal, Molecular Biology of the SARS-Coronavirus, 19/02/2010, Springer Berlin Heidelberg, Allemagne, (ISBN 9783642036835), 364203683X, p. 78, aperçu consultable en ligne sur Google livres
- Neeraj Mittal, Sanjay Kumar Bhadada, O. P. Katare, Varun Karg, Covid-19 Diagnosis and Management, Bentham Books, partie 1, 04/06/2021, (ISBN 978-1-68108-808-2), aperçu consultable en ligne
-
(en) Bin Xiong et al., « A 3D model of SARS_CoV 3CL proteinase and its inhibitors design by virtual screening », Acta Pharmacologica Sinica, vol. 24, no 6, , p. 497-504 (PMID 12791174, lire en ligne
 )
) -
(en) Muhammad Tahir ul Qamar, Safar M. Alqahtani, Mubarak A. Alamri et Ling-Ling Chen, « Structural basis of SARS-CoV-2 3CLpro and anti-COVID-19 drug discovery from medicinal plants », Journal of Pharmaceutical Analysis, vol. 10, no 4, , p. 313-319 (PMID 32296570, PMCID 7156227, DOI 10.1016/j.jpha.2020.03.009, lire en ligne
 )
) -
(en) Mariusz Jaskolski, Zbigniew Dauter, Ivan G Shabalin, Miroslaw Gilski, Dariusz Brzezinski, Marcin Kowiel, Bernhard Rupp et Alexander Wlodawer, « Crystallographic models of SARS-CoV-2 3CLpro: in-depth assessment of structure quality and validation », IUCrJ, vol. 8, no Pt 2, , p. 238-256 (PMID 33708401, PMCID 7924243, DOI 10.1107/S2052252521001159, lire en ligne
 )
) -
(en) François-Xavier Cantrelle, Emmanuelle Boll, Lucile Brier, Danai Moschidi, Sandrine Belouzard, Valérie Landry, Florence Leroux, Frédérique Dewitte, Isabelle Landrieu, Jean Dubuisson, Benoit Deprez, Julie Charton et Xavier Hanoulle, « NMR Spectroscopy of the Main Protease of SARS-CoV-2 and Fragment-Based Screening Identify Three Protein Hotspots and an Antiviral Fragment », Angewandte Chemie International Edition, vol. 60, no 48, , p. 25428-25435 (PMID 34570415, PMCID 8653025, DOI 10.1002/anie.202109965, Bibcode 2021AngCh.13325632C, lire en ligne
 )
) - L. Enjuanes Sànchez, Coronavirus Replication and Reverse Genetics, Physica-Verlag, 2006, page 72, aperçu consultable en ligne avec l'autorisation de l'éditeur
- Gu-Gang, Chang, Quaternary Structure of the SARS Coronavirus Main Protease, Molecular Biology of the SARS-Coronavirus, pp. 115–128, 22/07/2009, Springer, Berlin, Heidelberg, consultable en accès libre en ligne
-
(en) Sin-Yee Fung, Kam-Leung Siu, Huayue Lin, Man Lung Yeung et Dong-Yan Jin, « SARS-CoV-2 main protease suppresses type I interferon production by preventing nuclear translocation of phosphorylated IRF3 », International Journal of Biological Sciences, vol. 17, no 6, , p. 1547-1554 (PMID 33907518, PMCID 8071772, DOI 10.7150/ijbs.59943, lire en ligne
 )
) -
(en) Mehdi Moustaqil, Emma Ollivier, Hsin-Ping Chiu, Sarah Van Tol, Paulina Rudolffi-Soto, Christian Stevens, Akshay Bhumkar, Dominic J. B. Hunter, Alexander N. Freiberg, David Jacques, Benhur Lee, Emma Sierecki et Yann Gambin, « SARS-CoV-2 proteases PLpro and 3CLpro cleave IRF3 and critical modulators of inflammatory pathways (NLRP12 and TAB1): implications for disease presentation across species », Emerging Microbes & Infections, vol. 10, no 1, , p. 178-195 (PMID 33372854, PMCID 7850364, DOI 10.1080/22221751.2020.1870414, lire en ligne
 )
) - Maxim Ryadnov, Frenc Hudecz, Amino Acids, Peptides and Proteins, The Royal Society of Chemistry, 2018, volume 42, p. 238, (ISBN 978-1-78801-002-3), aperçu consultable en ligne
-
(en) Marina Macchiagodena, Marco Pagliai et Piero Procacci, « Characterization of the non-covalent interaction between the PF-07321332 inhibitor and the SARS-CoV-2 main protease », Journal of Molecular Graphics and Modelling, vol. 110, , article no 108042 (PMID 34653812, DOI 10.1016/j.jmgm.2021.108042, lire en ligne
 )
) - Pfizer’s Novel COVID-19 Oral Antiviral Treatment Candidate Reduced Risk Of Hospitalization Or Death By 89% In Interim Analysis Of Phase 2/3 EPIC-HR Study, 05/11/2021, communiqué de Prizer, consultable en ligne sur BusinessWire.com
- FDA Authorizes First Oral Antiviral for Treatment of COVID-19, annonce du 22/12/2021 sur le site officiel de la FDA
- Portail de la biologie cellulaire et moléculaire
- Portail de la maladie à coronavirus 2019