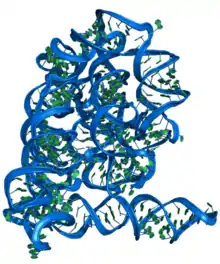
حمض نووي ريبوزي
الحمض النووي الريبوزي* ويُسمى اختصارًا رنا RNA1، هو جزيء حيوي يتواجد تقريبًا لدى كل الكائنات الحية والفيروسات، كما يلعب أدوارًا متعددة في نقل وتشفير وفك تشفير وتنظيم التعبير عن المعلومات الوراثية وتحفيز العديد من التفاعلات الكيميائية. الرنا والدنا (DNA)2 من أهم الأحماض النووية التي تُشكِّل إلى جانب كل من البروتينات والليبيدات والسكريات المتعددة الجزيئات الضخمة الأربعة الضرورية للحياة.
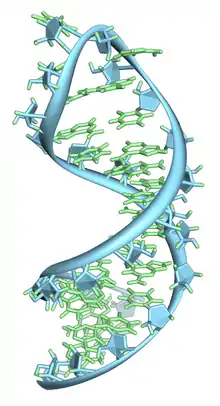
بنية الرنا مشابهة لبنية الدنا فكلاهما يتكون من تسلسل نوكليوتيداتٍ متعدد، لكنهما يختلفان في أن الرنا يتواجد في معظم الحالات على شكل سلسلة مفردة قصيرة يتراوح طولها بين عدة نوكليوتيدات إلى آلاف النوكليوتيدات بينما يكون الدنا دائمًا على هيئة لولب مزدوج يبلغ طوله من عدة ملايين إلى عدة مليارات من الأزواج القاعدية. فضلا عن ذلك، سكر العمود الفقري في الرنا هو الريبوز بدل الريبوز منقوص الأكسجين في الدنا والقاعدة المكملة للأدينين هي اليوراسيل بدل الثايمين.[2]
الرنا أكثر غنًى بالقواعد المعدلة من الدنا وذلك لقيامه بالعديد من الوظائف الخلوية، ويوجد فيه ما يزيد عن 100 نوكليوسيد معدل [3] يتواجد أكثرها في الرنا الناقل [4] ومن أكثر هذه التعديلات شيوعا السودويوريدين والنوكليوسيدات الممثيلة في أكسجين الذرة 2' المتواجدة في الرنا الريبوسومي.[5] لا توجد بنية مستقرة دائمة يتخذها تسلسل رنا معين، بل يمكن لنفس التسلسل اتخاذ العديد من البنى حسب نوع الربائط التي يترابط معها والظروف الفيسيولوجية الكيميائية الخلوية، ويمكنه مثل البروتين اتخاذ بنية ثانوية وثالثية ورابعية للقيام بوظائفه التحفيزية،[6] تتطلب هذه البُنى ترابط قواعدٍ غير معياريٍ لا يتواجد في الدنا مثل الترابط المقصوص وتآثر ريبوز-قاعدة، حيث لوحظ لدى الرنا ما يزيد عن 150 نوعا من الترابط.[7]
يُخلَّق الرنا من الدنا عبر عملية النسخ بواسطة إنزيمات بوليميراز الرنا وينتج رنا رسول أولي يخضع لتعديلات حتى يصبح رنا ناضجا جاهزا لأداء وظيفته. حوالي 97% من الرنا المخلَّق لا يشفِّر بروتينا [8][9][10][11] ومعظمه رنا ريبوسومي، أنواع الرنا كثيرة ووظائفها متنوعة منها: الرنا الرسول الذي ينقل المعلومة الوراثية من الدنا إلى الريبوسوم لتخليق البروتين عبر عملية الترجمة،[12] الرنا الناقل الذي ينقل الأحماض الأمينية المطلوبة لتخليق البروتين،[13] الرنا الريبوسومي الذي يدخل في تركيب الريبوسوم والريبوزيمات المحفزة الأخرى، الرنا الميكروي الذي ينظم عملية التعبير الجيني والرنا الصغير المتدخل الذي يقوم بعملية تداخل الرنا.[14]
جينومات العديد من الفيروسات والفيرويدات تحتوي على الرنا فقط كحامل للمعلومة الوراثية وتتضاعف إما بمساعدة إنزيمات الخلية المضيفة أو بتحويل الرنا الخاص بها إلى دنا ودمجه مع دنا الخلية المضيفة.[15][16] توجد دراسات لاستخدام جزيئات الرنا في العلاج؛ وبشكل خاص الرنا الميكروي والرنا الصغير المتدخل عبر استهداف الجينات الفيروسية لإسكاتها ومحاربة العدوى والأمراض، أو استهداف الجينات الورمية للقضاء على السرطان.[17]
التسمية
حَمْض نَّوَوِي رِّيبوزي^[18][19][20] (بالإنجليزية: Ribonucleic acid) ويُسمى أيضًا:
- حمض الريبونوكلئيك[19][20][21] أو حمض الريبونوكلييك[19][20][22]
- حمض نووي ريبي[19][20][21] أو حمض ريبي نووي[19][20][22]
كما يُعرف الحمض النووي الريبوزي (RNA) اختصارًا رنا[23][24][25][26] أو ر ن أ[23][25][26] أو آر أن أي[23]
مقارنة مع الدنا
البنية الكيميائية للرنا مماثلةٌ جدا لنظيرتها الخاصة بالدنا، لكن تختلف عنها في ثلاثة أمور أساسية:
- على عكس الدنا مزدوج السلاسل، الرنا جزيء ذو سلسلة مفردة[27] في معظم وظائفه البيولوجية ويتكون من سلسلة أقصر بكثير من النوكليوتيدات.[28] إلا أنه يمكن للرنا -بواسطة الازدواج القاعدي المكمل- تكوين لوالب مزدوجة بسلسلته المفردة كما هو الحال في الرنا الناقل.
- يحتوي العمود الفقري للدنا المتكون من سكر-فوسفات على الريبوز منقوص الأكسجين، بينما يحتوي عمود الرنا الفقري على الريبوز.[2] للريبوز مجموعة هيدروكسيل OH- مرتبطة بحلقة البنتوز في الموقع 2'، في حين أن الريبوز منزوع الأكسجين لا يملك مجموعة هيدروكسيل في تلك الموقع. تجعل مجموعات الهيدروكسيل في ريبوز العمود الفقري الرنا أقل استقرارا من الدنا وذلك لأنه أكثر عرضة للحلمأة.
- القاعدة المكملة للأدينين في الدنا هي الثايمين، لكن في الرنا هي اليوراسيل وهي هيئة غير ممثيلة من الثايمين.[29]
مثل الدنا، تملك معظم أنواع الرنا النشطة بما في ذلك الرنا الرسول، الرنا الناقل، الرنا الريبوسومي، الرنا الصغير النووي، وغيرها من جزيئات الرنا غيرِ المشفِّرة تسلسلاتٍ ذاتيةً مكملةً تسمح لأجزاءٍ منها بالتطوي[30] والازدواج مع نفسها لتكوين لوالب مزدوجة. كشفت تحليلات لجزيئات الرنا هذه أنها منتظمة للغاية، وعلى عكس الدنا لا تتكون بُنى الرنا من لوالب مزدوجة طويلة بل مجموعة من اللوالب القصيرة المجمعة في بنى تشابه تلك الخاصة بالبروتينات، وعليه يمكن للرنا تنفيذ تحفيزات كيميائية (مثل الإنزيمات)،[31] فعلى سبيل المثال كَشَفَ تحديد بنية الريبوسوم -وهو مركب رنا-بروتين يقوم بتحفيز تكوين الروابط الببتيدية- أن الموقع النشط الخاص به مكون بالكامل من الرنا.[32]
البنية
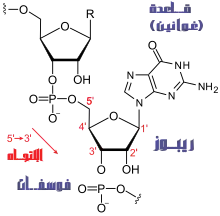
الرنا حمض نووي وذلك يعني أنه مكون من سلسلة من النوكليوتيدات، يتكون كل نوكليوتيد في الرنا من سكر ريبوز ذرات الكربون فيه مرقمة من 1' إلى 5'، بالإضافة إلى قاعدة نووية (نيتروجينية) ومجموعة فوسفات. ترتبط القواعد النووية بالريبوز عبر رابطة غليكوسيدية بين ذرة الكربون 1' وذرة نيتروجين وهذه القواعد هي: أدينين [A]، ثايمين [T]، غوانين [G]، يوراسيل [U]، حيث أن الأدينين والغوانين هما من البيورينات، في حين أن السايتوسين واليوراسيل هما من البيريميدينات. ترتبط مجموعة الفوسفور بالموقع 3' في الريبوز الأول وبالموقع 5' في الريبوز الثاني عبر روابط فوسفات ثنائية الإستر، لدى مجموعات الفوسفات شحنات سالبة وهذا يجعل الرنا جزيئا مشحونا (عديد الأنيون). تشكل القواعد روابط هيدروجينية بين السايتوسين والغوانين وبين الأدينين واليوراسيل.[33] استُبدِل الثايمين الخاص بالدنا باليوراسيل في الرنا،[34] والفرق بين هاتين القاعدتين هو استبدال مجموعة الميثيل في الموقع الخامسة للثايمين بذرة هيدروجين في اليوراسيل، هذا التعديل في البنية لا يؤثر على خصائص الارتباط مع الأدنين.[35][36] توجد تآثرت أخرى بين القواعد مثل ترابط مجموعة من قواعد الأدينين مع بعضها في انتباج،[37] أو الحلقة الرباعية GNRA التي يوجد فيها الزوج القاعدي غوانين-أدينين.[33]
الكيمياء الفراغية للريبوز
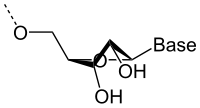
من الناحية التصاوغية، يؤثر تواجد ذرة أكسجين في الموقع 2' في الريبوز على تصاوغ حلقة الفورانوز الخاصة بالريبوز. هذه الحلقة المغايرة المكونة من خمس ذرات ليست ذات مستوٍ واحد، ويوجد لها مصاوغات فراغية عديدة منها مصاوغين أساسيين هما: C'2-إندو وC'3-إندو. المصاوغ C'3-إندو تكون فيه الذرة رقم 3 في نفس الجانب الذي تتواجد فيه القاعدة النيتروجينية والذرة 5' لسكر الريبوز وتكون المسافة فيه بين مجموعتي الفوسفات هي الأقصر 5.9 أنغستروم (شكل1)، أما المصاوغ C'2-إندو فتكون فيه ذرة الكربون 2 في نفس جانب القاعدة النيتروجينية والذرة 5' لسكر الريبوز ومسافة مجموعتي الفوسفات فيها هي 7 أنغستروم (شكل2).[38][39] في الرنا الذي يحتوي على ذرة أكسجين بالموقع 2' يكون المصاوغ C'3-إندو مفضلا[40] ما يعني أن لوالب الرنا المزدوجة تكون من النوع أ وهي مختلفة عن لوالب الدنا المزدوجة الشائعة والتي تكون أغلبها من النوع ب الذي يكون فيها الريبوز منقوص الأكسجين على هيئة المصاوغ C'2-إندو.[41]
النوكليوتيدات المعدلة
يُنسخ الرنا بأربع قواعد فقط (أدينين، سايتوسين، غوانين واليوراسيل)[42] لكن يمكن لهذه القواعد والريبوزات المرتبطة بها أن تُعدَّل بطرق مختلفة مع نضوج الرنا، فمثلا في نوكليوتيد السودويوريدين (Ψ) يتغير الربط بين اليوراسيل والريبوز من رابطة C–N إلى رابطة C–C، تتواجد القاعدة المعدلة 5-ميثيل يوريدين (T) في أماكن عديدة (أكثرها شهرة تواجدها في الحلقة TΨC الخاصة بالرنا الناقل).[43] من القواعد المعدلة الشهيرة كذلك الهيبوزانتين، وهي أدنين منزوع الأمين يسمى نوكليوسيدها إينوسين ويلعب الإينوسين دورا مفتاحيا في نظرية ووبل الخاصة بالشيفرة الجينية.[44]
يوجد أزيد من 100 نوكليوسيد معدل يظهر طبيعيا.[3] وأكثر أنواع التعديلات البنيوية يمكن أن تتواجد في الرنا الناقل، [4] في حين أن السودويوريدين والنوكليوسيدات الممثيلة في أكسجين الذرة 2' المتواجدة في الرنا الريبوسومي هي الأكثر شيوعا.[5] وظائف العديد من هذه التعديلات في الرنا مازالت غير مفهومة كليا، إلا أنه يتجلى في الرنا الريبوسومي أن العديد من تعديلات ما بعد النسخ تحدث في مناطق عالية الوظيفية مثل مركز بيبتيديل ترانسفيراز وواجهة الوحدة الفرعية، ما يشير إلى أنها ضرورية للنشاط العادي.[45]
الرنا مزدوج السلاسل ولولب الرنا المزدوج
الرنا مزدوج السلاسل (dsRNA) هو رنا له سلسلتين متكاملتين، وهو مشابه للدنا الموجود في الخلايا لكن مع استبدال الثايمين باليوراسيل. يشكل الرنا مزدوج السلاسل المادة الجينية لبعض الفيروسات (فيروسات الرنا مزدوجة السلاسل). ويمكن لأنواع منه مثل الرنا الفيروسي أو الرنا الصغير المتداخل (siRNA) إثارة تداخل الرنا لدى حقيقيات النوى وكذلك استجابة إنترفيرون لدى الفقاريات.[48][49][50][51]
اللولب المزدوج من النوع أ الذي يتخذه الرنا له خصائص هندسية مختلفة عن النوع ب الشائع في الدنا، فعدد أزواج القواعد لكل دورة هو 11 بدل عشرة لدى النوع ب. مستوى الزوج القاعدي ليس عموديا على محور اللولب المزدوج بل يشكل زاوية 75° درجة معه.[52][53] ونتيجة لذلك ينزاح محور الولب ويصبح غير مارٍ عبر مركز الزوج القاعدي بل داخل الثلم الكبير، وهذا يزيد قطر اللولب من 20 أنغستروم لدى النوع ب إلى 26 أنغستروم في النوع أ.[54] هندسة الثلمين تتغير كذلك بشكل كبير: الثلم الصغير يصبح قابلا للوصول بينما يصبح الثلم الكبير أكثر عمقا وحدة. وهذا له تأثير على كيفية تآثر لولب الرنا المزدوج مع البروتينات لأن حدة الثلم الكبير تصبح حاجزا أمام وصول الربائط البروتينية.[55]
البنية لدى الكائنات الحية
تتواجد معظم جزيئات الرنا الطبيعية على شكل سلسلة مفردة في الخلية، وذلك عكس الدنا الذي يشكل لولبا مزدوج السلاسل.[34] تتطوى سلسلة الرنا على نفسها مشكلة بنية مستقرة ومتماسكة جدا، أساس هذه البنية هو تشكل الترابط القاعدي بين القواعد المكملة. تكامل الروابط الداخلية بين قواعد الرنا يؤدي إلى تشكيل بنية ثانوية، يمكن أن تتآثر هذه البنية بتآثرات بعيدة مكملة وتشكِّل بنية ثلاثية الأبعاد أو بنية ثالثية. يعتمد تشكُّلُ بنى الرنا على شروط فيسيولوجية ومحيطية أهمها تواجد الكاتيونات المكافئة مثل أيون المغنزيوم +Mg2. تتفاعل هذه الكاتيونات مع مجموعات الفوسفات وتعمل على استقرار البنية، بعمل واجهة ضد قوة التنافر الهكروستاتية بين شحنات الفسفور السالبة.[6] البنية الثالثية للرنا هي أساس تنوع وظائفه وبشكل خاص قدرته على تحفيز التفاعلات الكيميائية (ريبوزيم).
بنية ثانوية

البنية الثانوية للرنا هي مجموع الروابط البينية داخل جزيء مفرد السلسلة.[56] يُحدِث مجموع الروابط بنيةً هيكلةً خاصةً تتكون من مناطق لولبية (جذوع مترابطة قاعديا) ومناطق غير مترابطة (حلقات). وعليه البنية الثانوية هي الهيكلة التي تحدثها الروابط الداخلية للجزيء.
يَحدُث تشكل البُنى الثانوية في جزيء رنا بتواجد مناطق تحتوي تسلسلات متناظرة يمكنها الترابط لتشكيل بنية لولبية مزدوجة محلية. على سبيل المثال إذا احتوى الرنا على التسلسلين --GUGCCACG------CGUGGCAC-- فإن بإمكانهما تشكيل تسلسل متناظر تكون فيه نوكليوتيدات التسلسل الثاني مكملة للأول، فبعد عكس اتجاه أحدهما يمكن أن يترابط التسلسلان عبر توازٍ متضاد لتشكيل منطقة محلية مزدوجة تسمى جذع وبين هذين التسلسلين تتكون «حلقة» وهذا ما يسمى بالحلقة الجذعية أو بنية دبوس الشعر.
في جزيئات الرنا الطويلة جدا يمكن أن تتواجد بنُى أكثر تعقيدا تنتج عن ترابط عدة مواقع متكاملة أو متناظرة. حسب طريقة تجمع هذه المناطق يمكن أن تنتج بُنى مختلفة ذات جذوع متعددة وأنواع مختلقة من الحلقات تشمل:[57]
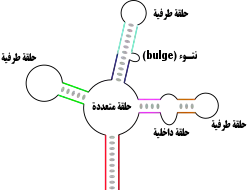
- الحلقات الطرفية أو النهائية، وتقع في نهاية الجذع.
- الحلقات الداخلية والتي تحدث وسط الجذع.
- الحلقات المتعددة والتي تحتوي على ثلاث جذوع أو أكثر وتشكل نقاط تفرع في البنية.
- النتوء (bulge) أو الحلقة الجانبية والتي تتشكل على إحدى سلسلتي الجذع فقط. واستمرارية الجذع (اللولب المزدوج) لا تتأثر بشكل عام وتبقى القواعد محزَّمةً بشكل محوري على كلا جانبي النتوء.
لا توجد بنية مستقرة دائمة يتخذها تسلسل رنا معين، بل يمكن لنفس التسلسل اتخاذ العديد من البنى حسب نوع الربيطة التي يترابط معها (بروتين، جزيء صغير...) أو الظروف الفيسيولوجية الكيميائية (قوة أيونية، أس هيدروجيني). يمكن بشكل عام تتبع تشكل أو اندماج بنية الرنا الثانوية بواسطة قياساتٍ مطيافيةٍ فمثلا: امتصاص قواعد الرنا للأشعة فوق البنفسجية يكون أكثر في حالة عدم التطوي وأقل عند التطوي (ظاهرة فرط الكرومية).[58]
بنية ثالثية

ترابطات غير معيارية
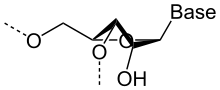
بالإضافة إلى هيكلة الحلقات واللوالب الخاصة بالبنية الثانوية والتي تنشأ من ترابطِ قواعدٍ معياريٍّ، يمكن للرنا اتخاذ بنية ثلاثية الأبعاد أو بنية ثالثية مثل البروتين. داخل هذه البنية تتكامل اللوالب بواسطة ترابط قاعدي غير معياري، أي أنه مختلف عن الترابط القاعدي الكلاسيكي من نوع واتسون-كريك (A=U و G≡C) وووبل (G=U). توجد في البنى الثالثية للرنا أنواع عديدة من الترابطات تم تحديدها بواسطة علم البلورات السيني أو الرنين المغناطيسي النووي منها على سبيل المثال: ترابط هوغستين،[59] والترابط المقصوص (الصورة).[60] كما توجد كذلك تآثرات ريبوز-قاعدة وخاصة مع مجموعة الهيدروكسيل 2' التي يمكنها تكوين روابط هيدروجينية. لوحظ ما يزيد عن 150 نوعا من الترابطات وتم تصنيفهم وتجميعهم في عدة عائلات تصنيفية، واقتُرح نظام تسمية لجميع هذه التآثرات بواسطة إريك ويستهوف وزملائه.[7] هذه الترابطات غير المعيارية هي دائمًا روابط هيدروجينية بين القواعد -المشتركة في المستوي- كما هو الحال في ارتباط واتسون-كريك.
تآثرات بعيدة المسافة
يمكن أن تظهر تآثرات معيارية أو غير معيارية بين مناطق بعيدة في البنية الثانوية، تكون في الغالب متواجدة في الحلقات وهذا يسمح باستقرار البنية والتطوي، ومن هذه التآثرات بعيدة المدى:
- العقدة الكاذبة: وهي بنية تتشكل بتآثر حلقة مع منطقة تتواجد خارج الجذع الذي يحددها.[61]
- اللولب الثلاثي: والذي يتشكل عندما تندرج سلسلة مفردة في الثلم الكبير لمنطقة لولب مزدوج.
- تآثرات مستقبِل الحلقة الرباعية: وهي تآثرات بين حلقات فائقة الاستقرار رباعية النوكليوتيدات ولوالب مزدوجة أو شبه مزدوجة.[62]
التخليق
يُحفَّز تخليق الرنا عادة بواسطة إنزيم بوليميراز الرنا باستخدام الدنا كقالب، وهي عملية تعرف بالنسخ. الشروع في النسخ يبدأ بارتباط الإنزيم بتسلسل محفز في الدنا (يتواجد عادة «عكس التيار» في الجين المستهدف). يتم فك لولبة اللولب المزدوج بواسطة إنزيم الهيليكاز، ثم يمضي البوليميراز على طول السلسلة القالب في الاتجاه 3' إلى 5' مخلقًا جزيء رنا مكمل تكون فيه الاستطالة في الاتجاه 5' إلى 3'. يحدد تسلسل الدنا كذلك مكان الانتهاء من الترجمة.[63] تخضع النسخة الأولية للرنا لتعديلات ما بعد النسخ بواسطة إنزيمات، على سبيل المثال: يضاف ذيل عديد الأدينيلات وقبعة 5' إلى الرنا قبل الرسول الخاص بحقيقيات النوى وتُزال الإنترونات بواسطة جسيم التضفير.
يوجد كذلك العديد من بوليميرازات الرنا المعتمدة على الرنا التي تستخدم الرنا كقالب لها لتخليق سلاسل جديدة من الرنا، فمثلا: عدد من فيروسات الرنا (مثل الفيروسة السنجابية) تستخدم هذا النوع من الإنزيم لمضاعفة معلوماتها الجينية.[64] كذلك بوليميراز الرنا المعتمد على الرنا جزء من مسار تداخل الرنا في العديد من الكائنات.[65]
البدء والاستطالة

البدء في النسخ بواسطة بوليميراز الرنا يتم على مستوى تسلسل محدد على الدنا يسمى محفز، يشمل هذا المحفز عنصرا أو عدة عناصر ذات تسلسل محفوظ.[67] والتي ترتبط بها بروتينات محددة تسمى عوامل النسخ. يكون العنصر المجاور لموقع بداية النسخ في العادة غني بنوكليوتيدات T وA ولهذا يطلق عليه صندوق TATA[68] عند حقيقيات النوى أو صندوق بربنو لدى البكتيريا.[69] تقوم عوامل النسخ بتوظيف بوليميراز الرنا للقيام بالنسخ في المحفز وفك لولبة اللولب المزدوج، فينشأ ما يسمى فقاعة النسخ حين يُفتح الدنا ويتم الشروع في نسخ إحدى السلسلتين (السلسلة القالب).
بعد أن يرتبط بوليميراز الرنا بالمحفز وتتشكل فقاعة النسخ، يقوم البوليميراز بنسخ النوكليوتيدات الأولى بطريقة ساكنة دون أن يغادر تسلسل المحفز. بعد ذلك تفك عوامل النسخ ارتباطها ويبدأ إنزيم البوليميراز بالتقدم.[70] ويتم النسخ في الاتجاه 5' إلى 3' باستخدام إحدى السلسلتين كقالب والريبونوكليوتيدات ثلاثية الفوسفات (ATP وGTP وCTP وUTP) كمواد أولية. في الكائنات الحية، تبلغ سرعة الاستطالة لدى الإشريكية القولونية حوالي 50 إلى 90 نوكليوتيد لكل ثانية.[71]
الإنهاء
آليات الإنهاء تختلف بشكل كامل بين البكتيريا وحقيقيات النوى، فلدى البكتيريا الآلية الرئيسية للإنهاء تكون بتدخل تسلسل خاص من الرنا يسمى المُنهي وهو مركب من ثلاث حلقات جذعية مستقرة متبوعة بسلسلة من وحدات اليوريدين (U). عندما يخلِّق بوليميراز الرنا هذا التسلسل يُحدث تطوي حلقة الرنا توقف البوليميراز.[72] بعد ذلك يقوم الرنا الذي لم يعد متصلا بالسلسلة القالب سوى بعدة أزواج قاعدية A-U ضعيفة بالانفصال من دون تدخل العوامل البروتينية الأخرى، يمكن أن يحدث الإنهاء كذلك بتدخل عامل بروتيني محدد يسمى رهو (Rho).[73]
لدى حقيقيات النوى إنهاء النسخ بواسطة بوليميراز الرنا 2 يكون مقترنا مع التذييل بعديد الأدينيلات. يقوم المنهي بإرسال إشارات تتعرف عليها بروتينات مصاحبة للبوليميراز 2 والتي تبدأ عملية الإنهاء بتوظيف بروتيني CPSF وCStF اللذان يتعرفان على إشارة التذييل بعديد الإدينيلات 5′-AAUAAA-3′ وإشارة قص الرنا، فيقومان بقص الرنا وعند انفصال البوليميراز 2 يقومان بتوظيف بوليميراز عديد الأدينيلات لإضافة ذيل عديد الإدينيلات.[74]
النضوج
- التقبيع: القبعة هي نوكليوتيد معدل يُضاف إلى النهاية 5' الخاصة بالرنا الرسول لدى حقيقيات النوى، ويتكون من جزيء غوانوزين ممثيل مرتبط برابطة 5'-5' ثلاثية الفوسفات مع أول نوكليوتيد يترجمه بوليميراز الرنا.[75] يتم هذا التعديل في النواة بواسطة عدة إنزيمات منها 5'-فوسفاتاز عديد النوكليوتيد، ناقلة غوانيليل الرنا الرسول وناقلة الميثيل. للقبعة أدوار عديدة فهي تساهم في استقرار الرنا الرسول وحمايته من التحلل بواسطة النوكليازات الخارجية 5'-3' وتسمح كذلك بتوظيف عوامل البدء الضرورية لتثبيت الريبوسوم على الرنا الرسول وبدء الترجمة.
- التذييل بعديد الأدينيلات: هو إضافة مجموعة من النوكليوتيدات للنهاية 3' الخاص بالرنا مكونة حصريا من نوكليوتيدات الأدينين (A)، ولهذا تسمى ذيل عديد الأدينين. يُضاف الذيل بواسطة إنزيم يسمى بوليميراز عديد الأدينين أو ناقلة أدينيليل عديد النوكليوتيد،[76] ولا يتم نسخه من الدنا لأنه غير مشفر به. يتواجد ذيل عديد الأدنين في العادة عند نهاية الرنا الرسول ويساهم بروتين PABP (بروتين مرتبط بعديد الأدينين) في عملية التذييل، لدى حقيقيات النوى تذييل الرنا الرسول ضروري لترجمته بواسطة الريبوسوم ويساهم في استقراره، أما لدى البكتيريا وبعض المتقدرات، التذييل بعديد الأدينين إشارة لتفكيك الرنا الرسول.[77]
- التضفير: هو تعديل يحدث بعد النسخ وتتم فيه إزالة الإنترونات ووصل (تضفير) الإكسونات لدى الرنا الرسول وبعض جزيئات الرنا الأخرى مثل الرنا الناقل.[34] تتواجد الإنترونات لدى حقيقيات النوى وهي قطع من الرنا مشفرة في الجينوم وتُنسَخ في الرنا الأولي، لكنها تُزال عند النضوج. في معظم الحالات يتم التضفير بواسطة مركب يسمى جسيم التضفير،[78] ويحدث داخل النواة لدى حقيقيات النوى قبل انتقال الرنا الناضج إلى السيتوبلازم.

دورة الرنا في الخلية
تخزين ومراقبة الرنا في حبيبات الرنا
الرنا الرسول الذي لا تتم ترجمته يتم تخزينه في العادة داخل حبيبات رنا، وتتواجد حبيبات الرنا هذه في الغالب بالسيتوبلازم وهي تكدسات كروية مميزة مكونة من الرنا والبروتينات. تُنظم حبيبات الرنا الترجمة وتحافظ على استقرار الرنا الرسول الموجود بداخلها، بالإضافة إلى الرنا الرسول يمكن أن تحتوي الحبيبات على الوحدات الفرعية الريبوسومية، عوامل الترجمة، الإنزيمات المسؤولة على تفكيك الرنا، مكونات تدخل الرنا، الهيليكازات، البروتينات البنيوية، وغيرها من البروتينات المرتبطة بالرنا،[79] ومن أهم أنواعها:
حبيبات الخلية المنتشة: والتي تظهر أثناء تطور هذه الخلايا وتبقى فيها بعد أن تصبح خلية بيضية. تحتوي هذه الحبيبات على جزيئات رنا رسول محددة تشفر البروتينات الضرورية لتطور الجنين، وتخزنها على هيئة غير نشطة حتى تحتاج إليها الخلية لاحقا. فضلا عن ذلك، تحتوي حبيبات الخلايا المنتشة على جزيئات الرنا المستعملة في تدخل الرنا وبشكل خاص الرنا المتآثر مع بيوي والذي يعطل الينقولات ويمنع تضرر الجينوم.[79][80]
حبيبات الإجهاد: تتشكل حين تتعرض الخلايا للإجهاد وتحتوي على جزيئات الرنا الرسول المشفرة للبروتينات الخلوية الشائعة. كما تحتوي هذه الحبيبات على ريبوسومات جاهزة لترجمة جزيئات الرنا الرسول، لكنها تكون مخزنة على هيئة غير نشطة طوال فترة الإجهاد لكى لا تتعرض للتضرر. تتآثر هذه الحبيبات في العادة مع أجسام P وتنقل إليها جزيئات الرنا لتقوم بتفكيكها.[81]
أجسام P (الأجسام المعالجة): تتواجد في الخلايا الجسدية وتساهم في تفكك الرنا الرسول وتتوسط عملية إسكات الرنا باستخدام جزيئات الرنا الميكروي. ونظرا لقدرتها على تفكيك الرنا، تعمل كذلك على مراقبة جودة الرنا الرسول الخلوي.[79]

الحبيبات العصبونية وتتواجد في العصبونات وتعمل على نقل الرنا الرسول إلى محواراتها. تحمل داخلها ريبوسوماتٍ وعوامل بدء الترجمة، لكن لا تتم الترجمة حتى تصل الحببيبة إلى المكان الصحيح. تلعب الحبيبات العصبونية دورا مهما في التجدد العصبي واضطرابات نقل الرنا الرسول بواسطتها تصاحبها اضطرابات عصبية.[82]
تحلل الرنا
تفكيك الرنا بواسطة الريبونوكلياز (رناز) هي عملية مراقبة وتنظيم تقوم بها الخلية لإزالة الرنا المتضرر أو الرنا الذي أدى وظيفته ولم تعد هنالك حاجة إليه، كما أنه آلية تستخدم في معالجة الرنا والدفاع ضد الفيروسات، فهو أساس أنظمة دفاعية أكثر تعقيدا مثل تدخل الرنا. بشكل عام، تتم عملية التفكيك إما من نهايتي جزيئة الرنا بواسطة النوكلياز الخارجي (يبدأ القص من النهاية 5' أو 3') أو بواسطة النوكلياز الداخلي أين يكون القص داخل السلسلة. توجد العديد من الريبونوكليازات ذات وظائف متداخلة، وكل رنا لا تتم معالجته ضد تأثيراتها يتم تفكيكه.
لدى البكتيريا تحدث عمليتي النسخ والترجمة في آن واحد وعمر النصف للرنا الرسول يتراوح بين ثانية وساعة. النهاية 5' للرنا محمية بمجموعة ثلاثية الفوسفات في النوكليوتيدة الأولى وإشارة البدء لعملية التفكيك هي قص جزيئتي فوسفات وإضافة ذيل عديد الأدينيلات.[83] لدى حقيقيات النوى عمر النصف للرنا الرسول أطول ويتراوح بين دقائق إلى عدة أيام، واستقراره ومنع تحلله يتم بإضافة القبعة للنهاية 5' وذيل عديد الأدينيلات للنهاية 3' والارتباط ببروتينات نووية ريبوزية لتجنب وصول الريبونوكليازات إليه.
أكبر مركبات التفكيك هما الإكسوسوم لدى حقيقيات النوى وجسيم التفكيك لدى البكتيريا.[84] ويحتوي هذان المركبان في العادة على نوكليازات داخلية وخارجية وفوسفوريلاز عديد النوكليوتيد الذي يقص قطع الرنا الصغيرة التي تنتجها النوكليازات الداخلية ويحولها إلى نوكليوتيدات. يساعد الهيليكاز في تحلل الرنا بفك تطوي البنية الثانوية للرنا. فضلا عن ذلك يحتوى جيسم التفكيك البكتيري على بوليميراز عديد الأدينيلات ويحتوي الإكسوسوم على بروتينات مسؤولة على التعرف وإزالة استقرار مناطق في الرنا، مثل العنصر الغني بـAU
الوظيفة

للرنا عدة وظائف خلوية أساسية منها:
- نقل المعلومة الوراثية: تستخدم الخلية الرنا الرسول لنقل المعلومة الوراثية المتعلقة بجين خارج النواة، ثم ترجمتها لتخليق بروتينات.
- تحفيز التفاعلات كالإنزيمات: يمكن للرنا -مثل البروتينات- التطوي لبنية ثلاثية الأبعاد لتشكيل بنية مركبة، تسمح هذه البنى لبعض جزيئات الرنا المحددة بالتصرف كأنزيمات ومن الأمثلة على ذلك: الريبوزيم، الريبوسوم، الريبونوكلياز P وبعض الإنترونات هي ريبوزيمات كذلك. توجد حجج تشير بصفة غير مباشرة أن جسيم توصيل وقص الرنا الرسول (جسيم التضفير) هو ريبوزيم كذلك[85] رغم أن الإثبات الرسمي لم يتم تقريره بعد.
- توجيه الإنزيمات: تُستخدم بعض جزيئات الرنا كعوامل مرافقة للبروتينات لتمكِّنها من تحديد واستهداف تسلسلات معينة، ومن الأمثلة على ذلك الرنا الصغير النويي (snoRNA) الذي يوجه إنزيمات تعديل الرنا الريبوسومي، الرنا التيلوميرازي وهو عامل مرافق لإنزيم التيلوميراز الذي ينشئ نهايات الصبغيات.
- تنظيم التعبير الجيني: تلعب بعض جزيئات الرنا غير المشفرة دورا في تثبيط التعبير عن بعض الجينات أو مجموعة من الجينات كما هو الحال لدى الرنا المضاد للاتجاه الذي يترابط مع رنا مستهدف يمنع ترجمته إلى بروتين.
- النقل: يقوم الرنا الناقل أثناء عملية الترجمة بنقل الأحماض الأمينية للارتباط بالبروتين الذي في طور التخليق.
الأنواع
يقوم الرنا الرسول بنقل المعلومة من الدنا إلى الريبوسوم وهو موقع تخليق البروتين (الترجمة) في الخلية، ويحدد تشفير تسلسله تسلسل الأحماض الأمينية في البروتين الناتج.[86] مع ذلك العديد من جزيئات الرنا لا تشفر البروتين (حوالي 97% من جزيئات الرنا الناتجة عن النسخ غير مشفرة للبروتين لدى حقيقيات النوى.[8][9][10][11]). يمكن تخليق جزيئات الرنا غير المشفرة مباشرة من جيناتها في الدنا، ويمكن كذلك أن تنتج من إنترونات الرنا الرسول.[87] من أشهر الأمثلة على جزيئات الرنا غير المشفرة: الرنا الناقل والرنا الريبوسومي وكلاهما له دور في عملية الترجمة.[29] يقوم الرنا غير المشفر بتنظيم التعبير الجيني ومعالجة الرنا وأدوار أخرى، بعض جزيئات الرنا قادرة على تحفيز تفاعلات كيميائية مثل القص والارتباط بجزيئات رنا أخرى،[88] وتحفيز تكوين الرابطة الببتيدية في الريبوسوم.[32]
الرنا الرسول
لا تُستخدم المعلومة الوراثية الموجودة في الدنا من طرف الخلية مباشرة لتخليق البروتينات، بل تُستخدم نسخٌ مؤقتة منها تسمى الرنا الرسول.[12] يحمل كل رنا رسول سيسترونا واحدا أو عدة سيسترونات أي نسخة من جين واحد أو عدة جينات (رنا رسول عديد السيسترونات).[89] لا يحتوى الرنا الرسول سوى على معلومة إحدى سلسلتي الدنا، أي السلسلة القالب ولا يحتوي على معلومة السلسلة المكملة. يختلف تسلسل الجين المتواجد في الدنا عن نظيره في الرنا الرسول الناضج الذي تعرض إلى تعديلات أهمها التضفير الذي يزيل المناطق غير المشفرة. يُخلَّقُ الرنا الرسول في نواة الخلية ثم ينتقل إلى السيتوبلازم ليُترجم بواسطة الريبوسوم إلى بروتين،[34] وعلى عكس الدنا الذي هو جزيء معمر يتواجد طوال فترة عمر الخلية، الرنا الرسول له فترة محدودة تتراوح بين عدة دقائق إلى عدة ساعة يتم بعدها تحليله وإعادة تدويره.
يتكون الرنا الرسول الناضج من ثلاث مناطق مميزة: منطقة 5' غير مترجمة (5′-UTR) تقع عكس التيار في السيسترون الذي يحملها، منطقة مشفرة، ومنطقة 3' غير مترجمة (3′-UTR).[34] تحتوى المنطقة 5' غير المترجمة على إشارات الترجمة التي تسمح بتوظيف الريبوسوم على السيسترون، يساهم الرنا الناقل في عملية الترجمة بنقل وإحضار الأحماض الأمينية الضرورية للتخليق الحيوي للبروتين. داخل الريبوسوم تقوم جزيئات الرنا الناقل باستخدام ضد الكودون الخاص بها للارتباط بثلاث قواعد -أو كودونات- من تسلسل الرنا الرسول، وحين يكون الارتباط كودون-ضد الكودون صحيحا يضيف الريبوسوم الحمض الأميني المحمول بواسطة الرنا الناقل إلى سلسلة البروتين التي في طور التخليق. العلاقة بين الكودونات والأحماض الأمينية تشكل الشيفرة الجينية.[90]

وظيفة الرنا الرسول متعددة، فهو يسمح بالحفاظ على مادة الدنا الأساسية من الاستخدام المباشر في الترجمة. يمنح تواجد الرنا الرسول للخلية آلية أساسية في تنظيم دورة إنتاج البروتين الذي يتغير الاحتياج الخلوي إليه حسب الوسط، نوع الخلية ومرحلة النمو، تنظيم نسخ الدنا إلى الرنا يتم بواسطة عوامل نسخ محددة تعمل على محفزات الجينات المستهدفة، فحين تبلغ كمية البروتين مستوى كافيا تُثبط عملية نسخ الرنا الرسول ويتم تحليله وتتوقف عملية إنتاج البروتين.

- بالأصفر: موقع ارتباط الحمض الأميني.
- بالبنفسجي: الجذع المستقبل. - بالأخضر: حلقة TΨC.
- بالأحمر: التفرع D. - بالبرتقالي: الحلقة المتغيرة.
- بالأزرق: حلقة الكودون المضاد. - بالرمادي: الكودون المضاد.
الرنا الناقل
الرنا الناقل هو أحد أنواع الرنا القصير ويتراوح طوله بين 70 و100 نوكليوتيد ويقوم بنقل الأحماض الأمينية إلى الريبوسوم أثناء الترجمة.[13] بنيته ثلاثية الأبعاد على شكل ورقة النفل وتتكون من أربع حلقات جذعية، تحتوي إحدى هذه الحلقات على الكودون المضاد وهو ثلاث نوكليوتيدات ترتبط بكودونٍ أثناء ترجمة الرنا الرسول في الريبوسوم.[92] الطرف المقابل لحلقة ضد الكودون يحمل الحمض الأميني الموافق للكودون ويكون مرتبطا بالرنا عبر رابطة إستر في النهاية 3′-OH. يُحفَّز تكوين رابطة الإستر هذه بواسطة إنزيمات خاصة تسمى مخلقات أمينوسيل الرنا الناقل. بمنظور ثلاثي الأبعاد، تتطوى بنية ورقة النفل الخاصة بالرنا الناقل على شكل حرف L يكون ضد الكودون في أحد الأطراف والحمض الأميني المؤستر في الطرف الآخر.[93]
جميع الخلايا الحية تمتلك مجموعة من جزيئات الرنا الناقل المختلفة التي تحمل عدة أحماض أمينية وقادرة على قراءة عدة كودونات. تعتبر جزيئات الرنا «موائمات» بين التسلسل الجيني والبروتيني، وكان فرنسيس كريك هو من اقترح وجود مثل هذه الموائمات قبل اكتشافها سنة 1958.[94]
الرنا الرسول الناقل
الرنا الرسول الناقل (tmRNA) يتواجد لدى العديد من البكتيريا والبلاستيدات، ويقوم بوسم البروتينات المشفرة بواسطة جزيئات الرنا الرسول التي لاتملك كودون توقف ليتم تفكيكها ومنع الريبوسوم من التوقف عن العمل.[95]
الرنا الريبوسومي
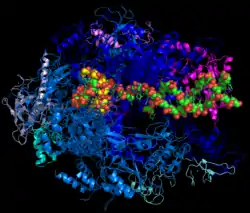
الرنا الريبوسومي هو المكون الذي يقوم بالتحفيز في الريبوسومات. تحتوي ريبوسومات حقيقيات النوى على أربع جزيئات رنا ريبوسومي مختلفة وهي: 18S و 5.8S و28S و5S، ثلاثة منها تُخلََّق في النواة والرابعة تخلق في مكان آخر. يرتبط الرنا الريبوسومي بالبروتين في السيتوبلازم لتشكيل بروتين نووي يسمى الريبوسوم، يرتبط الريبوسوم بالرنا الرسول ويقوم بعملية تخليق البروتين. يمكن للعديد من الريبوسومات الارتباط برنا واحد في أي وقت.[86] الرنا الريبوسومي سائد في معظم أنواع الخلايا ويشكل حوالي 80% من الرنا الخلوي.
الرنا المحفز أو الريبوزيم
اكتُشِف الرنا الذي يملك قدرات تحفيزية في العقد 1980 بواسطة فريق توماس تشيك الذي عمل على إنترونات جين الرنا الريبوسومي لكائن أولي مهدب يسمى رباعية الغشاء،[96] وبواسطة فريق سيدني ألتمان الذي درس الريبونوكلياز P وهو الإنزيم المسؤول عن نضوج الرنا الرسول.[97] وحصل تشيك وسيدني على جائزة نوبل في الكيمياء سنة 1989 تقديرا لاكتشافهم هذا.
في هاذين الاكتشافين، كان الرنا وحده قادرا على تحفيز عملية قص أو أسترة معينة بغياب البروتينات. سُميت جزيئات الرنا المحفزة هذه ريبوزيمات لكونها إنزيمات تتكون من حمض نووي ريبوزي. إنترون رباعية الغشاء قادرٌ على التضفير الذاتي وكان ركيزة نفسه، في حين أن الريبونوكلياز P هو إنزيم يعمل على عدة ركائز.

بعد هذه اكتشافات الأولية تم اكتشاف العديد من الريبوزيمات الطبيعية:
- الرنا الخاص بالفيرويدات أو الفيروسات الصغيرة (فيروسويدات) القادرة على قص نفسها.[98]
- توجد اليوم حجج قوية مبنية على صور ثلاثية الأبعاد تؤكد أن الريبوسوم في حد ذاته عبارة عن ريبوزيم.[99] الموقعان النشطان بالريبوسوم: مركز فك التشفير في الوحدة الفرعية الصغيرة ومركز بيبتيديل ترانسفيراز الذي يشكل الروابط الببتيدية مكونان حصريا من الرنا الريبوسومي.
- جسيم التضفير الذي يحفز قص الإنترونات ووصل الإكسونات في الرنا الرسول لدى حقيقيات النوى من الممكن كذلك أن يكون ريبوزيما.[85]
- بعض الريبوسويتشات -وهي مناطق تنظيمية في الرنا الرسول- تملك نشاطا إنزيميا في القص عند تواجد ربيطة.[100]
- توجد كذلك ريبوزيمات مصنعة، والتي يتم تصنيعها في المختبر بواسطة تقنيات مثل سيليكس.[101] حيث من الممكن تصنيع رنا تحفيزي قادر على تحفيز كمية كبيرة ومتنوعة من التفاعلات الكيميائية والارتباط بربائط مختلفة، وفُسر هذا الأمر على أنه حجة لصالح فرضية عالم الرنا. في بعض الأحيان تسمى جزيئات الرنا المصنعة هذه أبتمرات لأنها «قادرة، apte» على تنفيذ المهام الموكلة لها.[101]
بشكل عام، التطوي الخاص لهذه الريبوزيمات إلى بنى محددة هو ما يسمح لها بالتعرف على ركائزها وتحفيز تفاعلاتها، كما هو الحال عند الإنزيمات البروتينية.
رنا منظم
أول منظِّمات التعبير الجيني المعروفة كانت بروتينات تسمى مثبطات ومنشطات، وهي منظمات ذات مواقع ارتباط قصيرة محددة داخل مناطق المعزز بالقرب من الجينات المراد تنظيمها.[102] وُجد حديثا أن جزيئات الرنا تنظم التعبير الجيني كذلك، حيث توجد العديد من العمليات المعتمدة على الرنا لدى حقيقيات النوى تنظم التعبير الجيني في مختلف المراحل، مثل تداخل الرنا الذي يثبط الجينات بعد عملية النسخ، الرنا الطويل غير المشفر الذي يكبت تخليق مناطق من الكروماتين والرنا المُعزِّز الذي يزيد مقدار التعبير الجيني.[103] فضلا عن هذه الآليات لدى حقيقيات النوى، وُجد أن كلا من البكتيريا والعتائق تستخدم جزيئات رنا معدلة على نطاق واسع. الرنا الصغير البكتيري ونظام كريسبر أمثلة على أنظمة الرنا المعدلة لدى بدائيات النوى.[104] حصل فاير وميلو على جائزة نوبل في الطب أو علم وظائف الأعضاء سنة 2006 لاكتشافهما الرنا الميكروي (miRNA) وهو جزيء رنا قصير يمكنه الترابط قاعديا مع جزيئات الرنا الرسول.[105]

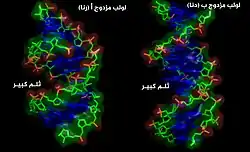
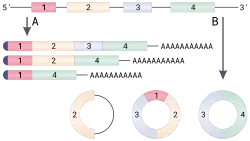
- تداخل الرنا بواسطة الرنا الميكروي: مستويات التعبير عن العديد من الجينات بعد النسخ يمكن التحكم فيها بواسطة تداخل الرنا والذي تترابط فيه جزيئات الرنا الميكروي قاعديا مع مناطق من الرنا الرسول وتستهدفه ليتم تحليله (الصورة).[14] هذه العملية المبنية على تضاد الاتجاه تتكون من مراحل أولها معالجة الرنا الميكروي لكي يتمكن من الترابط قاعديا مع مناطق من الرنا الرسول. وحين يحدث الترابط تقوم بروتينات أخرى بتوجيه الرنا الرسول ليتم تفكيكه بواسطة النوكلياز.
- الرنا النويي الصغير: (snoRNA) وهي جزيئات رنا قصيرة (60-300 نوكليوتيد) تتواجد داخل أنوية وجسيمات كاخال خلايا حقيقيات النوى وتقوم بتنظيم التعديلات على الرنا الريبوسومي، وبشكل خاص مثيلة أكسجين الذرة 2 بواسطة الرنا النويي C/D وتعديل اليوريدين بواسطة الرنا النويي H/ACA56. تسمح هذه الآلية بالتعديل المحدد لعدة مناطق في الرنا الريبوسومي بواسطة إنزيم واحد فقط باستخدام العديد من جزيئات الرنا النويي كموجهات. في الغالب يتم تشفير سلالسل الرنا النووي بواسطة تسلسلات الإنترونات.[106]
- الرنا الطويل غير المشفر: (lncRNA) هو رنا يزيد طوله عن 200 نوكليوتيدة ولا تتم ترجمته إلى بروتين.[107] دوره في البداية كان غامضا وكان يُعرف بأن Xist وجزيئات رنا طويلة غير مشفرة أخرى مسؤولة عن تعطيل الصبغي X، ثم أظهرت جيني لي وآخرون أنه يقوم بتثبيط كتل من الكروماتين عبر توظيف مركب عديد الأمشاط لكي لا يتم نسخ الرنا الرسول منها.[108] تساهم جزيئات رنا طويلة أخرى في تنظيم تعدد قدرات الخلية الجذعية وانقسام الخلية.[107] يسمح طول الرنا الكبير باتخاذ بنى ثلاثية الأبعاد معقدة تسمح له بالتآثر مع مختلف العوامل المرافقة مثل hnRNP-K وPRC2 (وهما مثبطان للنسخ) يتم توجيه هذه المركبات بفضل الرنا الطويل نحو تسلسلات التنظيم الخاصة بالجينات ليتم تثبيط التعبير عنها. علاقة الرنا الطويل مع الدنا تستلزم ترابطا قاعديا بينهما وهذا يعني تشكل لولب ثلاثي دنا-دنا-رنا.[109]
- الرنا المكون للتيلوميراز: (TERC) وحدة الرنا الفرعية الخاصة بالتيلوميراز تحتوي على رنا له علاقة بالنسخ العكسي الذي يُخلِّق القسيمات الطرفية في نهاية الصبغيات. يحتوي هذا الرنا على تسلسل يعمل كركيزة للتيلوميراز ليخلق دنا متكامل التسلسل.[110]
- رنا معزز: لم يتضح بعد إن كان الرنا المُعزِّز فئة مستقلة من الرنا ذات أطوال مختلفة أم فئة خاصة من الرنا الطويل. في كلا الحالتين فإن نسخه يتم من تسلسل المعزز وهي تسلسلات تنظيمية في الدنا بالقرب من الجينات التي تنظمها.[107][111] تقوم هذه الجزيئات بتنظيم نسخ الجين بزيادته عند الحاجة وذلك تحت رقابة المعزز الذي تم نسخها منه.[107][112]
الرنا الدائري
الرنا الدائري ويرمز له (circRNA) هو أحد أنواع الرنا منفردة السلاسل والتي تشكل -على عكس الرنا الخطي الشائع- حلقة تساهمية مغلقة تكون فيها النهايتان 3' و5' مترابطتان مع بعظهما. تمنح هذه الميزة العديد من الخصائص للرنا الدائري، والعديد من هذه الخصائص لم يتم اكتشافها إلا حديثا.
تنشأ جزيئات الرنا الدائرية من جينات مشفرة للبروتين، وقد صنفت على أنها رنا غير مشفر لكن ظهر حديثا أنها تشفِّر البروتينات.[113][114] بعض الرنا الدائري أظهر احتمالا لكونه رنا منظم، ومثل العديد من المصاوغات غير المشفِّرة، الوظيفة البيولوجية لمعظم جزيئات الرنا الدائري غير واضحة لحد الآن.
لأن الرنا الدائري لا يملك النهايتين 5' و3' فهو مقاوم للتفكك بواسطة النوكلياز الخارجي، ويفترض أنه أكثر استقرار من معظم الرنا الخطي المتواجد في الخلايا.[115]
رنا منظم لدى بدائيات النوى
اعتُقد في البداية أن الرنا المنظِم خاصٌ بحقيقيات النوى فقط، وكان ذلك جزءا من تفسير ملاحظةِ أن النسخ لدى الكائنات العليا كان أكثر من المتوقع، لكن بمجرد بدء البحث عن الرنا المنظِم في البكتيريا اكتشف أنه موجود لديها كذلك.[104] حاليا، الوفرة الطبيعية لآليات تنظيم التعبير الجيني بالرنا تُناقش على أنها حجة لصالح فرضية عالم الرنا.[103][116] يعمل الرنا الصغير البكتيري عبر الترابط عكس الاتجاه مع الرنا الرسول لتنظيم الترجمة بإنقاصها، سواء بالتأثير على استقرارية الرنا الرسول أو قابلية الإرتباط مقرون.[103] اكتشفت الريبوسويتشات كذلك لدى البكتيريا، وهي تسلسلات رنا تعمل على التنظيم تفارغيا، حيث تغير شكلها عند الارتباط بالمستقلبات لكي يكسبوا أو يفقدوا القدرة على التنظيم الجيني.[117][118] لدى العتائق أيضا آليات تنظيم بالرنا،[119] نظام كريسبر -الذي يُستخدم حديثا لتعديل الدنا في الموقع- يعمل بواسطة جزيئات رنا منظمة في العتائق والبكتيريا لتوفير حماية ضد الفيروسات الغازية.[103][120]
تدخل الرنا وتنظيم التعبير الجيني
بالإضافة إلى وظيفته في قراءة ونقل المعلومة الوراثية للتعبير عنها، يقوم الرنا كذلك بالتحكم في وتنظيم عملية التعبير هذه. لدى حقيقيات النوى أهم آلية لتنظيم التعبير الجيني هي تدخل الرنا والتي تحدث بعد عملية النسخ ويتم فيها قص جزيء رنا مزدوج السلاسل وإضافة إحدى سلسلتيه إلى مركب ريسك (RISC )، ثم إذا وجدت جزيئة رنا رسول مترابطة كليا مع الرنا الموجود في مركب ريسك فإنها تُقص بواسطة المركب ويتم تفكيكها، أما إن كان الترابط جزئيا فلا يتم قص الرنا الرسول لكن تُمنع عملية ترجمته.

يحدث تدخل الرنا بواسطة نوعين أساسيين من الرنا: الرنا المتدخل الصغير والرنا الميكروي. ينتج الرنا المتدخل الصغير من رنا مزدوج السلاسل ذي مصدر خارجي في الغالب (كالفيروسات مزدوجة السلاسل)، ولأن إحدى السلسلتين تضاف إلى المركب، فإن السلسلة الأخرى المتطابقة كليا معها يتم تفكيكها بواسطة مركب ريسك. أما الرنا الميكروي فمصدره داخلي من الخلية ويشفر سواء: من جيناتٍ خاصةٍ بجزيئات رنا ميكروي أو من مناطق داخل إنترونات جينات مشفرة للبروتين أو من جينات مشفرة لجزيئات رنا غير مشفرة أخرى (ليست رنا ميكروي). ينظم الرنا الميكروي التعبير الجيني بمنع ترجمة جزيئات الرنا الرسول غير المترابطة كليا معه، وتسمى هذه الآلية «إسكات الرنا». تبعا لإحصاءات المعلوماتية الحيوية حوالي 60% من الجينات البشرية تُنظَّم بواسطة الرنا الميكروي [121] وحاليا يتم إجراء بحوثات مكثفة لربط جزيئات الرنا الميكروي بمختلف الأمراض كالسرطان مثلا.[122]
بالإضافة إلى هذين النوعين الأساسيين، توجد أنواع أخرى تتحكم في تدخل الرنا مثل الرنا المتآثر مع بيوي الذي يلعب دورا في الدفاع ضد الينقولات الراجعة وجزيئات الرنا المتدخل الصغير المشتقة من التسلسلات المتكررة. تُستخدم جزيئات رنا الحلقة الجذعية الصغير (shRNA) لأغراض البحث العلمي وإنتاج الدواء وهي جزيئات رنا تتعرف عليها عملية تدخل الرنا وتقوم بتعديلها أكثر هي وجزيئات الرنا الأولية الخاصة بالرنا الميكروي والرنا المتدخل الصغير.[123] رغم أن عملية تدخل الرنا معروفة لدى حقيقيات النوى فقط، إلا أن البكتيريا والعتائق طورتا عملية مماثلة لها تسمى نظام كريسبر للدفاع ضد العاثيات وعناصر الدنا الطفيلية الأخرى.[124]
تحدث عملية التنظيم الجيني بواسطة الرنا على عدة مستويات، على سبيل المثال: بتحوير بنية الكروماتين في النواة، ومن الأمثلة النموذجية على ذلك الرنا غير المشفر الطويل XIST الذي يُعطل لدى إناث الثدييات أحد الصبغيين X بالالتفاف حوله مانعا ترجمة الجينات الموجودة به.
جينومات الرنا

مثل الدنا يمكن للرنا حمل وتخزين المعلومة الوراثية، فيروسات الرنا لها جينومات مكونة حصريا من الرنا مثل فيروس الإنفلونزا، فيروس العوز المناعي البشري، فيروس التهاب الكبد C، فيروس شلل الأطفال وفيروس إيبولا وتقوم بتشفير بعض البروتينات. يتضاعف الجينوم الفيروسي بواسطة بعض هذه البروتينات في حين تقوم البروتينات الأخرى بحماية الفيروس أثناء انتقاله إلى خلايا مضيفة أخرى. الفيرويدات هي نوع آخر من الممرضات، وتتكون من الرنا فقط ولا تشفر أية بروتينات وتتضاعف بواسطة بوليميراز خلية النبات المضيفة.[125]
تنقسم فيروسات الرنا حسب طريقة تضاعفها إلى ثلاث أنواع:
- الرنا مزدوج السلاسل: ويحتوي من واحد إلى عدة أنواع من جزيئات الرنا، كل واحد منها يشفر بروتينا أو عدة بروتينات.[126]
- رنا أحادي السلسلة موجب الاتجاه: يستخدم جينومه مباشرة كرنا رسول، تترجمه ريبوسومات المضيف إلى بروتين وحيد يتم تعديله بواسطة بروتينات المضيف والفيروس لتشكيل مختلف البروتينات المطلوبة للتضاعف. أحد هذه البروتينات هو بوليميراز الرنا المعتمد على الرنا (منتسخة الرنا)، الذي ينسخ الرنا الفيروسي لتشكيل نسخة مزدوجة السلاسل. بدورها توجِّه هذه النسخة المزدوجة تكوين جزيئات رنا جديدة.[127]
- رنا أحادي السلسلة سالب الاتجاه: يتطلب نسخ جينومه بواسطة منتسخة الرنا لتشكيل رنا موجب الاتجاه، وهذا يعني أن على الفيروس إحضار إنزيم نسخ الرنا معه. بعدها يعمل جزيء الرنا موجب الاتجاه كرنا رسول ويترجم إلى بروتينات بواسطة الريبوسوم.[15]
لدى الفيروسات الراجعة جينوم ذو سلسلة رنا أحادية لكنها لا تعتبر من فيروسات الرنا لأنها تستخدم دنا وسيط للتضاعف. يحوِّل إنزيم النسخ العكسي الفيروسي -الذي يأتي من الفيروس نفسه بعد أن يزيل كسائه البروتيني- الرنا الفيروسي إلى سلسلة دنا مكملة والتي تُنسخ بدورها لتكوين جزيئة دنا فيروسية مزدوجة السلاسل. بعد ذلك يقوم إنزيم الدمج بدمج سلسلة الدنا المزدوجة هذه في جينوم المضيف، التعبير عن الجينات الفيروسية المدمجة قد يقود إلى نشوء فيروسات جديدة.[16]
طرق دراسة الرنا
دراسة الرنا مماثلة لدراسة الدنا في العديد من النواحي، فاستخلاص الرنا يتم بتحطيم الخلية وتمزيق غشائها، وتمسيخ البروتينات بواسطة مركبات كيميائية، وعكس استخلاص الدنا الذي يمكن فيه تثبيط الإنزيمات القاصة للدنا (دناز) بمجرد الارتباط بالأيونات ثنائية التكافؤ، يجب عند استخلاص الرنا إبقاء الإنزيمات القاصة للرنا (رناز) غير نشطة بالتبريد أو عبر عوامل تمسيخ شديدة الفعالية مثل كلوريد الغوانيدينيوم. حاليا أكثر الطرق شيوعا في استخلاص الرنا تستخدم استخلاص فينول-كلوروفورم بوجود كلوريد الغوانيدينيوم.[128] بشكل عام، الريبونوكليازات مشكل كبير في دراسة الرنا بسبب قدرتها على تحمل الحرارة وإعادة التشكل والتطوي عند التمسخ.
عند استخلاص مجموع الرنا الخلوي، حوالي 98% من العينة عبارة عن رنا ريبوسومي وهذا يعقد دراسة الأنواع الأقل تواترا. يمكن تفكيك الرنا الريبوسومي المتواجد بكثرة إنزيميا أو عبر استخدام طرق أخرى.[129] أكثر الطرق المستخدمة في استخلاص أنواع الرنا الأقل تواترا هي استخلاص الرنا الرسول الخاص بحقيقيات النوى باستخدام مُشرِع قليل الثيميدين (oligo(dt) primer) لتهجين ذيل عديد الأدينيلات الخاص بالرنا الرسول، وهذا يسمح باستخلاص جميع جزيئات الرنا الرسول وكذلك الأنواع الأخرى التي تحتوي على ذيل عديد الأدينين (مثل الرنا غير المشفر الطويل). من الطرق الأخرى لاستخلاص جزيئات الرنا المستهدفة من خلائطها: الطرد المركزي الفائق لفصل الرنا أو البروتينات النووية الريبوزية حسب كثافة الطفو وحسب الاستشراب بالألفة أو عبر الترسيب المناعي للمكون البروتيني الخاص بالبروتين النووي الريبوزي واستخلاص المكون الرنوي المتبقي.
يُستخدم الرحلان الكهربائي بهلام الأغاروز أو عديد الأكريلاميد بتواجد عوامل تمسيخ تقوم بفك البنية الثانوية للرنا لدراسة الرنا المستخلص. بعد الرحلان الكهربائي، من الممكن تحديد جزيئات معينة من الرنا عبر تقنية لطخة نورثرن التي يُنقل فيها الرنا من الهلام إلى غشاء يتم فيه تحديد أنواع الرنا عبر تهجينها بأحماض نووية مشعة أو موسومة بمواد فلورية والتي تعمل كمسابير تهجين.

رغم طرق الدراسة هذه، إلى أن تحديد أنواع وتسلسل الرنا حاليا يتم في أغلب الأحيان عبر نسخ الرنا إلى دنا ومضاعفته بواسطة تفاعل البوليمراز المتسلسل للنسخ العكسي وينتج عن ذلك دنا متمم للرنا المدروس. هنالك أسباب عديدة لاستخدام هذه الطريقة منها الاستقرارية العالية للدنا وحقيقة أن إنزيمات الدنا أكثر فعالية وأقل ميولا للخطأ. يمكن دراسة الدنا المتمم المتحصل عليه بطرق متعددة، على سبيل المثال تحديد الكمية الأصلية لنُسَخِ الرنا في عينة ما بواسطة تفاعل البوليميراز المتسلسل الكمي والذي يحدد عدد جزيئات الرنا الرسول المتواجدة في خلية ما لجين معين، أي مقدار وقوة نسخ الجين. يمثل مجموع جزيئات الدنا المتممة المتحصل عليها والذي يسمى مكتبة الدنا المتمم جميع الجينات التي نُسخت إلى رنا ويمكن دراستها بطرق متعددة. من الطرق المميزة في دراسة مجموعة كبيرة من الجينات هي مصفوفة الدنا الدقيقة التي تُسخدم فيها آلاف مسابير التهجين المتنوعة المشتقة من جينات معروفة، في هذه الطريقة ترتبط جزيئات الدنا المتممة في العينة بمسابير التهجين المثبتة على سطح الرقاقة (المصفوفة) ولأن العينة تكون موسومة في الغالب بالفلورية فإن الإرتباط يعطي إشارة معينة، بعد الغسيل لا تبقى الإشارة سوى في مكان ارتباط الدنا المتمم بالمسبار. بعد ذلك يتم التعرف على نُسَخ الجينات الجديدة عبر سَلسَلَة (تحديد تسلسل) تسلسلات الدنا المتممة القصيرة والتي تسمى تسلسل وسم معبر عنه (EST). حاليا سَلسَلة جميع جزيئات الرنا في عينة ما يتم باستخدام جيل جديد من طرق تحديد التسلسل يسمى سلسلة الرنا.
من بين الطرق الأخرى لدراسة الرنا: التهجين الموضعي المتألق ويسمح برؤية مكان رنا معين في الخلية، برفلة الريبوسوم الذي يحدد جزيئات الرنا الرسول التي في طور الترجمة، أو طرق تدخل الرنا لتنظيم التعبير الجيني في الخلايا.
استعمالات علاجية
الرنا الرسول
اقتٌرِح استخدام جزيئات رنا رسول كاملة الطول منذ بداية حقبة التقانة الحيوية، لكنها لم تلق اهتماما حتى العقد 2010، حين أُسِست شركة «مودرنا ثريبيوتيكا» وتمكنت من جمع مليار دولار تقريبا لتمويل المشروع خلال الأعوام الثلاثة الأولى.[130][131][132][133] نظريا جزيئات الرنا الرسول المعدلة يمكن أن تترجمها الخلية إلى بروتينات يمكنها معالجة الأمراض أو استخدامها كلقاحات، أو حتى أن تقود البروتينات الخلايا الجذعية الأصلية إلى التمايز بالطريقة المرغوب فيها.[134][135] لكن الإشكال الأساسي للعلاج بالرنا يتركز حول توصيل الرنا إلى الخلايا المستهدفة، أكثر من تركيزه على التسلسل الذي سيتم توصيله.[131] الرنا غير المعدل سيتم تفكيكه طبيعيا بعد تحضيره، وقد يثير النظام المناعي للجسم ليهاجمه كسجم دخيل، بالإضافة إلى أنه غير قابل للنفاذ عبر الغشاء الخلوي.[135] وحين يكون داخل الخلية يجب أن يغادر آلية النقل ليقوم بعمله في السيتوبلازم أين توجد الريبوسومات التي تترجمه إلى بروتينات.[134]
الرنا الصغير المتدخل
يُستخدم الرنا اليوم في العديد من التطبيقات في علم الأحياء الجزيئي، وبشكل خاص عبر عملية تدخل الرنا والتي يتم فيها إدراج جزيئات رنا قصيرة مزدوجة السلاسل تسمى الرنا الصغير المتدخل (siRNA) داخل خلايا حقيقيات النوى. تُستخدم جزيئات الرنا هذه التي طولها حوالي 20 زوج قاعدي بواسطة آلية خلوية قادرة على تفكيك الرنا بشكل محدد. لا يتم تفكيك سوى جزيئات الرنا الرسول التي تحوي تسلسلا مماثلا للرنا المتدخل، وهذا يتيح إمكانية تخفيض التعبير عن البروتينات بشكل محدد.[136] هذا النهج التقني أكثر بساطة وسرعة من تحضير سلالات فئران معطلة الجين ويسمى knock down. التجارب التي تستخدم هذه التقنية لأغراض علاجية هي في مرحلة الدراسة، على سبيل المثال: تُدرس إمكانية استهداف الجينات الفيروسية لمحاربة العدوى والأمراض، أو الجينات الورمية للقضاء على السرطان.[17] لكن يتطلب ذلك جعل جزيئات الرنا المتدخل مستقرة لمنع تحليلها بواسطة الريبونوكليازات وتوجيه نشاطها نحو الخلايا المستهدفة.
فرضية عالم الرنا وأصل الحياة
فرضية عالم الرنا هي مرحلة افتراضية قديمة لتاريخ الحياة في الأرض تميزت بجزيئات رنا ذاتية التضاعف والتحفيز قبل تطور الدنا والبروتين.[137] اعتقادُ أن الحياة على الأرض تطورت من عالم الرنا مقبولٌ على نطاق واسع[138][139] رغم أن الحياة المعتمدة على الرنا يمكن أن لا تكون أول أنواع الحياة وجودا.[140][141] جاءت هذه الخلاصة من العديد من الدلائل المستقلة عن بعضها، كملاحظة أن الرنا أساسي لعملية الترجمة وأن جزيئات الرنا الصغيرة يمكنها تحفيز جميع المجموعات الكيميائية ونقل المعلومة الضروري للحياة.[141][142] سميت بنية الريبوسوم «بالدليل القاطع» لأنها أظهرت أن الريبوسوم عبارة عن ريبوزيم، حيث يتميز بلب مركزي مكون من الرنا ولا توجد أي سلسلة ببتيدية على مسافة 18 أنغستروم من الموقع النشط أين يُحفز تكوين الرابطة الببتيدية.[140] اقترح مفهوم عالم الرنا لأول مرة سنة 1962 بواسطة ألكسندر ريتش[143] وصيغ المصطلح بواسطة ولتر غيلبرت سنة 1986.[141][144]
من الأسلاف المحتملة لتطور تخليق البروتين نشوء آلية لتخليق عوامل مرافقة ببتيدية قصيرة أو آلية لتضاعف الرنا. من المرجح أن الريبوسوم السلف كان مكونا بالكامل من الرنا، ثم تولت البروتينات القيام ببعض وظائفه. من الأسئلة الكبيرة المتبقية في هذا الموضوع هي تحديد القوة الانتقائية التي أدت إلى تطور الريبوسوم وكيفية نشوء الشيفرة الجينية على هيئتها الحالية.[145]
التاريخ

قاد البحث في الرنا إلى العديد من الاكتشافات البيولوجية والعديد من جوائز نوبل. اكُتشفت الأحماض النووية سنة 1868 بواسطة فريدريك ميسشر الذي أسماها «نوكليين» لأنه وجدها في النواة.[146] ثم اكتُشف لاحقا أن خلايا بدائيات النوى -التي ليس لديها نواة- تحتوي على أحماض نووية كذلك. دور الرنا في تخليق البروتين كان محل تخمين منذ 1939.[147] فاز سيفيرو أوتشوا بجائزة نوبل في الطب (مشاركة مع آرثر كورنبرغ) بعد اكتشافه إنزيما يمكنه تخليق الرنا في المختبر.[148] لكن اتضح لاحقا أن الإنزيم المكتشف بواسطة أوتشوا (فوسفوريلاز عديد النوكليوتيد) مسؤول عن تفكيك الرنا وليس تخليقه. في سنة 1956 هجَّن أليكس ريتش وديفيد دافيس سلسلتي رنا منفصلتين لتشكيل أول بلورة رنا كانت بنيته قابلة للتحديد بواسطة علم البلورات السيني.[149]
حدد روبرت هولي تسلسل نوكليوتدات الرنا الناقل الخاص بالخميرة والتي عددها 77 سنة 1965،[150] وفاز بجائزة نوبل للطب سنة 1968 (مشاركة مع هار غوبند خورانا ومارشال نيرنبرغ). خلال بداية العقد 1970 اكتُشفت الفيروسات الراجعة والمنتسخة العكسية، وأظهر ذلك لأول مرة أن الرنا يمكن نسخه إلى دنا. نتيجة لهذا الاكتشاف نال ديفيد بلتيمور وريناتو دولبيكو وهوارد تيمن جائزة نوبل سنة 1975. في سنة 1976 حدد وولتر فيرز وفريقه أول تسلسل نوكليوتيدات كامل لجينوم فيروس رنا، وهي العاثية MS2.[151] في سنة 1977 تم اكتشاف الإنترونات وتضفير الرنا عند كل من فيروسات الثدييات وفي الجينات الخلوية فحصل فيليب شارب وريتشارد روبرتس على جائزة نوبل سنة 1993. اكتُشِفت جزيئات الرنا المحفزة (الريبوزيمات) في بداية العقد 1980 وأدى ذلك إلى حصول توماس تشيك وسيدني ألتمان على جائزة نوبل سنة 1989. في العقد 1990 وُجد لدى البتونيا أن إضافة جينات يمكن أن يُسكِت جينات أخرى مماثلة خاصة بالنبتة، وهو ما يُعرف الآن بكونه نتيجة لتداخل الرنا.[152][153]
في نفس الوقت تقريبا، وُجد أن لجزيئات رنا طولها 22 نوكليوتيد -تعرف حاليا باسم الرنا الميكروي- دور في تطور الربداء الرشيقة.[154] وحصل أندرو فاير وكريغ ميلو على جائزة نوبل سنة 2006 نتيجة أبحاثهم في تداخل الرنا، كما نال روجر كورنبيرغ جائزة نوبل في نفس العام عن دراساته لنسخ الرنا. قاد اكتشاف جزيئات الرنا المنظِّمة إلى تطوير أدوية مصنوعة من الرنا مثل الرنا الصغير المتدخل لإسكات الجينات.[155] وفي سنة 2009 نال فينكاترامان راماكريشنان وتوماس ستايتز وعادا يونات جائزة نوبل لتوضيحهم البنية الذرية للريبوسوم.
ملاحظات
- 1 اعتُمد اختصار رنا في هذه المقالة، وذلك تيسيرًا لكتابتها ولقرائتها، وأيضًا كونه الأكثر شيوعًا بين الاختصارات الأُخرى.
- 2 حمض نووي ريبوزي منقوص الأكسجين ويُعرف اختصارًا دنا (DNA).
المراجع
- Toor (01 ديسمبر 2009)، Keating, K. S.; Fedorova, O.; Rajashankar, K.; Wang, J.; Pyle, A. M.، "Tertiary architecture of the Oceanobacillus iheyensis group II intron"، RNA، 16 (1): 57–69، doi:10.1261/rna.1844010.
- Analysis of Chromosomes، 2014، ISBN 978-93-84568-17-7، مؤرشف من الأصل في 03 مارس 2020، اطلع عليه بتاريخ أكتوبر 2020.
{{استشهاد بكتاب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة) - "The RNA Modification Database, RNAMDB: 2011 update"، Nucleic Acids Research، 39 (Database issue): D195–201، يناير 2011، doi:10.1093/nar/gkq1028، PMC 3013656، PMID 21071406.
- TRNA: Structure, biosynthesis, and function، ASM Press، 1995، ص. 165، ISBN 978-1-55581-073-3، OCLC 183036381.
- "Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs"، The EMBO Journal، 20 (14): 3617–22، يوليو 2001، doi:10.1093/emboj/20.14.3617، PMC 125535، PMID 11447102.
- (بالإنجليزية) S.A. Woodson, « Metal ions and RNA folding: a highly charged topic with a dynamic future », في Curr. Opin. Chem. Biol., vol. 9, no 2, Avril 2005, ص. 104-9 [النص الكامل, lien PMID (pages consultées le 6 novembre 2009)]
- (بالإنجليزية) N.B. Leontis et E. Westhof, « The non-Watson-Crick base pairs and their associated isostericity matrices. », في Nucleic Acids Res., vol. 30, no 16, 2002, ص. 3497-3531 [lien PMID]
- "The hidden genetic program of complex organisms"، Scientific American، 291 (4): 60–7، أكتوبر 2004، Bibcode:2004SciAm.291d..60M، doi:10.1038/scientificamerican1004-60، PMID 15487671.[وصلة مكسورة]
- "Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms" (PDF)، BioEssays، 25 (10): 930–9، أكتوبر 2003، CiteSeerX 10.1.1.476.7561، doi:10.1002/bies.10332، PMID 14505360، مؤرشف من الأصل (PDF) في 06 مارس 2009.
- "Non-coding RNAs: the architects of eukaryotic complexity"، EMBO Reports، 2 (11): 986–91، نوفمبر 2001، doi:10.1093/embo-reports/kve230، PMC 1084129، PMID 11713189.
- "The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms"، Molecular Biology and Evolution، 18 (9): 1611–30، سبتمبر 2001، doi:10.1093/oxfordjournals.molbev.a003951، PMID 11504843، مؤرشف من الأصل في 7 يوليو 2009.
- (بالإنجليزية) S. Brenner, F. Jacob et M. Meselson, « An unstable intermediate carrying information from genes to ribosomes for protein synthesis. », في نيتشر (مجلة), vol. 190, 1961, ص. 576-581
- (بالإنجليزية) M.B. Hoagland, M.L Stephenson, J.F. Scott, H.I. Hecht et P.C. Zamecnik, « A soluble ribonucleic acid intermediate in protein synthesis », في J. Biol. Chem., vol. 231, 1958, ص. 241-257 [lien PMID]
- Fire et al. 1998 “Potent and Specific Genetic Interference by double stranded RNA in Ceanorhabditis elegans” Nature 391:806–11.
- Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (2015) Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses. Elife 4. doi: 10.7554/eLife.05378.
- "Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes"، Genetics، 166 (3): 1437–50، مارس 2004، doi:10.1534/genetics.166.3.1437، PMC 1470764، PMID 15082561.
- Claude Hélène, « Les promesses de l'ARN thérapeutique = Genetic interference by RNA. », في Le Concours Médical, vol. 124, 2002, ص. 2550-2552
- "ترجمة (Ribonucleic acid) في المعجم الطبي الموحد"، مكتبة لبنان ناشرون، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة (Ribonucleic acid)"، www.alqamoos.org، مؤرشف من الأصل في 11 نوفمبر 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة ومعنى ribonucleic acid بالعربي في قاموس المعاني. قاموس عربي إنجليزي"، www.almaany.com، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة (Ribonucleic acid) في قاموس مرعشي الطبي الكبير"، مكتبة لبنان ناشرون، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة (Ribonucleic acid) في المعجم الطبي الموحد"، مكتبة لبنان ناشرون، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة (RNA)"، www.alqamoos.org، مؤرشف من الأصل في 10 نوفمبر 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة (RNA) على المعجم الطبي الموحد"، مكتبة لبنان ناشرون، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "LDLP - ترجمة (RNA) في المعجم الطبي"، مكتبة لبنان ناشرون، مؤرشف من الأصل في 4 يونيو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "ترجمة ومعنى rna بالعربي في قاموس المعاني"، www.almaany.com، مؤرشف من الأصل في 28 مايو 2019، اطلع عليه بتاريخ 4 يونيو 2019.
- "RNA: The Versatile Molecule"، جامعة يوتا، 2015، مؤرشف من الأصل في 20 يوليو 2016.
- "Nucleotides and Nucleic Acids" (PDF)، جامعة كاليفورنيا (لوس أنجلوس)، مؤرشف من الأصل (PDF) في 4 مارس 2016.
- Biochemistry (ط. 5th)، WH Freeman and Company، 2002، ص. 118–19, 781–808، ISBN 978-0-7167-4684-3، OCLC 179705944.
- "How RNA folds"، Journal of Molecular Biology، 293 (2): 271–81، أكتوبر 1999، doi:10.1006/jmbi.1999.3001، PMID 10550208.
- "RNA secondary structure: physical and computational aspects"، Quarterly Reviews of Biophysics، 33 (3): 199–253، أغسطس 2000، doi:10.1017/S0033583500003620، PMID 11191843.
- "The structural basis of ribosome activity in peptide bond synthesis"، Science، 289 (5481): 920–30، أغسطس 2000، Bibcode:2000Sci...289..920N، doi:10.1126/science.289.5481.920، PMID 10937990.
- "Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs"، Journal of Molecular Biology، 344 (5): 1225–49، ديسمبر 2004، doi:10.1016/j.jmb.2004.09.072، PMID 15561141.
- H., Lodish (2005)، Biologie moléculaire de la cellule (ط. 3)، Bruxelles: de Boeck، ISBN 978-2804148027.
{{استشهاد بكتاب}}: يحتوي الاستشهاد على وسيط غير معروف وفارغ:|month=(مساعدة) - Wolfram, Saenger (1984)، Principles of nucleic acid structure (باللغة الإنجليزية)، Springer، ISBN 0-387-90762-9.
{{استشهاد بكتاب}}: يحتوي الاستشهاد على وسيط غير معروف وفارغ:|month=(مساعدة) - Jan, Barciszewski (1999)، RNA biochemistry and biotechnology (باللغة الإنجليزية)، Springer، ISBN 0-7923-5862-7.
{{استشهاد بكتاب}}: يحتوي الاستشهاد على وسيط غير معروف وفارغ:|month=(مساعدة) - RNA biochemistry and biotechnology، Springer، 1999، ص. 73–87، ISBN 978-0-7923-5862-6، OCLC 52403776.
- Nucleic Acid Architecture-Conformational considerations-Ribose ring pucker نسخة محفوظة 17 مايو 2018 على موقع واي باك مشين.
- Sugar pucker correlates with phosphorus-base distance نسخة محفوظة 01 أغسطس 2018 على موقع واي باك مشين.
- (بالإنجليزية) M. Sudaralingam, « Stereochemistry of nucleic acids and their constituents. IV. Allowed and preferred conformations of nucleosides, nucleoside mono-, di-, tri-, tetraphosphates, nucleic acids and polynucleotides », في Biopolymers, vol. 7, no 6, 1969, ص. 821-860 [النص الكامل] نسخة محفوظة 29 نوفمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) R. Langridge et P.J. Gomatos, « The Structure of RNA. Reovirus RNA and transfer RNA have similar three-dimensional structures, which differ from DNA. », في ساينس, vol. 141, no 4, 1963, ص. 694-698 [lien PMID]
- Clinical gene analysis and manipulation: Tools, techniques and troubleshooting، Cambridge University Press، 1996، ص. 14، ISBN 978-0-521-47896-0، OCLC 33838261، مؤرشف من الأصل في 1 مايو 2021.
{{استشهاد بكتاب}}: صيانة CS1: التاريخ والسنة (link) - "Identification of critical elements in the tRNA acceptor stem and T(Psi)C loop necessary for human immunodeficiency virus type 1 infectivity"، Journal of Virology، 75 (10): 4902–06، مايو 2001، doi:10.1128/JVI.75.10.4902-4906.2001، PMC 114245، PMID 11312362.
- "Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine"، The Journal of Biological Chemistry، 259 (4): 2407–10، فبراير 1984، PMID 6365911.
- "Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center"، Molecular Cell، 11 (2): 425–35، فبراير 2003، doi:10.1016/S1097-2765(03)00040-6، PMID 12620230.
- (بالإنجليزية) H.R. Drew, R.M. Wing, T. Tanako, C Broka, S Tanaka, K Itakura et R.E. Dickerson, « Structure of a B-DNA dodecamer: conformation and dynamics. », في Proc. Natl. Acad. Sci. USA, vol. 78, no 4, avril 1981, ص. 2179-2183 [النص الكامل, lien PMID] نسخة محفوظة 29 نوفمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) Peter S. Klosterman, Sapan A. Shah et Thomas A. Steitz, « Crystal structures of two plasmid copy control related RNA duplexes: An 18 base pair duplex at 1.20 A resolution and a 19 base pair duplex at 1.55 A resolution. », في Biochemistry, vol. 38, no 45, 1999, ص. 14784-14792 [النص الكامل, lien PMID] نسخة محفوظة 16 ديسمبر 2019 على موقع واي باك مشين.
- "Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing"، Nucleic Acids Research، 34 (21): 6233–46، 2006، doi:10.1093/nar/gkl886، PMC 1669714، PMID 17090584.
- "RNA interference: potential therapeutic targets"، Applied Microbiology and Biotechnology، 65 (6): 649–57، نوفمبر 2004، doi:10.1007/s00253-004-1732-1، PMID 15372214.
- "The interferon system of non-mammalian vertebrates"، Developmental and Comparative Immunology، 28 (5): 499–508، مايو 2004، doi:10.1016/j.dci.2003.09.009، PMID 15062646.
- "Silencing or stimulation? siRNA delivery and the immune system"، Annual Review of Chemical and Biomolecular Engineering، 2: 77–96، 2011، doi:10.1146/annurev-chembioeng-061010-114133، PMID 22432611.
- (بالإنجليزية) J.M. Rosenberg, N.C. Seeman, J.J. Kim, F.L. Suddath, H.B. Nicholas et A. Rich, « Double helix at atomic resolution. », في Nature, vol. 243, no 5403, 1973, ص. 150-154 [النص الكامل, lien PMID (pages consultées le 6 novembre 2009)] نسخة محفوظة 29 يونيو 2016 على موقع واي باك مشين.
- (بالإنجليزية) R.O. Day, N.C. Seeman, J.M. Rosenberg et A. Rich, « A Crystalline Fragment of the Double Helix: The Structure of the Dinucleoside Phosphate Guanylyl-3',5'-Cytidine. », في Proc. Natl. Acad. Sci. USA, vol. 70, no 3, Mars 1973, ص. 849-853 [النص الكامل, lien PMID (pages consultées le 6 novembre 2009)] نسخة محفوظة 30 نوفمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) Alexander Rich et David R. Davies, « A new two stranded helical structure: Polyadenylic acid and polyuridylic acid », في J. Am. Chem. Soc., vol. 78, no 14, 1956, ص. 3548-3549 [النص الكامل (pages consultées le 6 novembre 2009)] نسخة محفوظة 8 أغسطس 2020 على موقع واي باك مشين.
- (بالإنجليزية) D.E. Draper, « Protein-RNA recognition », في Annu. Rev. Biochem., vol. 64, 1995, ص. 593-620 [النص الكامل, lien PMID (pages consultées le 6 novembre 2009)] نسخة محفوظة 29 نوفمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) P. Doty, H. Boedtker, J.R. Fresco, R. Haselkorn et M. Litt, « Secondary Structure in Ribonucleic Acids », في Proc. Natl. Acad. Sci. USA, vol. 45, no 4, 1959, ص. 482-499 [lien PMID]
- F., Dardel [الفرنسية] (2002)، Bioinformatique : génomique et post-génomique، Editions de l'École Polytechnique، ISBN 978-2730209274.
{{استشهاد بكتاب}}: يحتوي الاستشهاد على وسيط غير معروف وفارغ:|month=(مساعدة) - (بالإنجليزية) A.M. Michelson, « Hyperchromicity and nucleic acids. », في Nature, vol. 182, no 4648, 1958, ص. 1502-1503 [lien PMID]
- (بالإنجليزية) K. Hoogsteen, « The crystal and molecular structure of a hydrogen-bonded complex between 1-methylthymine and 9-methyladenine. », في Acta Cryst., vol. 16, 1963, ص. 907-916 [lien DOI]
- (بالإنجليزية) H.A. Heus et A. Pardi, « Structural features that give rise to the unusual stability of RNA hairpins containing GNRA loops. », في Science, vol. 253, no 5016, 1991, ص. 191-194 [lien PMID]
- (بالإنجليزية) D.W. Staple et S.E. Butcher, « Pseudoknots: RNA Structures with Diverse Functions. », في PloS Biol., vol. 3, no 6, 2005, ص. e213 [النص الكامل, lien PMID] نسخة محفوظة 24 سبتمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) M. Costa et F. Michel, « Frequent use of the same tertiary motif by self-folding RNAs », في مجلة EMBO العلمية, vol. 14, 1995, ص. 1276–1285 [النص الكامل, lien PMID] نسخة محفوظة 29 نوفمبر 2019 على موقع واي باك مشين.
- "Transcription termination and anti-termination in E. coli"، Genes to Cells، 7 (8): 755–68، أغسطس 2002، doi:10.1046/j.1365-2443.2002.00563.x، PMID 12167155.
- "Structure of the RNA-dependent RNA polymerase of poliovirus"، Structure، 5 (8): 1109–22، أغسطس 1997، doi:10.1016/S0969-2126(97)00261-X، PMID 9309225.
- "RNA-dependent RNA polymerases, viruses, and RNA silencing"، Science، 296 (5571): 1270–73، مايو 2002، Bibcode:2002Sci...296.1270A، doi:10.1126/science.1069132، PMID 12016304.
- (بالإنجليزية) B.A. Hamkalo et O.L. Miller, « Electronmicroscopy of genetic activity », في Annu. Rev. Biochem., vol. 42, 1973, ص. 376-396 [lien PMID]
- (بالإنجليزية) S.T. Smale et J.T. Kadonaga, « The RNA polymerase II core promoter », في Ann. Rev. Biochem., vol. 72, 2003, ص. 449-479 [النص الكامل, lien PMID] نسخة محفوظة 17 ديسمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) R.P. Lifton, M.L. Goldberg, R.W. Karp et D.S. Hogness, « The organization of the histone genes in Drosophila melanogaster: functional and evolutionary implications », في Cold Spring Harb. Symp. Quant. Biol., vol. 42, 1978, ص. 1047-1051 [النص الكامل, lien PMID] نسخة محفوظة 21 نوفمبر 2018 على موقع واي باك مشين.
- (بالإنجليزية) D. Pribnow, « Nucleotide sequence of an RNA polymerase binding site at an early T7 promoter », في Proc. Natl. Acad. Sci. USA, vol. 72, 1975, ص. 784-788 [النص الكامل, lien PMID] نسخة محفوظة 19 ديسمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) W.R. McClure et Y. Chow, « The kinetics and processivity of nucleic acid polymerases », في Methods Enzymol., vol. 64, 1980, ص. 277-297 [النص الكامل, lien PMID] نسخة محفوظة 1 مايو 2021 على موقع واي باك مشين.
- قالب:Chapitre "نسخة مؤرشفة" (PDF)، مؤرشف من الأصل (PDF) في 4 مارس 2016، اطلع عليه بتاريخ 15 مايو 2019.
- (بالإنجليزية) S. Adhya et M. Gottesman, « Control of transcription termination », في Annu. Rev. Biochem., vol. 47, 1978, ص. 967-996 [النص الكامل, lien PMID] نسخة محفوظة 23 ديسمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) M.S. Ciampi, « Rho-dependent terminators and transcription termination », في Microbiology, vol. 152, 2006, ص. 2515-2528 [النص الكامل, lien PMID] نسخة محفوظة 3 أكتوبر 2015 على موقع واي باك مشين.
- (بالإنجليزية) M. Edmonds, « A history of poly A sequences: from formation to factors to function », في Prog. Nucleic Acid res. Mol. Biol., vol. 71, 2002, ص. 285-389 [النص الكامل, lien PMID] نسخة محفوظة 29 نوفمبر 2019 على موقع واي باك مشين.
- (بالإنجليزية) A.K. Banerjee, « 5'-terminal cap structure in eucaryotic messenger ribonucleic acids », في Microbiol. Rev, vol. 44, no 2, 1980, ص. 175-205 [النص الكامل, lien PMID]
- (بالإنجليزية) M. Edmonds et R. Abrams, « Polynucleotide biosynthesis: formation of a sequence of adenylate units from adenosine triphosphate by an enzyme from thymus nuclei », في Journal of Biological Chemistry, vol. 235, 1960, ص. 1142-1149 [النص الكامل, lien PMID] نسخة محفوظة 25 مايو 2019 على موقع واي باك مشين.
- (بالإنجليزية) M. Dreyfus et P. Régnier, « The poly(A) tail of mRNAs: bodyguard in eukaryotes, scavenger in bacteria », في Cell, vol. 111, 2002, ص. 611-613 [النص الكامل, lien PMID]
- (بالإنجليزية) J.P. Staley et C. Guthrie, « Mechanical devices of the spliceosome: motors, clocks, springs, and things », في Cell, vol. 92, 1998, ص. 315-326 [النص الكامل, lien PMID] نسخة محفوظة 1 مايو 2021 على موقع واي باك مشين.
- Anderson, N.؛ Kedersha (مارس 2006)، "RNA granules."، J Cell Biol، 172 (6): 803–8، doi:10.1083/jcb.200512082، PMID 16520386.
- Voronina, P.؛ Seydoux (ديسمبر 2011)، et al.، "RNA granules in germ cells."، Cold Spring Harb Perspect Biol، 3 (12)، doi:10.1101/cshperspect.a002774، PMID 21768607.
- Kedersha؛ Stoecklin؛ Ayodele (يونيو 2005)، et al.، "Stress granules and processing bodies are dynamically linked sites of mRNP remodeling."، J Cell Biol، 169 (6): 871–84، doi:10.1083/jcb.200502088، PMID 15967811.
- Wang؛ van Niekerk؛ Willis (أغسطس 2007)، et al.، "RNA transport and localized protein synthesis in neurological disorders and neural repair."، Dev Neurobiol، 67 (9): 1166–82، doi:10.1002/dneu.20511، PMID 17514714.
- Kaberdin؛ Singh؛ Lin-Chao (2011)، "Composition and conservation of the mRNA-degrading machinery in bacteria."، J Biomed Sci، 18: 23، doi:10.1186/1423-0127-18-23، PMID 21418661.
- Carpousis (2007)، "The RNA degradosome of Escherichia coli: an mRNA-degrading machine assembled on RNase E."، Annu Rev Microbiol، 61: 71–87، doi:10.1146/annurev.micro.61.080706.093440، PMID 17447862.
- (بالإنجليزية) S. Valadkhan, A. Mohammadi, Y. Jaladat et S. Geisler, « Protein-free small nuclear RNAs catalyze a two-step splicing reaction. », في Proc. Natl. Acad. Sci. USA, vol. 106, 2009, ص. 11901-11906 [lien PMID]
- The Cell: A Molecular Approach (ط. 3rd)، Sinauer، 2004، ص. 261–76, 297, 339–44، ISBN 978-0-87893-214-6، OCLC 174924833.
- Mining the transcriptome – methods and applications، Stockholm: School of Biotechnology, Royal Institute of Technology، 2006، ISBN 978-91-7178-436-0، OCLC 185406288، مؤرشف من الأصل في 4 مارس 2016.
- "Ribozyme diagnostics comes of age"، Chemistry & Biology، 11 (7): 894–5، يوليو 2004، doi:10.1016/j.chembiol.2004.07.002، PMID 15271347.
- (بالإنجليزية) B.N. Ames et R.G. Martin, « Biochemical aspects of genetics: The operon. », في Annu. Rev. Biochem., vol. 33, 1964, ص. 235-258 [lien PMID]
- (بالإنجليزية) C. Yanofsky, « Establishing the triplet nature of the genetic code. », في Cell, vol. 128, 2007, ص. 815-818 [lien PMID]
- (بالإنجليزية) Huijing Shi et Peter B. Moore, « The crystal structure of yeast phenylalanine tRNA at 1.93 Å resolution: A classic structure revisited », في RNA, vol. 6, no 8, août 2000, ص. 1091-1105 [النص الكامل, lien PMID, lien DOI (pages consultées le 30 août 2014)] نسخة محفوظة 17 فبراير 2019 على موقع واي باك مشين.
- (بالإنجليزية) R.W. Holley, J. Apgar, G.A. Everett, J.T. Madison, M. Marquisee, S.H. Merrill, J.R. Penswick et A. Zamir, « Structure of a ribonucleic acid », في Science, vol. 147, 1965, ص. 1462-1465 [lien PMID]
- (بالإنجليزية) J.D. Robertus, J.E. Ladner, J.T. Finch, D. Rhodes, R.S. Brown, B.F. Clark et A. Klug, « Structure of yeast phenylalanine tRNA at 3 A resolution. », في نيتشر (مجلة), vol. 250, 1974, ص. 546–551 [lien PMID], (بالإنجليزية) S.H. Kim, F.L. Suddath, G.J. Quigley, A. McPherson, J.L. Sussman, A.H. Wang, N.C. Seeman et A. Rich, « Three-dimensional tertiary structure of yeast phenylalanine transfer RNA. », في ساينس, vol. 250, 1974, ص. 546–551 [lien PMID]
- (بالإنجليزية) Francis H. Crick, « On protein synthesis », في Symp. Soc. Exp. Biol., vol. 12, 58, ص. 138-163 [النص الكامل, lien PMID] نسخة محفوظة 29 يوليو 2020 على موقع واي باك مشين.
- "The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts"، Nucleic Acids Research، 32 (Database issue): D104-8، يناير 2004، doi:10.1093/nar/gkh102، PMC 308836، PMID 14681369.
- (بالإنجليزية) K. Kruger, P.J. Grabowski, A.J. Zaug, J. Sands, D.E. Gottschling et T.R. Cech, « Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena. », في Cell, vol. 31, 1982, ص. 147-157 [lien PMID]
- (بالإنجليزية) C. Guerrier-Takada, K. Gardiner, T. Marsh, N. Pace et S. Altman, « The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme. », في Cell, vol. 35, 1983, ص. 849-857 [lien PMID]
- (بالإنجليزية) A.C. Forster, Davies, C.J. Hutchins et R.H. Symons, « Characterization of self-cleavage of viroid and virusoid RNAs. », في Methods Enzymol., vol. 181, 1990, ص. 583-607 [lien PMID]
- (بالإنجليزية) T.R. Cech, « Structural biology. The ribosome is a ribozyme. », في ساينس, vol. 289, 2000, ص. 878-879 [lien PMID]
- (بالإنجليزية) J.E. Barrick, K.A. Corbino, W.C. Winkler, A. Nahvi, M. Mandal, J. Collins, M. Lee, A. Roth, N. Sudarsan, I. Jona, J.K. Wickiser et R.R. Breaker, « New RNA motifs suggest an expanded scope for riboswitches in bacterial genetic control. », في Proc. Natl. Acad. Sci. USA, vol. 101, 2004, ص. 6421-6426 [lien PMID]
- (بالإنجليزية) A.D. Ellington et J.W. Szostak, « In vitro selection of RNA molecules that bind specific ligands. », في نيتشر (مجلة), vol. 346, 1990, ص.
818-822 [lien PMID]
(بالإنجليزية) C. Tuerk et L. Gold, « Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. », في ساينس, vol. 249, 1990, ص. 505-510 [lien PMID] - F Jacob and J Monod (1961) "Genetic Regulatory Mechanisms in the Synthesis of Proteins. Journal of Molecular Biology 3: 318–56.
- Kevin Morris and John Mattick. (2014) “The rise of regulatory RNA” Nature Reviews Genetics15:423–37.
- S. Gottesman (2005) “Micros for microbes: non-coding regulatory RNAs in bacteria.” Trends in Genetics 21,399–404.
- "The Nobel Prize in Physiology or Medicine 2006". Nobelprize.org. Nobel Media AB 2014. Web. 6 Aug 2018. <http://www.nobelprize.org/nobel_prizes/medicine/laureates/2006/> "نسخة مؤرشفة"، مؤرشف من الأصل في 8 أغسطس 2018، اطلع عليه بتاريخ 16 سبتمبر 2020.
- (بالإنجليزية) J. Liu, « Novel intron-encoded small nucleolar RNAs. », في Cell, vol. 75, 1993, ص. 403-405 [lien PMID]
- John L. Rinn and Howard Y. Chang. (2012) “Genome regulation by long noncoding RNAs” Annu. Rev. Biochem 81:1–25. دُوِي:10.1146/annurev-biochem-051410-092902
- J Zhao, BK Sun, JA Erwin, JJ Song, and JT Lee. (2008) “Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome.” Science 322:750–56. [PubMed: 18974356],
- (بالإنجليزية) M. Huarte, T. Jacks et J.L. Rinn, « A Large Intergenic Noncoding RNA Induced by p53 Mediates Global Gene Repression in the p53 Response », في Cell, vol. 142, 2010, ص. 409-419 [lien PMID]
- (بالإنجليزية) D. Shippen-Lentz et E.H. Blackburn, « Functional evidence for an RNA template in telomerase. », في ساينس, vol. 247, 1990, ص. 546-552 [lien PMID]
- RJ Taft, CD Kaplan., C Simons, and JS Mattick, (2009). Evolution, biogenesis and function of promoter- associated RNAs. Cell Cycle 8, 2332–38.
- UA Orom, T Derrien, M Beringer, K Gumireddy, A. Gardini, et al.(2010) ‘Long noncoding RNAs with enhancer-like function in human cells.” Cell 143:46–58. [PubMed: 20887892]
- "New study shows circular RNA can encode for proteins"، ساينس ديلي، 23 مارس 2017، مؤرشف من الأصل في 27 يونيو 2018، اطلع عليه بتاريخ 3 مايو 2018.
- Pamudurti؛ Bartok؛ Jens؛ وآخرون (أبريل 2017)، https://linkinghub.elsevier.com/retrieve/pii/S1097276517301363?showall=true "Translation of CircRNAs"، Molecular Cell، 66 (1): 9–21.e7، doi:10.1016/j.molcel.2017.02.021، PMC 5387669، PMID 28344080، مؤرشف من https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS1097276517301363%3Fshowall%3Dtrue الأصل في 26 يناير 2020.
{{استشهاد بدورية محكمة}}: تحقق من قيمة|مسار أرشيف=(مساعدة)، تحقق من قيمة|مسار=(مساعدة)
- Jeck؛ Sorrentino؛ Wang؛ وآخرون (فبراير 2013)، "Circular RNAs are abundant, conserved, and associated with ALU repeats."، RNA، 19 (2): 141–57، doi:10.1261/rna.035667.112، PMC 3543092، PMID 23249747.
- J.W. Nelson, R.R. Breaker (2017) "The lost language of the RNA World."Sci. Signal.10,eaam8812 1–11.
- WC Winklef. (2005) “Riboswitches and the role of noncoding RNAs in bacterial metabolic control. “Curr. Opin. Chem. Biol. 9, 594–602.
- BJ Tucker and RR Breaker (2005). “Riboswitches as versatile gene control elements.”Curr. Opin. Struct. Biol. 15, 342–48.
- FJ Mojica, C Diez-Villasenor, E Soria, and G Juez, (2000) “Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria.”Mol. Microbiol. 36, 244–46.
- S Brouns, MM Jore, M Lundgren, E Westra, R Slijkhuis, A Snijders, M Dickman, K. Makarova, E. Koonin, J Van Der Oost. (2008) “Small CRISPR RNAs guide antiviral defense in prokaryotes” Science 321, 960–64. دُوِي:10.1126/science.1159689.
- Friedman (29 أكتوبر 2008)، "Most mammalian mRNAs are conserved targets of microRNAs"، Genome Research، 19 (1): 92–105، doi:10.1101/gr.082701.108.
{{استشهاد بدورية محكمة}}: الوسيط غير المعروف|spoluautoři=تم تجاهله (مساعدة) - Li (29 أكتوبر 2009)، "Therapeutic MicroRNA Strategies in Human Cancer"، The AAPS Journal، 11 (4): 747–757، doi:10.1208/s12248-009-9145-9.
{{استشهاد بدورية محكمة}}: الوسيط غير المعروف|spoluautoři=تم تجاهله (مساعدة) - Paddison (2002)، "Short hairpin RNAs (shRNAs) induce sequence-specific silencing in mammalian cells"، Genes & Development، 16 (8): 948–958، doi:10.1101/gad.981002.
- Horvath, R.؛ Barrangou (يناير 2010)، "CRISPR/Cas, the immune system of bacteria and archaea."، Science، 327 (5962): 167–70، doi:10.1126/science.1179555، PMID 20056882.
- "Viroids: an Ariadne's thread into the RNA labyrinth"، EMBO Reports، 7 (6): 593–8، يونيو 2006، doi:10.1038/sj.embor.7400706، PMC 1479586، PMID 16741503.
- "Analyses of the radiation of birnaviruses from diverse host phyla and of their evolutionary affinities with other double-stranded RNA and positive strand RNA viruses using robust structure-based multiple sequence alignments and advanced phylogenetic methods"، BMC Evol. Biol.، 13: 154، 2013، doi:10.1186/1471-2148-13-154، PMC 3724706، PMID 23865988.
- Francki؛ Fauquet؛ Knudson؛ Brown (1991)، Classification and nomenclature of viruses. Fifth report of the International Committee on Taxonomy of Viruses, Archives of Virology (Suppl. 2)، ISBN 978-3-7091-9163-7.
- Chomczynski؛ Sacchi (2006)، "The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on."، Nat Protoc، 1 (2): 581–5، doi:10.1038/nprot.2006.83، PMID 17406285.
- Peano (01 يناير 2013)، "An efficient rRNA removal method for RNA sequencing in GC-rich bacteria"، Microbial Informatics and Experimentation، 3 (1): 1، doi:10.1186/2042-5783-3-1.
{{استشهاد بدورية محكمة}}: الوسيط غير المعروف|spoluautoři=تم تجاهله (مساعدة) - Gregory Huang (6 ديسمبر 2012)، "Moderna, $40M in Tow, Hopes to Reinvent Biotech with "Make Your Own Drug""، Xconomy، مؤرشف من الأصل في 22 نوفمبر 2018.
- "Advances in the delivery of RNA therapeutics: from concept to clinical reality"، Genome Medicine، 9 (1): 60، يونيو 2017، doi:10.1186/s13073-017-0450-0، PMC 5485616، PMID 28655327.
- Crow (7 سبتمبر 2017)، "Moderna pledge sets investor pulses racing; Biotech group promises to rewrite rules of drug development - though sceptics say $5bn valuation is undeserved"، Financial Times، مؤرشف من الأصل في 3 فبراير 2019.
- Adams (14 سبتمبر 2017)، "Moderna changes up R&D model, posts cancer, CV trial updates"، FierceBiotech (باللغة الإنجليزية)، مؤرشف من الأصل في 2 أبريل 2019.
- "Tools for translation: non-viral materials for therapeutic mRNA delivery"، Nature Reviews Materials، 2 (10): 17056، 12 سبتمبر 2017، doi:10.1038/natrevmats.2017.56.
- "RNA-Based Therapeutics and Vaccines"، Genetic Engineering News، 15 سبتمبر 2015، مؤرشف من الأصل في 15 أكتوبر 2017.
- (بالإنجليزية) A. Fire, S. Xu, M. Montgomery, S. Kostas, S. Driver et C. Mello, « Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. », في نيتشر (مجلة), vol. 391, 1998, ص. 806-811
- Zimmer, Carl (25 سبتمبر 2014)، "A Tiny Emissary From the Ancient Past"، The New York Times، New York، مؤرشف من الأصل في 27 سبتمبر 2014، اطلع عليه بتاريخ 26 سبتمبر 2014.
-
- Copley, Shelley D.؛ Smith؛ Morowitz (ديسمبر 2007)، "The origin of the RNA world: Co-evolution of genes and metabolism" (PDF)، Bioorganic Chemistry، 35 (6): 430–443، doi:10.1016/j.bioorg.2007.08.001، PMID 17897696، مؤرشف من الأصل (PDF) في 05 سبتمبر 2013، اطلع عليه بتاريخ 08 يونيو 2015،
The proposal that life on Earth arose from an RNA world is widely accepted.
- Orgel, Leslie E. (أبريل 2003)، "Some consequences of the RNA world hypothesis"، Origins of Life and Evolution of the Biosphere، 33 (2): 211–218، doi:10.1023/A:1024616317965، PMID 12967268،
It now seems very likely that our familiar DNA/RNA/protein world was preceded by an RNA world...
- Robertson & Joyce 2012: "There is now strong evidence indicating that an RNA World did indeed exist before DNA- and protein-based life."
- Neveu, Kim & Benner 2013: "[The RNA world's existence] has broad support within the community today."
- Copley, Shelley D.؛ Smith؛ Morowitz (ديسمبر 2007)، "The origin of the RNA world: Co-evolution of genes and metabolism" (PDF)، Bioorganic Chemistry، 35 (6): 430–443، doi:10.1016/j.bioorg.2007.08.001، PMID 17897696، مؤرشف من الأصل (PDF) في 05 سبتمبر 2013، اطلع عليه بتاريخ 08 يونيو 2015،
- Wade, Nicholas (4 مايو 2015)، "Making Sense of the Chemistry That Led to Life on Earth"، The New York Times، New York، مؤرشف من الأصل في 09 يوليو 2017، اطلع عليه بتاريخ 10 مايو 2015.
- Robertson؛ Joyce (مايو 2012)، "The origins of the RNA world"، Cold Spring Harbor Perspectives in Biology، 4 (5): a003608، doi:10.1101/cshperspect.a003608، PMC 3331698، PMID 20739415.
- Cech, Thomas R. (يوليو 2012)، "The RNA Worlds in Context"، Cold Spring Harbor Perspectives in Biology، 4 (7): a006742، doi:10.1101/cshperspect.a006742، PMC 3385955، PMID 21441585.
- Yarus, Michael (أبريل 2011)، "Getting Past the RNA World: The Initial Darwinian Ancestor"، Cold Spring Harbor Perspectives in Biology، 3 (4): a003590، doi:10.1101/cshperspect.a003590، PMC 3062219، PMID 20719875.
- Neveu؛ Kim؛ Benner (22 أبريل 2013)، "The 'Strong' RNA World Hypothesis: Fifty Years Old"، Astrobiology، 13 (4): 391–403، Bibcode:2013AsBio..13..391N، doi:10.1089/ast.2012.0868، PMID 23551238.
- Gilbert, Walter (20 فبراير 1986)، "Origin of life: The RNA world"، Nature، 319 (6055): 618، Bibcode:1986Natur.319..618G، doi:10.1038/319618a0.
- Noller, Harry F. (أبريل 2012)، "Evolution of protein synthesis from an RNA world."، Cold Spring Harbor Perspectives in Biology، 4 (4): a003681، doi:10.1101/cshperspect.a003681، PMC 3312679، PMID 20610545.
- "Friedrich Miescher and the discovery of DNA"، Developmental Biology، 278 (2): 274–88، فبراير 2005، doi:10.1016/j.ydbio.2004.11.028، PMID 15680349.
- "Pentose nucleotides in the cytoplasm of growing tissues"، Nature، 143 (3623): 602–03، 1939، Bibcode:1939Natur.143..602C، doi:10.1038/143602c0.
- "Enzymatic synthesis of ribonucleic acid" (PDF)، Nobel Lecture، 1959، مؤرشف من الأصل (PDF) في 10 أغسطس 2017.
- "A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid"، Journal of the American Chemical Society، 78 (14): 3548–49، 1956، doi:10.1021/ja01595a086.
- "Structure of a ribonucleic acid"، Science، 147 (3664): 1462–5، مارس 1965، Bibcode:1965Sci...147.1462H، doi:10.1126/science.147.3664.1462، PMID 14263761.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=1غير صالح (مساعدة) - "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene"، Nature، 260 (5551): 500–7، أبريل 1976، Bibcode:1976Natur.260..500F، doi:10.1038/260500a0، PMID 1264203.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=1غير صالح (مساعدة) - "Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans"، The Plant Cell، 2 (4): 279–289، أبريل 1990، doi:10.1105/tpc.2.4.279، PMC 159885، PMID 12354959.
- "pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants"، Plant Physiology، 145 (4): 1272–81، ديسمبر 2007، doi:10.1104/pp.107.106062، PMC 2151715، PMID 17766396.
- "Molecular biology. Glimpses of a tiny RNA world"، Science، 294 (5543): 797–9، أكتوبر 2001، doi:10.1126/science.1066315، PMID 11679654.
- "The potential of oligonucleotides for therapeutic applications"، Trends in Biotechnology، 24 (12): 563–70، ديسمبر 2006، doi:10.1016/j.tibtech.2006.10.003، PMID 17045686.
وصلات خارجية
- موقع الرنا العالمي مجموعةٌ من الوصلات (الهياكل، التسلسلات، الأدوات، المجلات)
- قاعدة بيانات الحمض النووي صُورٌ للدنا والرنا والمُركبات.
- ندوة آنا ماري بيل: هيكل الحمض النووي الريبوزي، الوظيفة، والتعرف
- بوابة علم الأحياء
- بوابة علم الأحياء الخلوي والجزيئي
- بوابة الكيمياء الحيوية
- بوابة تقانة حيوية