حمض نووي ريبوزي منقوص الأكسجين
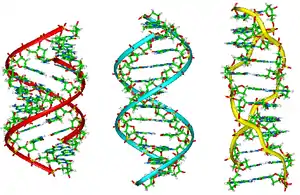
الحمض النووي الريبوزي منقوص الأكسجين أو حمض الديوكسي ريبونيوكليك[1] أو الحمض النووي الصبغي[2] (بالإنجليزية: Deoxyribonucleic acid) واختصارًا (دنا: DNA) هو جزيء ضخم يتواجد داخل خلايا كل الكائنات الحية والعديد من الفيروسات ويحتوي على المعلومات الوراثية التي تسمح بعمل وتكاثر وتطور هذه الكائنات، الدنا والحمض النووي الريبوزي (الرنا RNA) عبارة عن أحماض نووية تُشكِّل إلى جانب كل من البروتينات والليبيدات والسكريات المتعددة الجزيئات الضخمة الأربعة الضرورية للحياة، تتكون معظم جزيئات الدنا من سلسلتي بوليميرات بيولوجية ملتفة حول بعضها البعض على شكل لولب مزدوج.

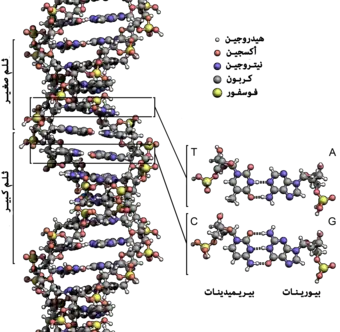
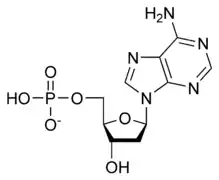
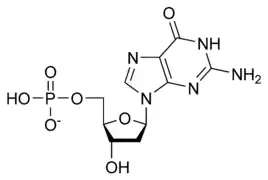
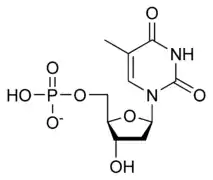
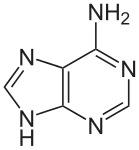
تسمى سلسلة الدنا الواحدة عديد النوكليوتيد وهي مكونة من وحدات أبسط تسمى نوكليوتيدات.[3][4] تتكون النوكليوتيدة الواحدة من سكر يسمى ريبوز منقوص الأكسجين ومجموعة فوسفات وقاعدة نيتروجينية واحدة من ضمنِ أربع قواعد نيتروجينية (أدينين [A]، ثايمين [T]، غوانين [G]، سايتوسين [C]). ترتبط النوكليوتيدات مع بعضها البعض في سلسلة عبر روابط تساهمية بين سكر أحد النوكليوتيدات وفوسفات النوكليوتيدة التالية مشكلة عمود الدنا الفقري، ترتبط قواعد سلسلتي عديد النوكليوتيد النيتروجينية مع بعضها البعض تبعا لقواعد الترابط الزوجي (A مع T وG مع C) بواسطة روابط هيدروجينية بشكل مكمل لتشكيل جزيء دنا بسلسلتين ضد متوازيتين.
فضلا عن القواعد الأربعة الأساسية تتواجد في الدنا قواعد غير معيارية مثل اليوراسيل والقاعدة ج وتختلف أسباب تواجدها بين تعرض القواعد الأساسية للمثيلة أو لكونها مواقع ارتباط إنزيمات محددة. بنية الدنا ليست ثابتة وتتحكم فيها ظروف الخلية من: مستوى التميؤ، تسلسلات الدنا، كمية واتجاه اللف الفائق، التغيرات الكيميائية للقواعد، نوع وتركيز الآيونات المعدنية ووجود متعددات الأمين في المحلول، حيث يتواجد الدنا على عدة أشكال منها الدنا أ، الدنا ب والدنا ز.
يتواجد الدنا لدى حقيقيات النوى داخل النواة على شكل كروماتين لتسهيل عملية التعبير الجيني ولا يصبح على هيئة كروموسومات سوى في مرحلة الانقسام المتساوي حيث تتم عملية التضاعف والتحزيم، في حين أنه يتواجد لدى بدائيات النوى (البكتيريا، البكتيريا القديمة) في السيتوبلازم. أثناء التعبير الجيني تتم عملية نسخ الجينات إلى جزيئات رنا رسول وترجمتها إلى بروتينات بواسطة الريبوسوم. كمية الدنا المشفِّر للبروتين لدى الإنسان لا تتجاوز 1.5% من إجمالي الدنا والباقي أكثر من 98% عبارة عن دنا غير مشفر للبروتين يمكن أن تشفر بعض تسلسلاته رنا غير مشفر مسؤول عن تنظيم التعبير الجيني. 55% من الدنا عبارة عن تسلسلات متكررة لا تعرف وظيفتها لحد الآن.
يتضرر الدنا سواء طبيعيا بسبب نشاطات الخلية الروتينية مثل نشاطات الحلمأة والإنزيمات أو بسبب العوامل الخارجية مثل المؤكسدات، عوامل الألكلة والإشعاعات الكهرومغناطيسية عالية الطاقة مثل الأشعة فوق البنفسجية والأشعة السينية حيث تُحدِث هذه الأضرار تغييرات كيميائية في طبيعة الدنا يمكن أن تؤدي إلى تثبيط أو تغيير وظيفة الجينات. توجد لدى الخلية آليات ترميم الدنا تعمل على اكتشاف وإصلاح هذه الأضرار ويعتمد معدل سرعة ترميم الدنا على عدة عوامل منها: عمر الخلية، نوعها، ومحيطها الخارجي، لكن عمليات الترميم هذه محدودة ويمكن أن تتراكم هذه التضررات مع مرور الزمن في الخلايا لتسبب السرطان، كما أن هذه التراكمات تبدو عاملا أساسيا مهما في الشيخوخة.
تقترح فرضية عالم الرنا أن أشكال الحياة الأولى من الممكن أن تكون استعملت الرنا كمادتها الوراثية قبل أن يتطور الدنا. تتطور المعلومة الوراثية عبر الزمن بسبب الطفرات والاحتياجات الطبيعية بواسطة العناصر الوراثية المتحركة كالجينات القافزة والينقولات الراجعة أو بسبب إعادة التركيب الجيني والنقل الأفقي للجينات.
للدنا استخدامات عديدة في التكنولوجيا، ففي الهندسة الوراثية يمكن تعديل النباتات والحيوانات جينيا لتحسين معدل إنتاجها ومقاومتها للأمراض والفيروسات، وفي علم الأدلة الجنائية يمكن استخلاص الدنا من مسارح الجرائم وإدانة المشتبه بهم كما يمكن باستخدام البصمة الوراثية إجراء اختبار الأبوة بالدنا لتحديد والد الطفل البيولوجي، وفي تخزين الدنا للبيانات الرقمية يمكن نظريا تخزين حوالي 455 مليون تيرابايت في كل غرام واحد من الدنا منفرد السلسلة، وفي تقنية الدنا النانوية يمكن إنشاء هياكل بأبعاد النانو لها استخدامات نظرية محتملة واسعة في: الفيزياء الحيوية مثل الأسلاك الجزيئية، علم البلورات لتحديد أشكال جزيئات كانت مستعصية من قبل، طب النانو لتوصيل الدواء الدقيق والقضاء على الخلايا الورمية. كما يستخدم الدنا في علم الوراثة العرقي وعلم الأنساب الجيني لتحديد تاريخ الشعوب وتحديد هجرات الإنسان العاقل عبر التاريخ.
اكتشاف الدنا وبنيته جاء نتيجة تراكم بحوث ومجهودات عدة علماء حيث استخلصه العالم السويسري فريدريك ميشر أول مرة سنة 1869، ثم اكتشف فيبوس ليفين مكونات النوكليوتيد، ثم اكتشف بعده إروين تشارغاف قواعده أواخر العقد 1940، وقامت روزاليند فرانكلين ببحوث وأخذ صور حيود الأشعة السينية للدنا، ليتمكن بعدها جيمس واتسون وفرنسيس كريك من تتويج مجهودات الجميع باكتشاف بنيته اللولبية.
الخصائص

الدنا عبارة عن بوليمر طويل مكون من وحدات متكررة تسمى نوكليوتيدات،[5][6] بُنية الدنا ديناميكية على طول امتدادها، كونها قادرة على الالتفاف إلى حلقات ضيقة وأشكال أخرى.[7] في كل الأصناف يتكون الدنا من سلسلتين لولبيتين مرتبطتين بعضهما البعض بواسطة روابط هيدروجينية، كلا السلسلتان ملتفتان حول نفس المحور ولهما نفس طول السن الولبي 34 أنغستروم (3.4 نانومتر) والقطر 10 أنغستروم (1 نانومتر).[8] تبعا لدراسة أخرى تم فيها القياس في محلول مختلف، وُجِد أن عرض سلسلة الدنا من 22 إلى 26 أنغستروم (2.2-2.6 نانومتر) وطول نوكليوتيدة واحدة 3.3 أنغستروم (0.33 نانومتر)،[9] رغم أن وحدة النوكليوتيد المتكررة صغيرة جدا إلا أن بوليمرات الدنـا يمكن أن تكون كبيرة جدا وتحوي ملايين أو مئات الملايين من النوكليوتيدات، مثلا: الدنا في أكبر كروموسوم بشري الكروموسوم 1 يتكون مما يقارب 220 مليون زوج قاعدي [10] وسيبلغ طوله 85 ملم لو تمت إطالته.
لا يتواجد الدنا عـادة في الكائنات الحية على شكل جزيء واحد، وإنما زوج من الجزيئات مشدود لبعضه بشدة،[8][11] تلتف هاتين السلسلتين الطويلتين حول بعضهما البعض على شكل لولب مزدوج، يحتوي النوكليوتيد على كل من العمود الفقري للجزيء (الذي يضمن تماسك السلسلة) وقاعدة نووية (التي تترابط مع قاعدة في الشريط الآخر باللولب). القاعدة النووية المرتبطة بسكر تسمى نيوكليوسيد والقاعدة المرتبطة بسكر ومجموعة من الفوسفات أو أكثر تسمى نوكليوتيد. البوليمر المتكون من عدة نوكليوتيدات مترابطة يسمى عديد النوكليوتيد.[12]
يتكون العمود الفقري للدنا من سكر وفوسفات،[13] السكر في الدنا هو ريبوز منقوص الأكسجين وهو بنتوز (سكر يحتوي على خمس ذرات كربون)، ترتبط جزيئات السكر ببعضها بواسطة مجموعات فوسفات والتي تشكل روابط فوسفات ثنائية الأستر بين الذرة الثالثة والخامسة لحلقتي سكر متجاورتين. هذه الروابط اللامتناظرة تعني أن لسلسلة الدنا اتجاه، وفي اللولب المزدوج اتجاه النوكليوتيدات في أحد السلسلتين مضاد للاتجاه في السلسلة الأخرى وهذا يعني أن السلسلتين مضادتا التوازي، نهايتا سلسلتا الدنا اللامتناظرتين يقال بأن لها اتجاهية خمسة فتحة (5') وثلاثة فتحة (3') حيث تحتوي النهاية 5' على مجموعة فوسفات طرفية والنهاية 3' على مجموعة هيدروكسيل طرفية، أحد الفروق الكبيرة بين الدنا والرنا هو السكر ففي الرنا يحل مكان الريبوز منقوص الأكسجين بنتوز وهو الريبوز.[11]
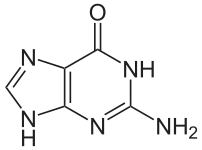
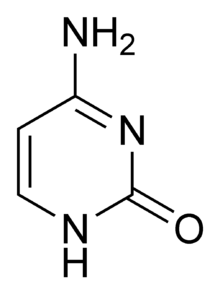
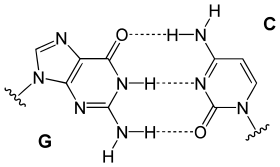
يستقر اللولب المزدوج للدنا أساسا بواسطة قوتين: الروابط الهيدروجينية بين النوكليوتيدات وتفاعلات التراص بين القواعد النووية العطرية.[14]، في الوسط المائي للخلية روابط باي (π) المترافقة لقواعد النوكليوتيدات تتراص عموديا مع محور جزيء الدنا مقللة تفاعلاتها مع غلاف التميّه. القواعد الأربعة المكونة للدنا هي أدينين (A)، سايتوسين (C)، غوانين (G) والثايمين (T)، وترتبط هذه القواعد بـ سكر-فوسفات لتشكل نوكليوتيدات كاملة (الصور بالأسفل)، يرتبط الأدينين مع الثايمين والغوانين مع سايتوسين ويمثلان بالزوج القاعدي A-T والزوج القاعدي G-C.[15][16]
تصنيف وترابط القواعد النووية
تنقسم قواعد الدنا النووية الأساسية إلى قسمين: البيورينات ويندرج ضمنها الأدنين والغوانين وهما جزيئان ثنائيا الحلقة يحويان حلقتين متغايرتين مندمجتين إحدهما سداسية والأخرى خماسية، والبريميدينات ويندرج ضمنها السايتوسين والثايمين وهما جزيئان أحاديا الحلقة عبارة عن حلقة سداسية متغايرة.[11] تتشكل الأزواج القاعدية للولب المزدوج للدنا بترابط بيورين في إحدى السلسلتين مع بيريميدين في السلسلة الأخرى بواسطة اثنتين أو ثلاثة من الروابط الهيدروجينية:
- يرتبط الأدينين مع الثايمين بواسطة رابطتي هيدروجين ليشكل الرابطة A=T.
- يرتبط الغوانين مع السايتوسين بواسطة ثلاث روابط هيدروجينية ليشكل الرابطة G≡C.
يسمى هذا الترابط لقاعدتين نوويتين من سلسلتي اللولب المزدوج بترابطة واتسون-كريك، يوجد نوع آخر من الترابط يسمى ترابط هوغستين يتم فيه ترابط قاعدتي الغوانين والسايتوسين بواسطة رابطتي هيدروجين.[17] كون الروابط الهيدروجينية غير تساهمية يسمح بكسرها وإعادة ربطها بسهولة معتبرة ومنه يمكن فصل سلسلتي اللولب المزدوج للدنا كالسحَّاب سواء بقوة ميكانيكية أو بدرجة حرارة مرتفعة.[18] ونتيجة لهذا التكامل في ترابط القواعد فإن جميع المعلومات التي تحويها إحدى سلسلتي اللولب المزدوج منسوخة في السلسلة الأخرى وهو أمر أساسي في عملية تضاعف الدنا ومختلف وظائفه الأخرى لدى الكائنات الحية.[6]
قواعد غير معيارية
لا يوجد اليوراسيل عادة في الدنا، ولا يظهر سوى كنتيجة تفكك للسايتوسين، لكن في العديد من عاثيات البكتيريا مثل عاثيتي العصويات الرقيقة PBS1 وPBS2 وعاثية اليرسينية piR1-37 تم استبدال الثايمين باليوراسيل،[19] كما تم تحديد عاثية أخرى - عاثية العنقودية S6 بجينوم استُبدِل فيه الثايمين باليوراسيل كذلك.[20]
5-هيدروكسي ميثيل يوريدين منقوص الأكسجين (هم5ي.م) (hm5dU) معروف كذلك بأنه يأخذ مكان الثيميدين في عدة جينومات بما في ذلك عاثيات العصويات: 2C، H1، SP8، ϕe، SPO1 وSP82. كما تم تحديد معدَّل يوراسيل آخر هو: 5-ثنائي هيدروكسي بينتا يوراسيل.[21]
القاعدة ج (D-β-غليكو بيرانوزيل أوكسي ميثيل يوراسيل) هو معدَّل من اليوراسيل يوجد كذلك لدى عدة كائنات: السوطية الديبلونيما واليوغلينا ولدى كل أجناس المحراكات،[22] الاصطناع الحيوي للقاعدة ج يظهر على مرحلتين: الأولى يحول فيها ثيميدين محدد في الدنا إلى 5-هيدروكسي ميثيل يوريدين منقوص الأكسجين وفي الثانية تتم غلكزة الـ(hm5dU) لتشكيل القاعدة ج،[23] وتم تحديد البروتينات التي ترتبط خصيصا بهذه القاعدة،[24][25][26] حيث تظهر لهذه البروتينات علاقة بعيدة بالجين الورمي Tet1 والذي له علاقة بنشوء مرض اللوكيميا النخاعية الحادة،[27] كما يظهر أن القاعدة ج تعمل كإشارة انتهاء لبوليميراز الرنا II.[28][29]
وجد سنة 1976 أن كل قواعد أدينين العاثية S-2L التي تصيب جنس بكتيريا السینكوسیستیس استبدلت بـ 2،6-ثنائي أمين بيورين،[30] ووُجد في 2016 أن الأرشايوزين[31] منقوص الأكسجين يتواجد في جينومات عدة بكتيريا وكذلك عاثية الإشريكية 9g.[32]
تظهر القواعد المعدلة في الدنا كذلك، وأول قاعدة تم التعرف عليها هي 5-ميثيل سايتوسين والتي عثر عليها في جينوم متفطرة سلية سنة 1925،[33] وتمت ملاحظة الاستبدال الكامل للسايتوسين بـ 5-غليكوزيل هيدروكسي ميثيل سايتوسين في عاثيات T المزدوجة ( T4 ،T2 وT6) سنة 1953،[34] وفي جينومات عاثية المستصفرة الرزية Xp12 وفيروس الهالو (FH) تم الاستبدال الكامل للسايتوسين بـ5-ميثيل سايتوسين.[35][36] اكتُشف أن 6N-ميثيل أدينين موجود في الدنا سنة 1955 [37] و6N-كربامويل ميثيل أدينين حُدِّد سنة 1975 [38] و6-O-ميثيل غوانين سنة 1976 [39] و4N-ميثيل سايتوسين سنة 1983،[40] وفي سنة 1985 عثر على 5-هيدروكسي سايتوسين في جينومات عاثيتي المستجذرة RL38JI وN17.[41] يظهر α-بوتريسكينل ثايمين في جينومي كل من عاثية بكتيريا الديلفتيا ΦW-14 وعاثية العصوية SP10.[42] كما وُجِـد α-غلوتاميل ثايميدين في عاثية العصوية SP01 و5- ثنائي هيدروكسي بينتيل يوراسيل في عاثية الطرفية SP15.
سبب تواجد هذه القواعد غير الأساسية أو المعدلة في الدنا غير معروف، ويبدو على الأرجح أن جزءا من السبب على الأقل في وجودها لدى الفيروسات البكتيرية (العاثيات) هو تجنب إنزيمات الاقتطاع الموجودة في البكتيريا، حيث يعمل نظام الإنزيم هذا على الأقل جزئيا كنظام مناعة جزيئي يحمي البكتيريا من الإصابة بالفيروسات.
لا يبدو بأن هذه هي نهاية الحكاية، حيث تم اكتشاف أربع تعديلات على جزيئات السايتوسين في دنا الإنسان.[43] هذه التعديلات هي إضافة مجموعات الميثيل (CH3)-، الهيدروكسي ميثيل (CH2OH)-، الفورميل (CHO) والكاربوكسيل (COOH)، حيث يُعتقد أن لهذه التعديلات وظائف تنظيمية في الدنا.
قائمة بالقواعد غير معيارية الموجودة في الدنا
توجد 17 قاعدة غير معيارية معروفة في الدنا، ومعظمها تعديلات للقواعد الأساسية فضلا عن اليوراسيل وهي:
|
|
|
|
الثلمان
يشكل زوج سلاسل لولبية العمود الفقري للدنا، يمكن إيجاد لولب مزدوج آخر بتتبع الفراغات -أو الأثلام- بين السلسلتين، هذه الفراغات مجاورة لأزواج القواعد ويمكن أن توفر مواقع ارتباط للبروتينات، ولأن السلسلتين غير متموضعتين بانتظام بالنسبة لبعضهما البعض نجد أن الثلمين لهما طولان غير متماثلان، الثلم الكبير عرضه 22 أنغستروم والصغير حوالي 12 أنغستروم.[44] عرض الثلم الكبير يعني أن حواف القواعد فيه أكثر قابلية للوصول إليها من حواف الثلم الصغير وكنتيجة لذلك، البروتينات التي يمكنها الارتباط بتسلسلات معينة في اللولب المزدوج -مثل عوامل النسخ- عادة ما تتصل بجوانب القواعد المكشوفة في الثلم الكبير.[45]
هذه الوضعية تختلف لدى بنيات وهياكل دنا أخرى مختلفة داخل الخلية (انظر أشكال الدنا) لكن الثلمين الكبير والصغير يُذكرَان دائمًا لتوضيح الفروق في الحجم التي يمكن رؤيتها إن تمت إعادة لولب الدنا إلى بنيته ب الطبيعية.
الاتجاه وضد الاتجاه

لإنتاج رنـا وظيفي لا يتم سوى نسخ جزء معين من إحدى سلسلتي الدنا التي تحتوي على الجين وذلك لأن السلسلتين ليستا متماثلتين، ويقال لتسلسل الدنا بأنه "اتجاه" إذا كان مماثلا للتسلسل على نسخة الرنا الرسول التي تُرجمت إلى بروتين [46] ويكون ذلك في السلسلة التي لم يتم نسخها (انظر الشكل)، أما تسلسل السلسلة القالب (التي تم نسخها) فيسمى "ضد الاتجاه" وذلك لأنه مكمل لتسلسل الرنا الرسول. تسمى أحيانا السلسلة التي يتم نسخها "سلسلة التشفير" والمقابلة لها "سلسلة اللاتشفير" إلا أن هذه الدلالة صالحة فقط بالنسبة لجين معين وذلك لأن سلسلتي نفس اللولب المزدوج من الدنا يمكن أن تشفرا بروتينات مختلفة. يمكن أن يتواجد كلا التسلسُلَين "الاتجاه" و"ضد الاتجاه" على أجزاء مختلفة من نفس سلسلة الدنا (أي يمكن لكلا السلسلتين أن تحويا كلا من الاتجاه وضد الاتجاه).
يتم إنتاج تسلسلات رنـا ضد الاتجاه في كل من حقيقيات وبدائيات النوى، إلا أن وظائفها غير واضحة بالكامل،[47] ويشير أحد الاقتراحات إلى أن سلاسل الرنا ضد الاتجاه تساهم في تنظيم التعبير الجيني عبر ترابط قواعد رنا-رنا.[48]
بعض تسلسلات الدنا في بدائيات وحقيقيات النوى وبنسبة أكثر في البلازميدات والفيروسات يتعسر فيها التمييز بين سلسلتي الاتجاه وضد الاتجاه لوجود جينات متداخلة.[49] في هذه الحالات تقوم تسلسلات الدنا بوظيفة مضاعفة، تشفير بروتين معين حين تتم قراءتها على طول إحدى السلسلتين، وبروتين آخر حين تقرأ في الاتجاه المعاكس على طول السلسلة الأخرى. في البكتيريا قد يساهم هذا التداخل في تنظيم نسخ الجينات [50]، أما لدى الفيروسات فيزيد في مقدار المعلومة التي يمكن تشفيرها داخل جينوم فيروسي صغير.[51]
دنا فائق الالتفاف
يكون الدنا خلال معظم عمر الخلية ملفوفا بخفة حول الهستونات ولا يكون مركزا على هيئة كروموسوم، ولا يظهر اللف الفائق للدنا على شكل كروموسومات إلا خلال مرحلة انقسام الخلية.[52] يمكن لف الدنا مثل الحبل خلال عملية تسمى لف الدنا الفائق، وفي حالة الراحة تلتف سلسلة الدنا حول محور اللولب المزدوج مرة كل 10.4 زوج قاعدي، أما إن كان الدنا ملفوفا فإن سلسلتيه تصبحان أشد أو أخف التفافا.[53] حيث إن كان ملفوفا في اتجاه اللولب المزدوج فإن القواعد تصبح متراصة أكثر ويسمى ذلك الالتفاف الفائق الموجب؛ أما إن كان ملفوفا عكس اتجاه اللولب المزدوج فإن القواعد تصبح أقل تراصا ويسمى ذلك بالالتفاف الفائق السالب. لِمعظمِ الدنا في الطبيعة التفافٌ فائق سلبي تُحدِثه إنزيمات التوبوإيزوميراز،[54] هذه الإنزيمات مطلوبة لتزيل التفاف سلسلتي الدنا أثناء عمليتي نسخ وتضاعف الدنا.[55]
الخصائص الفيزيائية والكيميائية للولب المزدوج
كما ذكر بالأعلى، معظم جزيئات الدنا عبارة عن سلسلتي بوليمير مرتبطة مع بعض على شكلٍ لولبيٍّ بواسطة روابط غير تساهمية، هذه البنية مزدوجة السلاسل تستقر بشكل كبير بواسطة تفاعلات القواعد المحزمة مع بعضها في سلسلة واحدة، وهي قوية في تسلسلات G،C. يمكن للسلسلتين أن تنفصلا - في عملية تسمى بالذوبان - لتشكيل جُزَيئَي سلسلتين منفردتين وهو ما يسمى إفساد الدنا. يحدث الذوبان عند درجة حرارة عالية، كميات أملاح منخفضة وأس هيدروجيني مرتفع (الأس الهيدروجيني المنخفض يذيب الدنا، لكن بما أن الدنا غير مستقر بسبب نزع البيورين الحمضي؛ نادرا ما يستخدم الأس الهيدروجيني المنخفض).
لا يعتمد استقرار بنية اللولب المزدوج على محتوى GC فقط (لأن أزواج القواعد CG تحتوي على ثلاث روابط هيدروجينية بينها، والأزوج AT رابطتين) ولكن على التسلسل كذلك (لأن التحزيم له علاقة بالتسلسل) وطوله (كلما كان الجزيء أطول، استقر أكثر). يمكن قياس الاستقرار بطرق عديدة، أشهرها هي "درجة حرارة الذوبان" وهي درجة حرارة تتحول فيها 50% من الجزيئات مزدوجة السلاسل إلى جزيئات منفردة السلاسل، وتعتمد درجة حرارة الذوبان على قوة الأيونات وتركيز الدنا. وكنتيجة يساهم كل من نسبة أزواج القواعد GC وطول اللولب المزدوج للدنا في تحديد قوة ترابط سلسلتي الدنا. لوالب الدنا الطويلة التي لديها محتوى CG كبير تترابط سلاسلها بشكل قوي، في حين أن اللوالب القصيرة التي لديها محتوى AT كبير تمتاز سلاسلها بترابط ضعيف.[56] في علم الأحياء أجزاء لولب الدنا التي يمكن فصلها بسهولة مثل صندوق بربنو ذي التسلسل TATAAT في بعض المحفزات، تميل أن يكون لديها محتوى AT كبير وهو ما يجعل انفصال سلاسلها أكثر سهولة.[57]
يمكن قياس قوة التفاعلات في المختبر بإيجاد درجة الحرارة الضرورية لكسر الروابط الهيدروجينية (التي تسمى كذلك قيمة Tm) والتي يمكن قياسها بتتبع الامتصاص الضوئي لأطوال الموجات 260 نانومتر في محلول يحتوي على الدنا، حيث يزداد هذا الامتصاص عند تفكك أزواج القواعد وهو ما يسمى فرط الكرومية. حين تذوب كل الروابط بين أزواج القواعد في لولب دنا مزدوج، تنفصل السلسلتان وتبقيان في المحلول كجزيئين مستقلين عن بعضهما تماما، جزيئات الدنا منفردة السلاسل هذه لا تكون لها بنية مألوفة، لكن بعض البنيات التي تتخذها أكثر استقرار من الأخرى.[58]
بنيات الدنا

يتواجد الدنا على عدة بنيات منها الدنا أ، الدنا ب والدنا ز، رغم أنه لم يتم سوى ملاحظة بنيتي الدنا أ والدنا ب في كائناتٍ وظيفيةٍ.[13] البنية التي يتخذها الدنا تعتمد على مستوى التميؤ، تسلسل الدنا، كمية واتجاه اللف الفائق، التغيرات الكيميائية للقواعد، نوع وتركيز الآيونات المعدنية ووجود متعددات الأمين في المحلول.[59]
لمعرفة بنية الدنا قام العلماء باستخدام تقنية دراسة البلورات بالأشعة السينية وكانت التقارير المنشورة الأولى حول أنماط حيود الأشعة السينية للدنا أ وب قد استخدمت دراسات مبنية على تحول باتيرسون التي أعطت معلومات محدودة عن هيئة سلال الدنا الموجهة.[60][61] وفي سنة 1953 تم اقتراح دراسات أخرى بواسطة ويلكنز وآل لأنماط حيود الأشعة السينية للدنا ب العالي التميؤ في الكائنات الحية وكانت عبارة عن مربعات دوال بيسل.[62] وفي نفس الصحيفة قدم جيمس واتسون وفرنسيس كريك دراساتهما حول النموذج الجزيئي لأنماط حيود الأشعة السينية للدنا مقترحين أن الهيئة كانت لولبا مزدوجا.[8]
رغم أن الهيئة ب هي الأكثر شيوعا في الظروف الموجودة داخل الخلايا [63] إلا أنها ليست هيئة محددة بدقة وإنما عائلة متقاربة من هيئات الدنا [64] التي تظهر في حالات التميؤ العالية الموجودة داخل الخلايا الحية، وأنماط حيود الأشعة السينية الخاصة بها هي سمة تميز البلورات الناقِصَة (paracrystals) مع درجة معتبرة من عدم الانتظام.[65][66]
مقارنة بالدنا ب فإن الدنا أ له قطر أكبر ويميني الإتجاه كذلك وله ثلم صغير سطحي وعريض وثلم كبير أضيق وأعمق، تظهر هذه الهيئة في الظروف غير الفيسيولوجية في عينات الدنا المجففة جزئيا، أما في الخلايا الحية فيمكن أن يتم إنتاجه بتزواج هجين لسلاسل الدنا والرنا وكذلك في مركبات إنزيم-دنا.[67][68] قِطعُ الدنا التي عُدِلت فيها القواعد كيميائيا بواسطة المثيلة يمكن أن تخضع لتحول كبير في الهيئة وتتخذ البنية ز التي تلتف فيها القواعد على يسار محور اللولب عكس البنية ب الأكثر شيوعا [69] هذا الشكل غير الاعتيادي يمكن التعرف عليه بواسطة بروتينات ترتبط بشكل خاص مع الدنا ز ويمكن أن يكون له دور في تنظيم النسخ.[70]
| خصائص | دنا أ | دنا ب | دنا ز |
|---|---|---|---|
| اتجاه اللولب المزدوج | يمين | يمين | يسار |
| تكرار الوحدة | 1 bp | 1 bp | 2 bp |
| زاوية دوران اللولب المزدوج لكل زوج قاعدي | 32,7° | 34,3° | 60°/2 |
| عدد الأزواج القاعدية لكل دورة للولب المزدوج | 11 | 10,5 | 12 |
| السن اللولبي للولب المزدوج لكل دورة | 2.82 نانومتر | 3.32 نانومتر | 4.56 نانومتر |
| البعد بين القواعد داخل اللولب المزدوج | 0.26 نانومتر | 0.34 نانومتر | 0.37 نانومتر |
| القطر | 2.3 نانومتر | 2.0 نانومتر | 1.8 نانومتر |
| انحناء الأزواج القاعدية بالنسبة لمحور اللولب المزدوج | +19° | −1,2° | −9° |
| متوسط الالتواء (propeller twist) | +18° | +16° | 0° |
| اتجاه القواعد بالنسبة للسكر | عكس | عكس | بيريميدين : عكس بيورين : مع |
| انحناء روابط ذرات السكر (Sugar pucker) | 'C3-إندو | 'C2 - إندو | سايتوسين : 'C2 - إندو غوانين : 'C2 - إكسو |
أشكال خاصة

- موصل هوليداي: يتم تكوين موصل هوليداي خلال التأشيب المتماثل بين جزيئي دنا يحملان نفس المعلومة الوراثية (صبغيات متماثلة، كروماتيدات شقيقة)، لدى هذا الموصل هيئة على شكل صليب مع تسلسلات متماثلة تسمح له بالتحرك نحو اتجاه واحد أو الآخر. يتم إنشاؤه بواسطة مركب إنزيمي يسمى ريزولفاز (رتإ 3.1.22.4) ويمكن أن يقود إلى تعابر بين الجزيئين ينتج عنه تبادلٌ للمعلومات الوراثية.[75]
- دنا دبوس الشعر: يمكن لتسلسلات الدنا المتناظرة الالتفاف وتشكيل بنية تسمى الحلقة الجذعية،[76] بعض التسلسلات وخاصة التسلسلات ثلاثية النوكليوتيد (CAG)n أو (CTG)n يمكن أن تشكل دبابيس شعر غير تامة تكون فيها جزيئات الغوانين والسايتوسين مترابطة وجزيئات الأدينين والثايمين غير مترابطة.[77]
- الدنا الثلاثي: هي هيئة دنا تقوم فيها ثلاث جزيئات قليل النوكليوتيد بالالتفاف حول بعضها لتشكيل لولب ثلاثي، في هذه البنية تقوم تقوم سلسة دنا منفردة بالارتباط بلولب دنا مزدوج من البنية ب بواسطة الروابط الهيدروجينية العادية أو المعكوسة لهوغستين، ويمكن أن تلعب هذه الهيئة دورا في التنظيم الوظيفي للتعبير الجيني عن طريق تعديل نسخها لجزيئات رنا.
- الدنا الرباعي: تحتوي نهاية الصبغي الخطي على مناطق خاصة تدعى قسيمات طرفية وظيفتها السماح للخلية بمضاعفة نهايات الصبغي باستخدام إنزيم تيلوميراز وذلك لأن الإنزيمات التي تُضاعِف الدنا عادة لا يمكنها نسخ النهايات 3' للصبغيات،[78] كما تساعد هذه المناطق في حماية نهايات الدنا وإيقاف أنظمة الترميم في الخلية من اعتبارها تلفا يجب إصلاحه.[79] في الخلايا البشرية القسيمات الطرفية عبارة سلسلة منفردة من الدنا تحوي الآلاف من الوحدات النوكليوتدية البسيطة TTAGGG المتكررة.[80]

الثالثة بالأصفر.
هذه التسلسلات الغنية بالغوانين يمكن أن تجعل نهايات الصبغي مستقرة بتشكيل هياكل مرصوصة بوحدات رباعية القواعد بدل ثنائية القواعد المعتادة الموجودة في هياكل الدنا الأخرى، حيث تقوم أربع قواعد غوانين بتشكيل وحدة رباعية مسطحة وبعدها تتراص هذه الوحدات الرباعية فوق بعضها لتشكيل بنية رباعي الصبغيد-غ مستقرة.[81] تستقر هذه الهياكل الرباعية بواسطة روابط هيدروجينية بين نهايات هذه القواعد وتمخلب أيون معدني في مركز كل وحدة رباعية.[82]
 |
 |
| تفرع واحد | تفرع متعدد |
بالإضافة إلى هذه الهياكل المرصوصة، تشكل القسيمات الطرفية كذلك هياكل تسمى حلقات القسيم الطرفي أو حلقات ت تقوم فيها سلسلة دنا منفردة بالالتواء في حلقة طويلة تستقر بواسطة إنزيمات ترتبط بالقسيمات الطرفية.[83] في نهاية الحلقة ت يتم ضم السلسلة المنفردة للقسيم الطرفي إلى منطقة لدنًا مضاعفِ السلسلة وهذا يتسبب في إخلال التزاوج القاعدي لإحدى سلسلتي هذا اللولب المزدوج وتشكيل بنية ثلاثية السلاسل تسمى حلقة الإزاحة أو الحلقة د.[81]
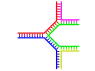
- الدنا المتفرع: يحدث انسلال الدنا في إحدى نهايتي اللولب المزدوج حين تتوقف تسلسلات سلسلتَيْهِ عن كونها متكاملة حيث تفترق السلسلتان عن بعضهما وتتخذان هيئة Y، ويمكن للدنا أن يتشعب إن وُجدت سلسلة دنا ثالثة تحوي تسلسلا مكملا لكلا تسلسلي السلسلتين المفترقتين، فتتشكل هيئة مزدوجة متكاملة على شكل Y. مع أن أبسط أمثلة الدنا المتفرع تحوي ثلاث سلاسل دنا فقط فإن تواجد مركبات متفرعة تحوي سلاسل إضافية وعدة تفرعات أخرى ممكن كذلك.[84] ويتم استخدام الدنا المتفرع في تقنية النانو للدنا لإنشاء أشكال هندسية.
تركيب كيميائي بديل للدنا
لأعوام عديدة اقترح علماءٌ في علم الأحياء الفلكي وجود محيط حيوي خفي، وهو محيط حيوي بكتيري افتراضي للأرض يستخدم عمليات كيمياء حيوية وجزيئية مختلفة جذريا عما هو معروف في الحياة حاليا. أحد المقترحات هو وجود أشكال حياة تستخدم الزرنيخ بدل الفسفور في الدنا. وتم نشر تقرير سنة 2010 حول إمكانية ذلك لدى البكتيريا الزرنيخية (GFAJ-1)،[85][85][86] إلا أن البحوث اختُلف حولها،[86][87] وأظهرت أدلةٌ أن البكتيريا تمنع بشكل نشط إدماج واستخدام الزرنيخ في العمود الفقري للدنا الخاص بها والجزيئات الحيوية الأخرى.[88]
التغيرات الكيميائية وتحزيم الدنا المعدل
تغير القواعد وتحزيم الدنا
 |
 |
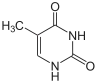 |
| سايتوسين | 5-ميثيل سايتوسين | ثايمين |
يتأثر التعبير الجيني بطريقة تحزيم (بالإنجليزية: Packaging) الدنا في الكروموسومات على هيئة تسمى كروماتين، يمكن لبعض القواعد المعدلة أن تكون لها علاقة بتكوين الكروماتينات في المناطق ضعيفة أو عديمة التعبير الجيني والغنية بقواعد السايتوسين عن طريق المثيلة خاصة في المناطق CpG، يمكن للهستونات التي يلتف الدنا حولها في الكروماتين أن تتغير بطريقة تساهمية ويمكن للكروماتين بحد ذاته أن يتغير بواسطة مركبات تغيير بنية الكروماتين، علاوة على ذلك فإن مثيلة الدنا والتعديل التساهمي للهستونات يساهمان معا في التأثير على الكروماتين والتعبير الجيني.[89]
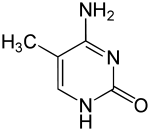
على سبيل المثال تُنتج مثيلة السايتوسين 5-ميثيل سايتوسين وهو ضروري في تعطيل الصبغي X،[90] يختلف متوسط معدلات المثيلة بين الكائنات فالدودة الربداء الرشيقة تعوز مثيلة السايتوسين في حين أن الفقاريات لديها معدل مرتفع يصل حتى 1% من الدنا الخاص بها يحوي 5-ميثيل سايتوسين،[91] رغم أهمية الـ5-ميثيل سايتوسين إلا أنه قد يتعرض إلى نزع أمين ويصبح ثايمين وكذلك السايتوسين الذي يصبح يوراسيل، قواعد السايتوسين المُمَثْيَلة عرضة للطفرات.[92] من التغييرات الأخرى للقواعد مثيلة الأدنين عند البكتيريا ووجود 5-هيدروكسي ميثيل سايتوسين في الدماغ.[93] وغلكزة اليوراسيل لإنتاج القاعدة ج لدى ذوات منشأ الحركة.[94][95] التي تعمل كإشارة إنهاء النسخ للأنزيم بوليميراز الرنا 2.[96][97]
تضرر
يمكن للدنا التضرر بأنواع مختلفة من المطفِّرات التي تغيّر تسلسله، تتضمن هذه المطفرات: المؤكسدات، عوامل الألكلة والإشعاعات الكهرومغناطيسية عالية الطاقة مثل الأشعة فوق البنفسجية والأشعة السينية. يتعلق نوع الضرر الحاصل بنوع المطفّر فعلى سبيل المثال يمكن للأشعة فوق البنفسجية الإضرار بالدنا بإنتاج مثنويات الثايمين وهي روابط جانبية بين قواعد البيريميدين.[99] من جهة أخرى تسبب المؤكسدات مثل الجذور الكيميائية الحرة أو بيروكسيد الهيدروجين أنواع مختلفة من الأضرار منها تغيير القواعد -خاصة الغوانوزين- وشروخ في السلسلة المزدوجة للدنا،[100] حيث يبلغ متوسط عدد القواعد التي عانت أضرارا من المؤكسدات في خلية نموذجية حوالي 150 ألف قاعدة.[101] أخطر هذه الآفات التأكسدية هي شروخ السلاسل المزدوجة وذلك لأنها صعبة الإصلاح وقد تُنتج طفرات نقطية، غرز، أو حذف في تسلسللات الدنا وكذلك انتقالات صبغية.[102] يمكن لهذه الطفرات أن تسبب السرطان بسبب آليات إصلاح الدنا المحدودة طبيعيا، لذا إن عاش البشر لوقت طويل بما فيه الكفاية فإن جميعهم في النهاية سيصاب بالسرطان.[103][104]
تضررات الدنا التي تحدث طبيعيا بسبب النشاطات الطبيعية للخلية التي تنتج كائنات متفاعلة مع الأكسجين، نشاطات الحلمأة لماء الخلية تظهر بشكل متواتر كذلك رغم أن معظم هذه الأضرار يتم إصلاحها. في أي خلية تبقى بعض تضررات الدنا رغم عمليات الإصلاح وتتراكم هذه التضرارت مع مرور الزمن في الخلايا تالية التفتل لأنسجة الثدييات، هذا التراكم يبدو عاملا أساسيا مهما في الشيخوخة.[105][106][107]
تدخل العديد من المطفِّرات في الفراغ الموجود بين زوجين قاعديين متجاورين فيما يعرف بـ الإقحام ومعظم المقتحمات جزيئات عطرية ومستوية ومن الأمثلة: بروميد الإيثيديوم، الأكريدينات وداونوروبيسين، لكي يدخل المقتحم بين القواعد يجب عليه فصلها وهذا يشوه سلسلتي الدنا بسبب فك التواء اللولب المزدوج، هذا التشوه يمنع كلا من نسخ وتضاعف الدنا ويسبب تسمما وطفرات.[108] وكنتيجة لذلك يمكن لمقتحمات الدنا أن تكون مسرطنات وفي حالة الثاليدوميد، ماسخات،[109] مقتحمات أخرى مثل بنزو(a)بيرين ديول إيبوكسيد وأفلاتوكسين يشكلان نواتج إضافة تُحدث أخطاءً في التضاعف.[110] مع ذلك وبسبب قدرتها على تعطيل ومنع نسخ وتضاعف الدنا؛ تستخدم مقتحمات سامة أخرى مشابهة في العلاج الكيميائي لمنع النمو السريع للخلايا السرطانية.[111]
وظائف بيولوجية

عادة ما يكون الدنا على شكل صبغيات خطية عند حقيقيات النوى وصبغيات دائرية عند بدائيات النوى ومجموع الصبغيات في خلية واحدة يشكل الجينوم، يحتوي جينوم الإنسان على ما يقارب 3 مليارات زوج قاعدي منتظمة في 46 صبغيا.[112] المعلومات المحمولة في الدنا موجودة في تسلسلات قطع منه تسمى الجينات، يتم تناقل المعلومة الوراثية في الجينات عبر ترابط قاعدي متكامل مثلا: حين تستخدم الخلية المعلومة الموجودة في الجين يتم نسخ تسلسل الدنا إلى تسلسل رنا مكمل عبر التجاذب بين الدنا والنوكليوتيدات الصحيحة للرنا، وتستخدم هذه النسخة -عادة- لاصطناع تسلسل بروتين مماثل عبر عملية تسمى الترجمة والتي تعتمد على نفس التجاذب بين نوكليوتيدات الرنا، ومن جهة أخرى يمكن للخلية نسخ معلوماتها الوراثية خلال عملية تسمى تضاعف الدنا، تفاصيل هذه العمليات ستذكر في العناوين التالية وسيكون التركيز في العوان التالي على التفاعلات بين الدنا والجزيئات الأخرى التي تتدخل في وظائف الجينوم.
الجينات والجينومات

يُحزم جينوم الدنا بشدة وانتظام خلال عملية تسمى تكثيف الدنا ليسع الأحجام الصغيرة المتوفرة في الخلية، يتواجد الدنا عند حقيقيات النوى في النواة مع كميات صغيرة في الميتوكندريون والبلاستيدات الخضراء أما عند بدائيات النوى فإن الدنا مخزن في جسيم غير منتظم الشكل يسمى نوواني.[113] المعلومات الجينية في الجينوم موجودة داخل الجينات ومجموع هذه المعلومات في كان حي يسمى نمط جيني، والجين هو وحدة وراثة وهو عبارة عن منطقة من الدنا تؤثر في صفة معينة لكائن ما، تحتوي الجينات قالب قراءة مفتوح يمكن نسخه بالإضافة إلى تسلسلات منظم مثل المحفزات والمعززات والتي تتحكم في عملية نسخ قالب القراءة المفتوح.
في العديد من الأجناس لا يٌشفِّر البروتينَ سوى كمية صغيرة من المجموع الكلي للجينوم، على سبيل المثال عند البشر 1.5% من الجينوم البشري عبارة عن إكسونات تشفر بروتينات، مع كون 50% من الدنا البشري عبارة عن تسلاسلات متكررة غير مُشفِّرة.[114] يمثل سبب وجود كم كبير من الدنا غير المشفر في جينوم حقيقيات النوى والفروق الكبيرة في حجم الجينوم -القيمة س- بين الأجناس لغزا طويل الأمد يعرف بمعضلة القيمة س.[115] رغم ذلك بعض تسلسلات الدنا غير المشفرة يمكن أن تشفر رنا غير مشفر وظيفيا مهمته ضبط التعبير الجيني.[116]
بعض تسلسلات الدنا غير المشفرة تلعب أدوارا بنيوية في الصبغيات، تحتوي القسيمات الطرفية والقطع المركزية على بعض الجينات لكنها مهمة لوظيفة واستقرار الصبغيات.[79][117] الجينات الكاذبة نوع وافر في الجينوم البشري من الدنا غير المشفر وهي نسخ من جينات تم تعطيلها بواسطة الطفرات،[118] هذه الجينات الكاذبة عبارة عن مستحاثات جزيئية رغم أنها تلعب أحيانا دور المادة الجينية الخام لإنشاء جينات جديدة عبر عملية التضاعف الجيني وعملية التطور التباعدي.[119]
تضاعف الدنا
الانقسام الخلوي عملية أساسية لنمو الكائن، وحين تتم هذه العملية فعلى الخلية مضاعفة الدنا في جينومها لكي تحصل الخليتيان البنت على نفس المعلومات المتواجدة في الخلية الأم، توفر هيئة الدنا اللولبية المزدوجة طريقة بسيطة لتضاعف الدنا. أثناء عملية التضاعف تقوم إنزيمات التوبوإيزوميراز بفك التواء السلسلة المضاعفة ثم تقوم إنزيمات الهيليكاز بحل الروابط الهيدروجينية بين النوكليوتيدات وفصل السلسلتين عن بعضهما وبعدها يتم إنتاج سلسلتين متكاملتين بواسطة إنزيم بوليميراز الدنا، تقوم هذه الإنزيمات بإنتاج سلسلة مكملة بإيجاد القاعدة الصحيحة عبر ترابط القواعد المكمل ثم ربطها بالسلسلة الأصلية، ولأن إنزيمات بوليمراز الدنا لا تُنتج السلاسل سوى في الاتجاه 5' إلى 3' يتم استخدام طريقة أخرى لنسخ السلاسل المقابلة في لولب الدنا المزدوج.[120] بهذه الطريقة القاعدة في السلسلة القديمة تحدد القاعدة التي ستظهر في السلسلة الجديدة ويغدو لدى الخلية نسخة جديدة من دناها.

التعبير الجيني
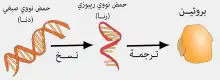
التعبير الجيني هو تحول النمط الجيني لكائن ما إلى نمط ظاهري؛ أي إلى مجموعة الخصائص المميزة للكائن، تتأثر هذه العملية بالعديد من المحفزات الخارجية المختلفة وتحتوي ثلاث مراحل أساسية كبيرة هي:
- نسخ الدنا إلى رنا وهو حمض نووي مختلف يملك وظيفة بيولوجية معينة على حسب نوعه (ناقل، ريبوسومي..إلخ) أو الرنا الرسول الذي يعمل كوسيط وحامل المعلومة لاصطناع البروتين.[121]
- ترجمة الرنا الرسول إلـى بروتين.
- نشاط جزيئات الرنا غير المشفرة والبروتينات داخل الكائنات.
مع ملاحظة أنه يمكن لنفس الدنا الخاص بكائن ذو مرحلتي تطور أن يُعبر عن نفسه بشكل غير متشابه تماما (بسبب مثبطات ومحفزات مختلفة)، أفضل مثال معروف عن ذلك هو اليسروع والفراشة حيث أنهما شكليا مختلفان كثيرا.

النسخ والترجمة
النسخ عملية بيولوجية يتم فيها نسخ المعلومة الوراثية المشفَّرة في جين معين من تسلسلات نوكليوتيدات الدنا الموافقة له وإنتاج جزيئات رنا مختلفة مشفِّرة وغير مشفِّرة بواسطة إنزيمات بوليميراز الرنا، سلسلة هذه الجزيئات مماثلة في تركيبها لسلسلة الدنا باستثناء احتواء وحدتها الأساسية على ريبوز بدل الريبوز منقوص الأكسجين وقاعدة اليوراسيل مكان الثايمين، تخضع جزيئات الرنا الأولية الناتجة والتي تسمى "الرنا قبل الرسول" إلى تعديلات ما بعد الترجمة من تقبيع وتذييل بعديد الأدينيلات لإنتاج رنا رسول مستقر وناضج قادرٍ على تحمل عملية الترجمة أثناء تخليق البروتين.[122] العلاقة بين تسلسل نوكليوتيدات الجين والحمض الأميني محددة بقواعد الترجمة المعروفة بالشيفرة الجينية، تتكون الشيفرة الجينية من كلمات ذات ثلاث أحرف تسمى رامزات أو كودونات مكونة من تسلسل حروف ثلاث نوكليوتيدات (مثال ACT، CAG ،TTT).[123]
الترجمة هي عملية تحويل المعلومة الوراثية المحمولة في جزيء الرنا الرسول الناضج المغادر للنواة إلى بروتين (أوفي بعض الأحيان عدة بروتينات)، حيث يتم فك شيفرته بواسطة الريبوسوم الذي يقرأ تسلسلات الرنا وذلك بربط تسلسلات قواعد الرنا الرسول مع قواعد ضد الكودون للرنا الناقل الذي يحمل معه الأحماض الآمينية،[124] وبما أنه توجد أربع قواعد؛ في توليفة ثلاثة حروف يكون هنالك 64 كودونا ممكنا (43 توليفة) هذه التوليفات تشفِّر العشرين حمض أميني الأساسية، مانحة معظم الأحماض الأمينية أكثر من كودون واحد ممكن، كما توجد ثلاث رامزات 'توقف' أو 'بلا معنى' تشير إلى نهاية المنطقة المشفرة وهذه الرامزات هي TAA، TGA وTAG.
ترميم الدنا

ترميم الدنا هو مجموعة عمليات تتعرف بها الخلية على أضرار في جزيئات الدنا وتصححها، في الخلايا البشرية يسبب كل من نشاط الأيض وعوامل طبيعية مثل الإشعاعات أضرارا للدنا تقدر بحوالي 1 مليون آفة جزيئية لكل خلية كل يوم.[125] حيث تسبب العديد من هذه الآفات ضررا هيكليا لجزيء الدنا يمكن أن يغير أو يقضي على قدرة الخلية على ترجمة جيناتها المتضررة، بعض الآفات الأخرى تحدث طفرات محتملة الضرر في جينوم الخلية وهو ما يؤثر على قدرة بقاء الخلايا البنت بعد عملية الانقسام المتساوي، ونتيجة لهذا فإن نظام ترميم الدنا نشط باستمرار لترميم الضرر في بنية الدنا، حين يفشل نظام الترميم ولا يحدث نظام الاستماتة الخلوي فإنه يحدث ضرر في الدنا غير قابل للترميم بما في ذلك شروخات في السلسلة المزدوجة، والارتباط التشابكي للدنا (ترابط شبكي بين السلاسل)،[126][127] يمكن أن يقود هذا إلى أورام خبيثة أو إلى السرطان حسب فرضية ندسون.
معدل سرعة ترميم الدنا يعتمد على عدة عوامل منها: عمر الخلية، نوعهـا، ومحيطها الخارجي. الخلية التي تراكمت فيها كمية كبيرة من أضرار الدنا والخلية التي لا يمكنها ترميم أضرار الدنا الخاص بها بفعالية يمكن أن ينتهي بها الحال إلى أحد ثلاث إمكانيات:
- حالة متعذرة العكس من السكون تعرف بالهرم.
- انتحار للخلية يعرف بالاستماتة أو موت الخلية المبرمج.
- انقسام خلوي غير منتظم يقود إلى تشكيل ورم سرطاني.
قدرة الخلية على ترميم الدنا أساسية لسلامة جينوميها ومنه قيام الكائن بالوظائف بشكل طبيعي، العديد من الجينات التي ظهر في البداية أنها تؤثر على متوسط العمر اتضح أن لها علاقة بحماية الدنا وترميم أضراره.[128]، يوجد ثلاث أنواع من الترميم: استئصال النوكليوتيد، استئصال القاعدة وترميم الدنا غير المتطابق.
الأحماض النووية خارج الخلية
يصدر معظم الحمض النووي الصبغي الخارجي المجرد (eDNA) بموت الخلية، وهو واسع الانتشار في الطبيعة حيث يبلغ تركيزه في التربة حوالي 2 ميكروغرام/ل وتركيزه في الأوساط المائية الطبيعية قد يبلغ 88 ميكروغرام/ل.[129] تم اقتراح العديد من الوظائف للدنـا الخارجي: فقد تكون له علاقة في نقل الجينات الأفقي؛[130] قد يوفر مغذيات؛[131] وقد يعمل كمنظم لجذب أو معايرة الأيونات أو المضادات الحيوية.[132] يعمل الدنا الخارجي كمكون -عبارة عن مصفوفة وظيفية خارجية- في بيـوفيلم العديد من الأجناس البكتيرية، كما يمكن أن يعمل كعامل تعرّف لتنظيم ارتباط وتشتيت أنواع خلايا محددة في البيوفيلم،[133] كما يمكن أن يساهم في تكون البيوفيلم،[134]، ويمكن أن يساهم في القوة الفيزيائية للبيوفيلم ومقاومته للضغظ البيولوجي.[135]
يوجد الدنا الجنيني من دون خلايا في دم الأم، ويمكن فحص تسلسله لمعرفة معلومات كثيرة حول تطور الجنين.[136]
تفاعلات مع البروتينات
جميع وظائف الدنا تعتمد على التفاعلات مع البروتينات، قد تكون هذه التفاعلات غير محددة أو يمكن للبروتين الارتباط بشكل محدد على تسلسل معين للدنا، يمكن للإنزيمات الارتباط بالدنـا كذلك ومن بين هذه الإنزيمات البوليميرايزات التي تنسخ تسلسل قواعد الدنا أثناء عمليتي النسخ والتضاعف والتي تعتبر ذات أهمية كبيرة.
بروتينات مرتبطة بالدنا
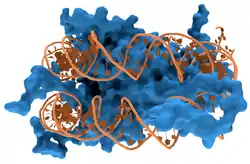
البروتينات البنيوية التي ترتبط بالدنا هي أمثلة مفهومة جدا عن التفاعلات غير المحدَّدَة للدنا مع البروتين. يوضع الدنا داخل الكروموسومات في مركبات مع بروتينات بنيوية، تنظم هذه البروتينات الدنا على شكل هيئة متراصة تدعى كروماتين، لدى حقيقيات النوى يتطلب حدوث هذه الهياكل ترابط الدنا مع بروتينات صغيرة تسمى الهستونات، أما عند بدائيات النوى فيتطلب ذلك أنواعا عديدة من البروتينات.[137][138] تشكل الهستونات مركب على شكل قرص يسمى جسيم نووي يحوي دورتين كاملتين لدنا مزدوج السلاسل ملفوف على سطحه، تشكلت هذه التفاعلات غير المحددة بسبب قيام الوحدات الأساسية في الهستونات (أحماض أمينية) بتشكيل روابط أيونية مع مجموعات الفوسفات الحمضية في الوحدة الأساسية للدنا ومنه لا تعتمد هذه التفاعلات بشكل كبير على تسلسل القواعد.[139] التغيرات الكيميائية لهذه الأحماض الآمينية تشمل المثيلة، الفسفرة والأستلة.[140] تُغيِّر هذه التغيرات الكيميائية قوة التفاعل بين الدنا والهستونات، جاعلة الدنا أكثر أو أقل قابلية لوصول عوامل النسخ إليه وتغير معدل سرعة النسخ.[141] بروتينات الترابط غير المحدد بالدنا الأخرى في الكروماتين تشمل مجموعة البروتين عالية الحركة التي ترتبط بالدنا لتحنِيَه أو تلويه.[142] وهذه البروتينات ضرورية لطي مصفوفات من الجسيمات النووية وترتيبها في هيئة أكبر تشكل الكروموسومات.[143]
توجد مجموعة مميزة من البروتينات المرتبطة بالدنا ترتبط بشكل محدد في سلسلة واحدة من الدنا، لدى البشر بروتين التضاعف A هو أفضل عنصر مفهوم من هذه العائلة ويستخدم في عمليات يكون فيها اللولب المزدوج مفصولا، بما في ذلك تضاعف الدنا، إعادة التركيب وترميم الدنا،[145] يبدو أن هذه البروتينات تعمل على استقرار سلسلة الدنا المفردة ومنعها من تشكيل حلقة جذعية أو تحللها بواسطة النوكليازات.
في المقابل تطورت بروتينات أخرى لترتبط بتسلسل دنا محدد، أكثر أنواع هذه البروتينات دراسة هي عوامل النسخ المختلفة، وهي بروتينات تنظم عملية النسخ حيث يرتبط كل عامل نسخ بتسلسل دنا واحد مخصص لتفعيل أو تثبيط نسخ الجينات التي يتواجد تسلسلها بالقرب من محفزاتها. تُنجز عوامل النسخ ذلك بطريقتين، أولا يمكنها الارتباط ببوليميراز الرنا المسؤول عن عملية النسخ سواء مباشرة أو عبر بروتينات وسيطة أخرى، هذا يجعل البوليميراز يتواجد على المحفز ويسمح ببدء النسخ.[146] ثانيا يمكن لعوامل النسخ الارتباط بالإنزيمات المعدلة للهستونات في المحفز، وهذا يغير قابلية الوصول لسلسة الدنا القالب بالنسبة للبوليميراز.[147]
لأن مواقع الارتباط بالدنا هذه يمكن أن تظهر في مختلف مناطق جينوم الكائن؛ فإن تغيرات في نشاط أحد أنواع عوامل النسخ يمكن أن يؤثر على الآلاف من الجينات.[148] ونتيجة لذلك فإن هذه البروتينات عادة أهدافٌ لتوصيل الإشارة التي تتحكم بالردود اتجاه التغيرات المحيطية أو التمايز الخلوي والنمو. اختصاص تفاعلات عوامل النسخ هذه مع الدنا يأتي من قيام هذه البروتينات باتصالات عديدة مع حواف قواعد الدنا، وهذا يسمح لها بقراءة تسلسل الدنا. تتم معظم تفاعلات القواعد هذه في الثلم الكبير أين يكون الوصول لمعظم القواعد ممكنا.[45]
النوكلياز والليغاز

النوكليازات هي إنزيمات تقوم بقص سلاسل الدنا بتحفيز حلمـأة روابط الفوسفات ثنائية الأستر. تسمى النولكيازات التي تقوم بحلمأة النوكليوتيدات عند نهايات سلسلتي الدنا نوكليازات خارجية وتسمى التي تقوم بالقص داخل السلاسل نوكليازات داخلية، أكثر إنزيمات النوكلياز استخدما في علم الأحياء الجزيئي هي إنزيمات التقييد الداخلية التي تقطع الدنا في تسلسلات محددة، على سبيل المثال: إنزيم إيكور5 الموضح في الصورة يمينا يتعرف على على التسلسل السداسي 5′-GATATC-3′ ويقوم بالقطع على الخط الأفقي. في الطبيعة تحمي هذه البروتينات البكتيريا من عدوى العاثيات وذلك بهضم دنا العاثية لدى دخوله خلية البكتيريا، متصرفة كجزء من نظام منع التغييرات،[150] وفي التكنولوجيا تستخدم هذه الإنزيمات المختصة بتسلسلات معية في التنسيل الجزيئي والبصمة الوراثية.
يمكن لإنزيمات تسمى ليغازات الدنا إعادة وصل سلاسل الدنا المقطوعة أو المبتورة،[151] وهي مهمة بشكل خاص للسلسة المتأخرة في عملية تضاعف الدنا حيث تقوم بتجميع قطع أوكازاكي القصيرة في شوكة التضاعف إلى نسخة كاملة من السلسلة الدنا القالب، كما تستخدم في ترميم الدنا وإعادة التركيب الجيني.[151]
توبوإيزوميراز والهيليكاز
التوبوإيزوميرازات هي إنزيمات تجمع نشاط كل من النوكلياز والليغاز، حيث تغير هذه البروتينات في كمية لف الدنا الفائق، تعمل بعض هذه الإنزيمات على قطع لولب الدنا والسماح لأحد مقاطعه بالدوران ومنه التخفيف في مستوى اللف الفائق وبعدها يقوم الإنزيم بإعادة وصل القطع،[54] أنواع أخرى من هذه الإنزيمات يمكنها قطع إحدى سلسلتي لولب الدنا ثم تمرير السلسلة الأخرى عبر ذلك القطع قبل إعادة جمع اللولب.[152] وهذه الإنزيمات مطلوبة في العديد من العمليات المتعلقة بالدنا مثل تضاعف الدنا والنسخ.[55]
الهيليكازات هي بروتينات تعتبر كنوع من المحركات الجزيئية، حيث تستخدم الطاقة الكيميائية في النوكليوسيدات ثلاثية الفوسفات وفي الغالب أدينوسين ثلاثي الفوسفات لكسر الروابط الهيدروجينية بين القواعد وفك لولب الدنا المزدوج لسلسلتين مفردتين.[153] هذه الإنزيمات ضرورية في معظم العمليات التي تتطلب وصول الإنزيمات إلى قواعد الدنـا.

بوليميراز
البوليميرازات هي إنزيمات تقوم بتخليق سلاسل عديدة النوكليوتيد من نوكليوسيدات ثلاثية الفوسفات، تسلسل نواتجها يخلَّق اعتمادا على سلاسل عديدة النوكليتويد موجودة مسبقا تسمى السلاسل القالب، تعمل هذه الإنزيمات بإضافة نوكليوتيد بشكل مستمر لمجموعة هيدروكسيل النهاية 3' لسلسلة عديد نوكليوتيد متنامية، وكنتيجة لذلك تعمل البوليميرازات في الاتجاه 5' إلى 3'.[154] في الموقع النشط لهذه الإنزيمات تترابط قاعديا النوكليوسيدات ثلاثية الفوسفات القادمة مع السلسة القالب: وهذا يسمح للبوليميرازات بتخليق سلسلة مكملة لسلسلة القالب بشكل دقيق. تصنف البوليميرازات حسب نوع السلسلة القالب التي يستخدمونها.
في عملية تضاعف الدنا تقوم بوليمرازات الدنا -المتعلقة بالدنا- بعمل نسخ لسلاسل الدنا عديدة النوكليوتيد. للحفاظ على المعلومة البيولوجية؛ أمر جوهري أن يتم نسخ تسلسل القواعد في كل نسخة بشكل دقيق ومكمل لقواعد السلسة القالب. لدى العديد من بوليميرازات الدنا نشاط التصحيح حيث تتعرف على الأخطاء التي تقع أحيانا في تفاعلات التخليق بواسطة عدم وجود ترابط قاعدي بين النوكليوتيدات غير المتكاملة. إن تم اكتشاف عدم تكامل يُنَشّط إنزيم نوكلياز خارجي 3′ إلى 5′ وتتم إزالة القاعدة.[155] يَنشُط البوليميراز لدى معظم الكائنات في مركبات كبيرة تسمى ريبليسومات والتي تحتوي على العديد من المركبات الإنزيمية الفرعية مثل رابط الدنا والهيليكاز.[156]
بوليمرازات الدنا -المتعلقة بالرنا- هي قسم خاص من البوليمرازات التي تقوم بنسخ تسلسل سلسلة رنا في الدنا، حيث تشمل إنزيم النسخ العكسي وهو إنزيم شائع له علاقة بإصابة الخلايا بالفيروسات الراجعة، إنزيم التيلوميراز الضروري لتضاعف القسيمات الطرفية.[78][157] على سبيل المثال إنزيم النسخ العكسي لفيروس العوز المناعي البشري هو إنزيم لتضاعف فيروس الإيدز.[157] التيلوميراز هو بوليميراز غير اعتيادي لأنه يحوي سلسلة الرنا القالب خاصته كجزء من بنيته، ويقوم بتصنيع القسيمات الطرفية في نهايات الصبغيات حيث تمنع القسيمات الطرفية نهايات الصبغيات من الاندماج مع نهايات الصبغيات المجاورية وتحميها من التضرر كذلك.[79]
تتم عملية النسخ بواسطة بوليمرازات الرنا -المتعلقة بالدنا- حيث تقوم بنسخ تسلسل سلسلة دنا إلى رنا. للبدء في عملية نسخ جين يرتبط بوليميراز الرنا بتسلسل دنا محدد يسمى المحفز وبعدها يقوم بنسخ تسلسل الجين لإنتاج رنا رسول حتى يصل منطقة تسمى المنهي حيث يتوقف وينفصل عن الدنا. كما هو الحال بالنسبة لبوليميرازات الدنا المتعلقة بالدنا؛ يعمل إنزيم بوليميراز الرنا 2 الذي ينسخ معظم الجينات في الجينوم البشري في مركبات إنزيمية كبيرة تحوي العديد من الوحدات الثانوية التنظيمية والملحقة.[158]
تطور المعلومة الوراثية
طفرات

الطفرة هي تغير دائم في تسلسل نوكليوتيدات جينوم كائن حي، فيروس، دنا غير صبغي أو أي عناصر جينية أخرى، وتنتج الطفرات من أخطاء أثناء عملية تضاعف الدنا أو أي أنواع أخرى من تضررات الدنا مثل التعرض للأشعة أو المسرطنات، والتي تخضع لعملية تصحيح ميّالة للخطأ خاصة (وصل النهاية الميكرو تناظري المتوسط [159]) أو أي خطأ في أنواع أخرى من التصحيح،[160][161] كما يمكن أن تنتج الطفرات من إدخال أو حذف قطع دنا بسبب عناصر وراثية متحركة.[162][163][164] قد وقد لا تُحدِث الطفرات تغيرات قابلة للتمييز في الخصائص المرئية (النمط الظاهري) للكائن وتلعب الطفرات دورا في كل العمليات البيولوجية الطبيعية وغير الطبيعية: السرطان، التطور وتطور الجهاز المناعي بما في ذلك التنوع الوصلي.
يمكن للطفرات التسبب بأنواع مختلفة من التغير في التسلسل، والطفرات في الجين إما أن لا يكون لها تأثير أو أن تُغيِّر ناتج الجين أو تمنع الجين من النشاط كما ينبغي أو بشكل كامل، يمكن للطفرات الحدوث في مناطق غير جينية، تقترح إحدى الدراسات المتعلقة بالتغيرات الجينية بين مختلف أجناس الدروسوفيلا أنه إن غيرت الطفرة بروتينا يُنتَج من الجين فإن النتيجة تكون على الأرجح ضارة مع تقدير 70 بالمئة من تعددات الأشكال بسبب الحمض الأميني لها آثار ضارة والباقي يكون إما حيادي التأثير أو له فائدة هامشية.[165] بسبب التأثيرات الضارة التي يمكن أن تلحقها الطفرات بالجينات؛ يوجد لدى الكائنات آليات مثل ترميم الدنا لمنع أو تصحيح الطفرات بإعادة التسلسل المطفَّر إلى حالته الأصلية.[162]
إعادة التركيب الجيني
 |
 |

في العادة لا يتفاعل لولب الدنا مع قطع أخرى من الدنا، وفي الخلايا البشرية تشغل الكروموسومات مناطق منفصلة في النواة تسمى مناطق الكروموسوم.[167] هذا الفصل الفيزيائي لمختلف الكروموسومات مهم لقدرة الدنا على العمل كمستودع للمعلومات، من المرات القليلة التي تتفاعل فيها الكروموسومات فيما بينها هي في التعابر الكروموسومي الذي يحدث أثناء التكاثر الجنسي والذي يحدث فيه التأشيب الجيني. التعابر الجيني هو انفكاك كروموسومين وتبادل قطعة دنا بينهما ثم إعادة تشكلهما.
يسمح التأشيب الجيني للكروموسومات بتبادل المعلومات الجينية وإنتاج توليفات جديدة من الجينات، والتي تزيد فعالية الاصطفاء الطبيعي ويمكن أن تكون مهمة لسرعة تطور بروتينات جديدة.[168] يمكن أن يكون للتأشيب الجيني علاقة بترميم الدنا خاصة في رد الخلايا على شروخ السلاسل المزدوجة.[169]
أكثر أنواع التعابر الكروموسومي شيوعا هو التأشيب المتماثل حيث يتشارك فيه كروموسومان تسلسلات مماثلة جدا، أما التأشيب غير المتماثل فيمكن أن يكون مضرا بالخلايا كما يمكن أن يُنتِج انتقالات كروموسومية وتشوهات كروموسومية. تُحفَّز عملية التأشيب بواسطة إنزيمات تسمى ريكومبيناز مثل RAD51.[170] أول خطوة في التأشيب هو تقطُّع السلسلة المزدوجة إما بواسطة نوكلياز داخلي أو ضرر في الدنا،[171] بعدها يحفز الريكومبيناز سلسلة من الخطوات تقود إلى تجميع لولبي الدنا بواسطة موصل هوليداي واحد على الأقل والتي يتم فيها تثبيت قطعة من كل سلسلة مفردة لكلا اللولبين في السلسلة المكملة للولب الآخر. موصل هوليداي بنية رباعية يمكنها التحرك على طول زوج الكروموسومات مُبدِّلّة إحدى السلسلتين بالأخرى، بعدها تُوقَفُ عملية التأشيب بفصل الموصل وإعادة تركيب الدنا المنفصل.[172] السلاسل المتماثلة في القطبية وحدها من يتبادل الدنا فيما بينها أثناء التأشيب، هناك نوعان من القص: قص شرق-غرب وقص شمال-جنوب، يشمل القص شمال-جنوب كلا السلسلتين ويقطعهما في حين أن قص شرق-غرب يبقي إحدى السلسلتين سليمة. تَشكُّل موصل هوليداي أثناء التأشيب يسمح بالتنوع الجيني، تبادل الجينات في الكروموسومات، والتعبير عن الجينومات الفيروسية المتنوعة.
عناصر وراثية متحركة


المعلومة الجينية المشفَّرَة في الدنا ليست ثابتة عبر الزمن وبعض التسلسلات عرضة للانتقال من مكان إلى آخر في الجينوم، وهذه التسلسلات هي العناصر الوراثية المتحركة. وهي عبارة عن مطفِّرات يمكنها تغيير جينوم الخلايا ونجد من بينها خاصة الينقولات والينقولات الراجعة، تعمل هذه الأخيرة -عكس الأولى- عبر رنا وسيط مُنتِجَةً تسلسل دنا بواسطة عملية النسخ العكسي. تتحرك هذه العناصر داخل الجينوم تحت تأثير الترانسبوزازات وهي إنزيمات خاصة تفصل هذه العناصر من جهة وتربطها في جهة أخرى من الجينوم، وهي مسؤولة عن انتقال ما لا يقل عن 40% من الجينوم البشري خلال تطور الإنسان العاقل.[173]
تشكل هذه العناصر القابلة للنقل نسبة مهمة من الجينوم لدى الكائنات الحية، خاصة لدى النبات حيث تمثل فيه النسبة الأكبر من الحمض النووي، فمثلا لدى الذرة 49-78% من جينومه عبارة عن ينقولات راجعة،[174] جينوم القمح 90% منه عبارة عن تسلسلات متكررة و68% منه عناصر متحركة،[175] لدى الثدييات نصف الجينوم تقريبا (45-48%) عبارة عن عناصر متحركة أو بقايا عناصر متحركة، وحوالي 42% من الجينوم البشري مكون من الينقولات الراجعة في حين أن 2-3% منه عبارة عن ينقولات دنا.[176] ومنه فهي عناصر مهمة لعمل وتطور جينوم الكائنات الحية.[177]
الإنترونات التي تصنف إلى مجموعة I ومجموعة II هي نوع آخر من العناصر الوراثية المتحركة وهي عبارة عن ريبوزيمات أي تسلسللات رنا تملك قدرة التحفيز مثل الإنزيمات وقادرة على تحفيز وصلها ذاتيا. إنترونات المجموعة I تحتاج نوكليوتيدات غوانين لكي تعمل على عكس إنترونات المجموعة الثانية. توجد على سبيل المثال إنترونات المجموعة I بصورة فردية لدى البكتيريا وبعدد أكبر قليلا لدى حقيقيات النوى البسيطة وبأعداد كبيرة جدا لدى النباتات الوعائية. كما نجدها لدى عدد كبير من عاثيات البكتيريا موجبة الغرام،[178] ولدى القليل من العاثيات سالبة الغرام مثل العاثية T4.[178][179][180][181]
النقل الأفقي للجينات

يمكن لمعلومة الخلية الوراثية التطور تحت تأثير دمج وإضافة مادة وراثية خارجية مُمتصة عبر الغشاء الخلوي ويسمى ذلك النقل الأفقي للجينات، وهو عكس النقل العمودي الناتج من تكاثر الكائنات الحية ويتم بين وحيدات الخلية و/أو متعددات الخلية،[182] وهو عامل أساسي في تطور العديد من الكائنات.[183][184]
النقل الأفقي للجينات هو الآلية الأساسية في انتشار مقاومة المضادات الحيوية لدى البكتيريا [183][185][186][187][188] ويلعب دورا مهما في تطور البكتيريا التي يمكنها تحليل المركبات الصناعية الجديدة التي يصنعها الإنسان مثل المبيدات الحشرية،[189] وتطور وصيانة وانتقال حدة الجرثوم [190] وغالبا ما يتعلق ذلك بالعاثيات والبلازميدات.[191][192][193] يمكن أن تنتقل الجينات المسؤولة عن مقاومة المضادات الحيوية لدى إحدى أنواع البكتيريا إلى أنواع أخرى عبر عدة آليات منها F-pilus أو الشعرة الإخصابية مانحة إياها مقاومة لهذه المضادات وهو أمر طبي صار التعامل معه عسيرا، ويفترض كذلك أن النقل الأفقي للجينات يعزز المحافظة على حياة كيموحيوية شاملة، وكنتيجة لذلك على شمولية الشيفرة الجينية.[194]
تَركَّز معظم الاهتمام والبحث في علم الوراثة على النقل العمودي، لكن بدأت أهمية النقل الأفقي للجينات بين وحيدات الخلية تلقى الاهتمام.[195][196]
يمكن اعتبار توصيل الجين كنقل أفقي للجينات وهو نوع من الهندسة الوراثية.
تطور
يحتوي الدنا على المعلومة الوراثية التي تسمح لكل الكائنات الحية المعاصرة بالتكاثر والنمو والقيام بمختلف الوظائف، لكن من غير الواضح منذ متى في تاريخ الحياة الممتد إلى 4 مليارات سنة كان الدنا يقوم بهذه الوظيفة، وقد اقتُرح أن أشكال الحياة الأولى من الممكن أن تكون استعملت الرنا كمادتها الوراثية.[197][198] يمكن أن يكون الرنا قد لعب دورا رئيسيا في أيض الخلايا الأولية حيث يمكنه القيام بكل من نقل المعلومة الوراثية وبدأ تحفيزات التفاعلات لكونه جزءا من الريبوزيم،[199] يمكن لعالم الرنا القديم هذا الذي كانت تستخدم فيه الأحماض النووية لكل من التحفيز والوراثة أن يكون أثّـر على تطور الشيفرة الجينية الحالية التي أساسها أربع قواعد نووية، وهذا سيحدث لأن رقم مختلف القواعد في كائن بتلك الصفة عبارة عن توافق بين رقم صغير من القواعد يزيد في دقة النسخ ورقم كبير من القواعد يزيد في فعالية التحفيز لدى الريبوزيمات.[200] بيد أنه لا يوجد دليل مباشر على الأنظمة الجينية القديمة لأن استخلاص الدنا من المستحثات مستحيل لكون الدنا يعيش في الطبيعة لأقل من مليون سنة، ثم يتحلل تدريجيا إلى قطع صغيرة في المحاليل.[201] هنالك ادعاءات بوجود دنا أقدم من ذلك وخاصة التقرير الذي يتحدث عن استخلاص بكتيريا حية من بلورة ملح عمرها 250 مليون سنة،[202] لكن هذه الادعاءات مثيرة للجدل وغير متفق عليها.[203][204]
يمكن أن تكون مكونات الدنا (الأدينين، الغوانين وما يتعلق بها من مركبات عضوية) قد تشكلت خارج الأرض في الفضاء الخارجي.[205][206][207] مكونات مركبات الدنا والرنا بما في ذلك اليوراسيل، السايتوسين والثايمين تم تشكيلها كذلك في المخابر تحت ظروف تماثل تلك الموجودة في الفضاء الخارجي باستخدام متفاعلات بدء كيميائية مثل البيريميدينات التي عثر عليها في الأحجار النيزكية، يمكن للبيريميدينات مثل الهيدرو كربونات الأروماتية متعددة الحلقات -أغنى مركب كيميائي بالكربون وجد في الكون- أن يكون تشكل في عملاق أحمر أو في غبار كوني وسحب غاز بين النجوم.[208]
استخدامات في التكنولوجيا
هندسة وراثية
تم تطوير طرق عديدة لاستخلاص الدنا من الكائنات الحية مثل طريقة استخلاص فينول-كلوروفورم وطرقٍ لمعالجته وتعديله في المختبر مثل طريقتي اهتضام التقييد وتفاعل البوليميراز المتسلسل. يستخدم علما الأحياء والكيمياء الحيوية الحديثين هذه الطرق بشكل كبير في تكنولوجيا الدنا المؤشب، الدنا المؤشب هو تسلسل دنا من صنع الإنسان تم تجميعه من تسلسللات دنا أخرى، حيث يمكن نقله إلى كائنات مختلفة على شكل بلازميدات أو على الشكل المناسب باستخدام ناقل فيروسي.[211]
الكائنات المعدلة وراثيا المنتَجَة يمكن استخدامها لإنتاج منتجات مثل البروتينات المؤشبة المستخدمة في البحث الطبي [212] أو لزرعها وتحسين منتجات الزراعة،[213][214] تم تعديل نباتاتٍ لحمايتها من الحشرات وجعل مقاومتها أكبر ضد مبيدات الحشرات، الفيروسات والضغوط الطبيعية وتحسين قيمتها الغذائية ولإنتاج اللقاحات الصالحة للأكل.[215] معظم الكائنات المعدلة وراثيا المروج لها هي إما محاصيل نباتات مقاومة للحشرات أو للمبيدات.[216] يتم استخدام الحيوانات المعدلة جينيا في البحوث أو لتحسين نماذجها أو لإنتاج منتجات زراعية وصيدلية. وتشمل هذه التعديلات إزالة جيناتٍ لجعلها أقل عرضة للمرض، وزيادة الهرمونات لنمو أكبر وللقدرة على توفير البروتينات في الحليب.[217]
إنزيمات الدنا أو دنا محفز
الريبوزيمات منزوعة الأكسجين وتسمى كذلك إنزيمات الدنا أو الدنا المُحفِّز اكتُشِفت أول مرة سنة 1994.[218] وهي في الغالب تسلسل دنا منفرد معزول من كمية كبيرة من تسلسلات دنا عشوائية عبر عملية توليفية تسمى اختيار في المختبر أو تطور منظومي للربائط بواسطة الاغناء المطرد (SELEX). تحفز إنزيمات الدنا مختلف التفاعلات الكيميائية بما في ذلك قص مركبات دنا-رنا، ربط مركبات دنا-رنا، فسفرة ونزع فوسفات الأحماض الآمينية، تشكيل روابط كاربون-كربون ...إلخ. يمكن لإنزيمات الدنا تحسين معدل تحفيز العمليات الكيميائية إلى 100 مليار مرة العملية غير المحفزة.[219] أكثر أصناف إنزيمات الدنا المدروسة دراسة وافية هي الأنواع القاصَّة للرنا والتي تستخدم لكشف مختلف الأيونات الفلزية وتصميم عوامل علاجية. تم تقرير العديد من إنزيمات الدنا المختصة بالفلزات منها: إنزيم الدنا GR-5 (المختص بالرصاص)،[218] إنزيمات الدنا CA1-3 (المختصة بالنحاس)،[220] إنزيم الدنا 39E (المختص باليورانيل) وإنزيم الدنا NaA43 (المختص بالصوديوم).[221] إنزيم الدنا NaA43 الذي أشير إلى أنه يختار أيونات الصوديوم 10.000 مرة عن الآيونات الفلزية الأخرى، استُخدم لإنشاء مستشعرات صوديوم حقيقية في خلايا حية.
الشرطة العلمية والطب الشرعي

يمكن لعلماء الأدلة الجنائية استخدام الدنا الموجود في الدم، المني، الجلد، اللعاب والشعر المعثور عليه في مسرح الجريمة لتحديد دنا مطابق لفرد ما، كالجاني مثلا،[222] ويطلق على هذه العملية البصمة الوراثية. يُقارن في البصمة الوراثية تسلسل أطوال مختلفة متكررة من الدنا مثل التكرارات المترادفة القصيرة ودنا الساتل الصغير بين الناس. هذه الطريقة في العادة موثوق بها جدا لتحديد دنا متطابق،[223] لكن يمكن أن يكون التحديد معقدا إذا كان المسرح ملوثا بدنا من عدة أشخاص.[224] تم ابكتار تقنية البصمة الوراثية سنة 1984 بواسطة عالم الوراثة البريطاني أليك جيفريز،[225] واستخدمت أول مرة في علم الأدلة الجنائي لإدانة كولين بيتشفورك سنة 1988 في قضية قتيلتي إندربي.[226]
تُستخدم البصمة الوراثية كذلك في اختبار الأبوة بالدنا لتحديد إن كان أحدهم الوالد أو الجد البيولوجي للطفل مع احتمال تطابق تصل نسبته إلى 99.99% حين يكون الوالد المفترض قريبا بيولوجيا للطفل. وتُستخدم لذلك طرق كشف التسلسل العادية بعد الولادة، في حين تستخدم طرق جديدة لاختبار الأبوة حين تكون الأم مازالت حاملا.[227]

المعلوماتية الحيوية
تتضمن المعلوماتية الحيوية تطوير تقنياتٍ لتخزين وتنقيب وبحث ومعالجة البيانات البيولوجية بما في ذلك بيانات تسلسل الأحماض النووية، وقاد ذلك إلى تطورات مستخدمة بشكل واسع في علم الحاسوب خاصة في خوارزميات البحث التسلسلية، تعلم الآلة ونظرية قواعد البيانات،[228] تم تطوير البحث التسلسلي أو خوارزميات التطابق التي تعثر على وجود تسلسل حروف معين داخل تسلسل حروف أكبر خصيصا للبحث عن تسلسلات نوكليوتيدات معينة.[229]
يمكن أن يتم صف تسلسل دنا مع تسلسل دنا آخر لتحديد التسلسللات المتماثلة وتعيين مواضع الطفرات التي تجعلهما مختلفين، تُستخدم هذه التقنيات وخاصة تقنية تراصف التسلسلات المتعدد في دراسة العلاقات في علم الوراثة العرقي ووظيفة البروتين.[230] مجموعات البيانات التي تمثل تسلسلات جينومات كاملة مثل تلك الناتجة عن مشروع الجينوم البشري يصعب استخدامها من دون حاشية شرح لتحديد أماكن الجينات والعناصر المنظِّمة في كل كروموسوم، مناطق الدنا التي لديها الخصائص النمطية المتعلقة بجينات البروتين -أو الرنا- يمكن تحديدها بخوارزميات البحث عن الجين، وهو ما يسمح للباحثين بتوقع وجود نواتج جينية معينة ووظائفها المحتملة في الكائنات قبل حتى استخلاصها تجريبيا ودراستها.[231] كما يمكن مقارنة جينومات بأكملها وهو ما يسمح بتسليط الضوء على تاريخ تطور بعض الكائنات وفحص أحداث تطورية معقدة.
التقنية النانوية للدنا

تَستخدِم تقنيات الدنا النانوية خصائص التعرف الجزيئي الفريدة للدنا والأحماض النووية الأخرى لإنشاء مركبات دنا متفرعة ذاتية التجميع لها خصائص مفيدة،[232] ومنه فإن الدنا يُستخدم كمادة هيكلية بدلا من أن يُستخدم كحامل للمعلومة البيولوجية، وقاد هذا إلى إنشاء شبكيات دورية ثنائية الأبعاد باستخدام طريقة التجميع بالوحدات الأساسية (tile-based) وكذلك طريقة أوريغامي الدنا وإنشاء هياكل ثلاثية الأبعاد على شكل متعدد سطوح.[233] كما تم إنجاز أدوات نانومترية وخوارزمية التجميع الذاتي.[234] وتم استخدام هياكل الدنا هذه كقالب تُرتَّبُ فيه جزيئات أخرى مثل الجسيمات النانوية من الذهب وبروتينات الستربتافيدين [235] وهي بروتينات تشكل مركبات مع البيوتين.
قادت البحوث في مجال الإلكترونيات الجزيئية المبنية على الدنا شركة مايكروسوفت لتطوير لغة برمجة تسمى إزاحة سلسلة الدنا DNA Strand Displacement (DSD) [236] وتم استخدامها في تجسيد بعض المكونات النانوإلكترونية الجزيئية المبنية على الدنا.[237][238]
التاريخ والأنثروبولوجيا
لأن الطفرات تتراكم في الدنا مع مرور الوقت وبعدها يتم توريثها للأجيال التالية، فإن الدنا يحتوي على معلومات تاريخية وبمقارنة تسلسلات الدنا المختلفة يمكن لعلماء الوراثة استنتاج التاريخ التطوري للكائنات وهذا ما يعرف بعلم الوراثة العرقي.[239] يشكل هذا المجال -بوضع علم الوراثة في خدمة علم أحياء الحفريات- أداة قوية في علم الأحياء التطوري، إذا تمت مقارنة سلاسل الدنا داخل نفس النوع يمكن لعلماء الوراثيات السكانية معرفة تاريخ شعوب معينة، ويمكن استخدام ذلك في دراسات تترواح بين علم الوراثة البيئي إلى الأنثروبولوجيا.
تُستخدم دراسة دنا الميتوكندريا داخل التجمعات البشرية لتحديد هجرات الإنسان العاقل، على سبيل المثال تمت دراسة النمط الجماعي X في علم الديموغرافيا القديمة لتقييم القرابة المحتملة لأسلاف الهنود بالشعوب الأوربية التي تعود أصولها إلى العصر الحجري القديم العلوي.[240]
 شجرة تطور السلالات توضح نطاقات الأحياء الثلاثة: حقيقيات النوى بالأحمر، العتائق بالأخضر والبكتيريا بالأزرق.[241]
شجرة تطور السلالات توضح نطاقات الأحياء الثلاثة: حقيقيات النوى بالأحمر، العتائق بالأخضر والبكتيريا بالأزرق.[241] خريطة الهجرة البشرية المستنتجة من دراسات علم الوراثة العرقي لدنا المتقدرة البشري، المقياس 1000 سنة قبل الحاضر.[242]
خريطة الهجرة البشرية المستنتجة من دراسات علم الوراثة العرقي لدنا المتقدرة البشري، المقياس 1000 سنة قبل الحاضر.[242]
تخزين المعلومات
يُستخدَم الدنا بواسطة الكائنات الحية لتخزين معلوماتهم الوراثية ولهذا تقوم مجموعات من الباحثين بدراسته كوسيلة تخزين للبيانات الرقمية تماما مثل وسائط التخزين الحاسوبية وذلك لأن لدى الأحماض النووية كثافة تخزين نظرية تفوق بشكل كبير وسائل التخزين التقلدية وتملك عمرا فتراضيا أكبر بكثير كذلك.
نظريا يمكن تشفير حتى 2 بت من المعلومات في كل نوكليوتيد وهذا يسمح بقدرة تخزين تصل إلى 455 مليون تيرابايت لكل غرام واحد من الدنا منفرد السلسلة، حيث تبقى صالحة للقراءة لعدة ألفيات حتى في ظروف تخزين غير مثالية،[243] تم اقتراح تقنية تخزين تصل إلى 215 ألف تيرابايت لكل غرام من الدنا سنة 2017 [244] بالمقارنة مع سعة قرص دي في دي بوجهي تخزين ذو طبقتين والتي بالكاد تصل 17 جيغابايت لوزن نموذجي يصل 16غ نجد أن قدرته على التخزين أقل 400 مليار مرة لكل غرام. قامت مجموعة باحثين من معهد المعلوماتية الحيوية الأوروبي سنة 2012 بتشفير 757 كيلوبايت من البيانات في 17 940 195 نوكليوتيدة [245] وهو ما يعادل كثافة تخزين تصل حوالي 2200 تيرابايت لكل غرام من الدنا.[246] ومن جانبه؛ نشر فريق سويسري في فبراير 2015 دراسة توضح متانة الدنا المغلف بالسيليكا كداعم مستدام للمعلومة.[247]
من جانب آخر تعمل فرق أخرى على إمكانية تخزين المعلومات مباشرة في الخلايا الحية، لغايات مختلفة على سبيل المثال لتشفير عدّادات في دنا الخلية لتحديد عدد الانقسامات أو التمايزات وهو أمر يسمح بإيجاد تطبيقات له في مجال البحوثات حول السرطان والشيخوخة.[248]
تاريخ البحث في الدنا

تم استخلاص الدنا لأول مرة سنة 1869 بواسطة العالم السويسري فريدريك ميشر الذي اكتشف مادة مجهرية في قيحٍ على ضمادة جراحية مستعملة، ولأن مكانها كان في نواة الخلية فقد أسماها "نوكليين".[249][250] وفي سنة 1878 استخلص ألبرشت كوسل مكون غير بروتيني للنوكليين، وحمضا نوويا وتمكن لاحقا من استخلاص قواعده النووية الخمس الأساسية،[251][252] وفي 1919 قام فيبوس ليفين بتعريف قاعدةِ وسكرِ وفوسفاتِ وحدةِ النوكليوتيد [253] واقترح أن الدنا يتكون من سلسلة من وحدات النوكليوتيد مترابطة مع بعضها بواسطة مجموعات فوسفات، ولكن اعتقد أن السلسلة مكونة من تكرار قواعد قصير له ترتيب ثابت. في 1927 اقترح نيكولاي كولتسوف أن الصفات الوراثية يتم توارثها عبر "جزيء وراثي عملاق مكون من سلسلتين متناظرتين يقوم بالتضاعف بطريقة نصف محافظة باستخدام كل سلسلة كقالب.[254][255] سنة 1928 اكتشف فريدريك غريفيث في تجربته أن صفات "النعومة" لدى المكورات الرئوية يمكن أن تتحول إلى "خشنة" من نفس البكتيريا وذلك بخلط البكتيريا الناعمة الميتة مع الحية الخشنة.[256][257] منح هذا النظام أول اقتراح واضح بأن الدنا يحمل المعلومة الوراثية.
وفي سنة 1933 اقترح جان براشيه - بينما كان يدرس بيوض قنفذ البحر- أن الدنا يتواجد في نواة الخلية وأن الرنا يتواجد حصريا في السيتوبلازم. اعتُقِد في ذلك الوقت أن "حمض الخميرة النووي" (الرنا) لا يظهر سوى عند النباتات وأن حمض الغدة الزعترية النووي (الدنا) لا يظهر سوى عند الحيوانات، وأن هذا الأخير عبارة عن جزيء رباعي له وظيفة تنظيم الأس الهيدروجيني (PH) الخلوي. وفي 1937 أنتج وليام أستبري أول نمط حيود للأشعة السينية أظهر أن للدنا بنية منتظمة.[258] في سنة 1943 حدد أوزوالد آفري مع زميليه كولين ماكلويد وماكلين ماكرتي أن الدنا هو أساس التحول، داعمين مقترح غريفث (تجربة أيفري-ماكلويد-ماكرتي)،[259] تم تأكيد دور الدنا في الوراثة سنة 1952 بواسطة ألفرد هيرشي ومارثا تشايس في تجربة هيرشي-تشايس التي أوضحت أن الدنا هو المادة الوراثية للعاثية T2.[260]


في نهاية 1951 بدأ فرنسيس كريك بالعمل مع جيمس واتسون في مختبر كافندش داخل جامعة كامبريدج وفي 1953 اقترحا ما هو الآن مقبول كأول بنية لولب مزدوج صحيحة للدنا في مجلة نيتشر.[8] نموذج لولبهما المزدوج كان معتمدا على صورة حيود أشعة إكس (سميت الصورة 51) [261] أخذت بواسطة روزاليند فرانكلين وريموند غوسلين في ماي 1952 ومعتمدا كذلك على معلومة أن قواعد الدنا مترابطة زوجيا، في 28 فبراير 1953 قاطع كريك وقت غذاء مموله في حانة إيجل ليعلن أنه بمعية واتسون اكتشفا سر الحياة.[262]
بشهور قبل ذلك -في فبراير 1953- اقترح لينوس باولنغ وروبرت كوري نموذجا للدنا يحتوي ثلاث سلاسل لولبية، مع كون مجموعات الفوسفات متجهة للداخل والقواعد للخارج.[263] تم نشر دليل تجريبي يدعم نموذج كريك وواتسون على شكل سلسلة خمس مقالات في نفس إصدار المجلة نيتشر [264]، ومن تلك المقالات كان بحث فرانكلين وغوسلين أول مقالة نشرت حول بيانات صورة حيود الأشعة أكس خاصتهم وطريقة التحليل الأصلية التي دعمت نموذج واتسون وكريك جزئيا،[61][265] كما حوى هذا الإصدار على مقال حول بنية الدنا بواسطة موريس ويلكنز واثنين من زملائه، الذين دعمت تحاليلهم وأنماط حيود الأشعة السينية خاصتهم لبنية الدنا ب كذلك وجود لولب مزدوج كبنية للدنا تماما مثل ما اقترح كريك وواتسون في صفحتين قبلهم من المجلة.[62] في سنة 1962 وبعد وفاة فرانكلين حصل واتسون وكريك وويلكنز جماعيا على جائزة نوبل في الطب أو علم وظائف الأعضاء،[266] جائزة نوبل لا تمنح سوى للأحياء ولهذا مازال هنالك جدال حول من يستحق فضل هذا الاكتشاف.[267]
نشر كريك سنة 1957 وثيقة يوضح فيها ما يعرف حاليا بهدف علم الأحياء الجزيئي الرئيسي والذي وضح العلاقة بين الدنا والرنا والبروتينات وطرح "نظرية الموَصِّل" [268] وجاء آخر تأكيد على آلية التضاعف التي طرحت نتيجة هيئة اللولب المزدوج عبر تجربة ميسلسون-ستال سنة 1958.[269] وأوضحت أعمال أخرى لكريك وزملائه أن الشيفرة الجينية مبنية على أساس قواعد ثلاثية غير متداخلة تسمى الرامزات أو الكودونات وهو ما سمح لهار غوبند خورانا وروبرت هولي ومارشال نيرنبرغ بفك شيفرة الشيفرة الجينية،[270] وتمثل هذه الاكتشافات ميلاد علم الأحياء الجزيئي.[271]
التسمية
تُذكر المَعاجم والقواميس العَربية تسمياتٍ عدة للحمض النووي الريبوزي منقوص الأكسجين (Deoxyribonucleic acid)، ومن أَهمِها:
- حَمْض الدَّيوكْسِي رِيبونوكلئيك أو الحَامِض النَّوَوِيّ الرِّيبي المَنْقُوص الأُكْسجِين، وذلك حَسبَ معجم مرعشي الطبي الكبير.[272]
- حَمْض الديوكسي ريبونوكلييك أو الحَمْض الرِّيبي النَّوَوِيُّ المنزوعُ الأُكسجين أو حَمْض الديوكسي ريبونيوكليك، وذلك حَسبَ المعجم الطبي الموحد.[273][274]
- يُترجم حرفيًا إلى حمض الديوكسيريبونكلييك، وذلك حَسب قاموس المعاني.[275]
يُدعى اختصارًا دنا أو د.ن.أ أو دي.إن.إيه (DNA).[276]
انظر أيضًا
المراجع
- القاموس الطبي الموحد Deoxyribonucleic acid تاريخ الولوج 14 كانون الثاني 2014 نسخة محفوظة 27 أغسطس 2017 على موقع واي باك مشين.
- تسمية الحمض النووي الصبغي في موقع قاموس Riverso نسخة محفوظة 2020-08-21 على موقع واي باك مشين.
- Molecular Biology of the Cell (ط. 6th)، Garland، 2014، ص. Chapter 4: DNA, Chromosomes and Genomes، ISBN 9780815344322، مؤرشف من الأصل في 14 يوليو 2014.
- Purcell, Adam، "DNA"، Basic Biology، مؤرشف من الأصل في 05 يناير 2017.
- Saenger, Wolfram (1984)، Principles of Nucleic Acid Structure، New York: Springer-Verlag، ISBN 0-387-90762-9.
- Alberts, Bruce؛ Johnson, Alexander؛ Lewis, Julian؛ Raff, Martin؛ Roberts, Keith؛ Peter, Walters (2002)، Molecular Biology of the Cell (ط. Fourth)، New York and London: Garland Science، ISBN 0-8153-3218-1، OCLC 145080076، مؤرشف من الأصل في 18 أغسطس 2020.
- "Structural diversity of supercoiled DNA"، Nature Communications، 6: 8440، أكتوبر 2015، Bibcode:2015NatCo...6E8440I، doi:10.1038/ncomms9440، PMC 4608029، PMID 26455586.
- "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF)، Nature، 171 (4356): 737–8، أبريل 1953، Bibcode:1953Natur.171..737W، doi:10.1038/171737a0، PMID 13054692، مؤرشف من الأصل (PDF) في 04 فبراير 2007.
- "The dimensions of DNA in solution"، Journal of Molecular Biology، 152 (1): 153–61، أكتوبر 1981، doi:10.1016/0022-2836(81)90099-1، PMID 7338906.
- Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, et al. (May 2006). "The DNA sequence and biological annotation of human chromosome 1". Nature. 441 (7091): 315–21. Bibcode:2006Natur.441..315G. doi:10.1038/nature04727. PMID 16710414.
- Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company (ردمك 0-7167-4955-6)
- Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents نسخة محفوظة 5 February 2007 على موقع واي باك مشين. IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Retrieved 3 January 2006.
- "A glossary of DNA structures from A to Z"، Acta Crystallographica Section D، 59 (Pt 4): 620–6، أبريل 2003، doi:10.1107/S0907444903003251، PMID 12657780.
- "Base-stacking and base-pairing contributions into thermal stability of the DNA double helix"، Nucleic Acids Research، 34 (2): 564–74، 2006، doi:10.1093/nar/gkj454، PMC 1360284، PMID 16449200.
- Burton E. Tropp – "Molecular Biology"- Jones and Barlett Learning, (ردمك 978-0-7637-8663-2)
- "Watson-Crick Structure of DNA – 1953"، Steven Carr، Memorial University of Newfoundland، مؤرشف من الأصل في 19 يوليو 2016، اطلع عليه بتاريخ 13 يوليو 2016.
- "A historical account of Hoogsteen base-pairs in duplex DNA"، Biopolymers، 99 (12): 955–68، 2013، doi:10.1002/bip.22334، PMC 3844552، PMID 23818176.
- "Mechanical stability of single DNA molecules"، Biophysical Journal، 78 (4): 1997–2007، أبريل 2000، Bibcode:2000BpJ....78.1997C، doi:10.1016/S0006-3495(00)76747-6، PMC 1300792، PMID 10733978.
- "Yersiniophage phiR1-37 is a tailed bacteriophage having a 270 kb DNA genome with thymidine replaced by deoxyuridine"، Microbiology، 151 (Pt 12): 4093–102، ديسمبر 2005، doi:10.1099/mic.0.28265-0، PMID 16339954.
- "Intragenus generalized transduction in Staphylococcus spp. by a novel giant phage"، The ISME Journal، 8 (9): 1949–52، سبتمبر 2014، doi:10.1038/ismej.2014.29، PMC 4139722، PMID 24599069.
- Casella E, Markewych O, Dosmar M, Heman W (1978) Production and expression of dTMP-enriched DNA of bacteriophage SP15. J Virology 28 (3) 753–766
- "A base called J"، Proceedings of the National Academy of Sciences of the United States of America، 95 (5): 2037–8، مارس 1998، Bibcode:1998PNAS...95.2037S، doi:10.1073/pnas.95.5.2037، PMC 33841، PMID 9482833.
- "Base J: discovery, biosynthesis, and possible functions"، Annual Review of Microbiology، 62: 235–51، 2008، doi:10.1146/annurev.micro.62.081307.162750، PMID 18729733.
- "The modified base J is the target for a novel DNA-binding protein in kinetoplastid protozoans"، The EMBO Journal، 18 (22): 6573–81، نوفمبر 1999، doi:10.1093/emboj/18.22.6573، PMC 1171720، PMID 10562569.
- "Regulation of trypanosome DNA glycosylation by a SWI2/SNF2-like protein"، Molecular Cell، 17 (3): 441–51، فبراير 2005، doi:10.1016/j.molcel.2004.12.022، PMID 15694344.
- "Evidence that J-binding protein 2 is a thymidine hydroxylase catalyzing the first step in the biosynthesis of DNA base J"، Molecular and Biochemical Parasitology، 164 (2): 157–61، أبريل 2009، doi:10.1016/j.molbiopara.2008.12.001، PMID 19114062.
- "Prediction of novel families of enzymes involved in oxidative and other complex modifications of bases in nucleic acids"، Cell Cycle، 8 (11): 1698–710، يونيو 2009، doi:10.4161/cc.8.11.8580، PMC 2995806، PMID 19411852.
- "Glucosylated hydroxymethyluracil, DNA base J, prevents transcriptional readthrough in Leishmania"، Cell، 150 (5): 909–21، أغسطس 2012، doi:10.1016/j.cell.2012.07.030، PMC 3684241، PMID 22939620.
- "Transcription: base J blocks the way"، Current Biology، 22 (22): R960-2، نوفمبر 2012، doi:10.1016/j.cub.2012.10.010، PMC 3648658، PMID 23174300.
- "Cyanophage S-2L contains DNA with 2,6-diaminopurine substituted for adenine"، Virology، 88 (1): 8–18، 1978، PMID 676082.
- ncbi. نسخة محفوظة 20 أبريل 2018 على موقع واي باك مشين.
- "Novel genomic island modifies DNA with 7-deazaguanine derivatives"، Proceedings of the National Academy of Sciences of the United States of America، 113 (11): E1452–9، 2016، doi:10.1073/pnas.1518570113، PMC 4801273، PMID 26929322.
- "Pyrimidines. CIII. The discovery of 5-methylcytosine in tuberculinic acid, the nucleic acid of the tubercle bacillus."، Journal of the American Chemical Society، 47: 2838–44، 1925.
- "The bases of the nucleic acids of some bacterial and animal viruses: the occurrence of 5-hydroxymethylcytosine"، The Biochemical Journal، 55 (5): 774–82، 1953، PMC 1269533، PMID 13115372.
- "5-Methylcytosine replacing cytosine in the deoxyribonucleic acid of a bacteriophage for Xanthomonas oryzae"، Journal of Molecular Biology، 34 (2): 373–5، 1968، PMID 5760463.
- Vogelsang-Wenke, Heike؛ Oesterhelt, Dieter (مارس 1988)، "Isolation of a halobacterial phage with a fully cytosine-methylated genome"، MGG Molecular & General Genetics، 211 (3): 407–414، doi:10.1007/BF00425693.
- "Occurrence of a new base in the deoxyribonucleic acid of a strain of Bacterium coli"، Nature، 175 (4451): 336–7، 1955، PMID 13235889.
- "Analysis of bacteriophage mu and lambda-mu hybrid DNAs by specific endonucleases"، Journal of Molecular Biology، 92 (4): 529–40، 1975، PMID 1097703.
- "Methylated guanine derivative as a minor base in the DNA of phage DDVI Shigella disenteriae"، Biochimica et Biophysica Acta، 435 (2): 206–10، 1976، PMID 779843.
- "Cytosine modification in DNA by BcnI methylase yields N4-methylcytosine"، FEBS Letters، 161 (1): 131–4، 1983، PMID 6884523.
- "Replacement of the deoxycytidine residues in Rhizobium bacteriophage RL38JI DNA"، FEBS Letters، 184 (2): 294–8، 1985، PMID 2987032.
- "5-[(Hydroxymethyl)-O-pyrophosphoryl]uracil, an intermediate in the biosynthesis of alpha-putrescinylthymine in deoxyribonucleic acid of bacteriophage phi W-14"، Biochemistry، 20 (12): 3586–91، 1981، PMID 7260058.
- "Non-canonical bases in the genome: The regulatory information layer in DNA"، Angewandte Chemie (International Ed. in English)، 2017، doi:10.1002/anie.201708228، PMID 28941008.
- "Crystal structure analysis of a complete turn of B-DNA"، Nature، 287 (5784): 755–8، أكتوبر 1980، Bibcode:1980Natur.287..755W، doi:10.1038/287755a0، PMID 7432492.
- "Protein-DNA recognition"، Annual Review of Biochemistry، 53: 293–321، 1984، doi:10.1146/annurev.bi.53.070184.001453، PMID 6236744.
- Designation of the two strands of DNA نسخة محفوظة 24 April 2008 على موقع واي باك مشين. JCBN/NC-IUB Newsletter 1989. Retrieved 7 May 2008
- "Non-coding RNAs: hope or hype?"، Trends in Genetics، 21 (5): 289–97، مايو 2005، doi:10.1016/j.tig.2005.03.007، PMID 15851066.
- "Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns"، Journal of Cellular Biochemistry، 93 (4): 664–71، نوفمبر 2004، doi:10.1002/jcb.20252، PMID 15389973.
- "Overlapping genes in vertebrate genomes"، Computational Biology and Chemistry، 29 (1): 1–12، فبراير 2005، doi:10.1016/j.compbiolchem.2004.12.006، PMID 15680581.
- "Properties of overlapping genes are conserved across microbial genomes"، Genome Research، 14 (11): 2268–72، نوفمبر 2004، doi:10.1101/gr.2433104، PMC 525685، PMID 15520290.
- "Diversity of coding strategies in influenza viruses"، Trends in Genetics، 7 (8): 261–6، أغسطس 1991، doi:10.1016/0168-9525(91)90326-L، PMID 1771674.
- What Is the Advantage of Having the DNA Tightly Wrapped Into the Chromosomes?. نسخة محفوظة 13 فبراير 2018 على موقع واي باك مشين.
- "DNA mechanics"، Annual Review of Biomedical Engineering، 7: 21–53، 2005، doi:10.1146/annurev.bioeng.6.062403.132016، PMID 16004565.
- "DNA topoisomerases: structure, function, and mechanism"، Annual Review of Biochemistry، 70: 369–413، 2001، doi:10.1146/annurev.biochem.70.1.369، PMID 11395412.
- "Cellular roles of DNA topoisomerases: a molecular perspective"، Nature Reviews. Molecular Cell Biology، 3 (6): 430–40، يونيو 2002، doi:10.1038/nrm831، PMID 12042765.
- "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques"، Proceedings of the National Academy of Sciences of the United States of America، 96 (14): 7853–8، يوليو 1999، Bibcode:1999PNAS...96.7853C، doi:10.1073/pnas.96.14.7853، PMC 22151، PMID 10393911.
- "Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA"، Molecular Microbiology، 16 (5): 817–24، يونيو 1995، doi:10.1111/j.1365-2958.1995.tb02309.x، PMID 7476180.
- "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern" (PDF)، Biochemistry، 43 (51): 15996–6010، ديسمبر 2004، doi:10.1021/bi048221v، PMID 15609994، مؤرشف من الأصل (PDF) في 10 يونيو 2007.
- "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies"، Journal of Biomolecular Structure & Dynamics، 6 (2): 299–309، أكتوبر 1988، doi:10.1080/07391102.1988.10507714، PMID 2482766.
- "The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content" (PDF)، Acta Crystallogr، 6 (8–9): 673–7، 06 مارس 1953، doi:10.1107/S0365110X53001939، مؤرشف من الأصل (PDF) في 09 يناير 2016.
"The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function"، Acta Crystallogr، 6 (8–9): 678–85، 1953، doi:10.1107/S0365110X53001940. - "Molecular configuration in sodium thymonucleate" (PDF)، Nature، 171 (4356): 740–1، أبريل 1953، Bibcode:1953Natur.171..740F، doi:10.1038/171740a0، PMID 13054694، مؤرشف من الأصل (PDF) في 03 يناير 2011.
- "Molecular structure of deoxypentose nucleic acids" (PDF)، Nature، 171 (4356): 738–40، أبريل 1953، Bibcode:1953Natur.171..738W، doi:10.1038/171738a0، PMID 13054693، مؤرشف من الأصل (PDF) في 13 مايو 2011.
- "Polymorphism of DNA double helices"، Journal of Molecular Biology، 143 (1): 49–72، أكتوبر 1980، doi:10.1016/0022-2836(80)90124-2، PMID 7441761.
- Baianu, I.C. (1980)، "Structural Order and Partial Disorder in Biological systems"، Bull. Math. Biol.، 42 (4): 137–141، doi:10.1007/BF02462372. "Archived copy"، مؤرشف من الأصل في 25 يوليو 2009، اطلع عليه بتاريخ 06 مايو 2009.
{{استشهاد ويب}}: صيانة CS1: الأرشيف كعنوان (link) - Hosemann R., Bagchi R.N., Direct analysis of diffraction by matter, North-Holland Publs., Amsterdam – New York, 1962.
- Baianu, I.C. (1978)، "X-ray scattering by partially disordered membrane systems"، Acta Crystallogr A، 34 (5): 751–753، Bibcode:1978AcCrA..34..751B، doi:10.1107/S0567739478001540.
- "Crystal structures of A-DNA duplexes"، Biopolymers، 44 (1): 45–63، 1997، doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#، PMID 9097733.
- "A-form conformational motifs in ligand-bound DNA structures"، Journal of Molecular Biology، 300 (4): 819–40، يوليو 2000، doi:10.1006/jmbi.2000.3690، PMID 10891271.
- "DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles"، Immunological Reviews، 184: 286–98، ديسمبر 2001، doi:10.1034/j.1600-065x.2001.1840125.x، PMID 12086319.
- "Z-DNA-binding proteins can act as potent effectors of gene expression in vivo"، Proceedings of the National Academy of Sciences of the United States of America، 99 (26): 16666–71، ديسمبر 2002، Bibcode:2002PNAS...9916666O، doi:10.1073/pnas.262672699، PMC 139201، PMID 12486233.
-
DNA structure and function، Academic Press، ISBN 0-12-645750-6.
{{استشهاد بكتاب}}: يحتوي الاستشهاد على وسيط غير معروف وفارغ:|month=(مساعدة) -
Rich A, Norheim A, Wang AHJ، "The chemistry and biology of left-handed Z-DNA"، Annual Review of Biochemistry، 53: 791–846، doi:10.1146/annurev.bi.53.070184.004043، PMID 6383204.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Ho PS (27 سبتمبر 1994)، "The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA"، Proc Natl Acad Sci USA، 91 (20): 9549–9553، Bibcode:1994PNAS...91.9549H، doi:10.1073/pnas.91.20.9549، PMC 44850، PMID 7937803.
- Created from نسخة محفوظة 17 October 2016 على موقع واي باك مشين.
- Frédéric Pâques et James E. Haber, « Multiple Pathways of Recombination Induced by Double-Strand Breaks in Saccharomyces cerevisiae », في Microbiology and Molecular Biology Reviews, vol. 63, no 2, juin 1999, ص. 349-404 [النص الكامل, lien PMID (pages consultées le 19-02-2018)]
- Irina Voineagu, Vidhya Narayanan, Kirill S. Lobachev et Sergei M. Mirkin, « Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins », في Proceedings of the National Academy of Sciences of the United States of America, vol. 105, no 29, 22 juillet 2008, ص. 9936-9941 [النص الكامل, lien PMID, lien DOI (pages consultées le 14 mars 2015)]
- Guy-Franck Richard, Alix Kerrest et Bernard Dujon, « Comparative Genomics and Molecular Dynamics of DNA Repeats in Eukaryotes », في Microbiology and Molecular Biology Reviews, vol. 72, no 4, décembre 2008, ص. 686–727 [النص الكامل, lien PMID, lien DOI (pages consultées le 14 mars 2015)]
- "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts"، Cell، 43 (2 Pt 1): 405–13، ديسمبر 1985، doi:10.1016/0092-8674(85)90170-9، PMID 3907856.
- "The telomerase reverse transcriptase: components and regulation"، Genes & Development، 12 (8): 1073–85، أبريل 1998، doi:10.1101/gad.12.8.1073، PMID 9553037.
- "Normal human chromosomes have long G-rich telomeric overhangs at one end"، Genes & Development، 11 (21): 2801–9، نوفمبر 1997، doi:10.1101/gad.11.21.2801، PMC 316649، PMID 9353250.
- "Quadruplex DNA: sequence, topology and structure"، Nucleic Acids Research، 34 (19): 5402–15، 2006، doi:10.1093/nar/gkl655، PMC 1636468، PMID 17012276.
- "Crystal structure of parallel quadruplexes from human telomeric DNA"، Nature، 417 (6891): 876–80، يونيو 2002، Bibcode:2002Natur.417..876P، doi:10.1038/nature755، PMID 12050675.
- "Mammalian telomeres end in a large duplex loop"، Cell، 97 (4): 503–14، مايو 1999، doi:10.1016/S0092-8674(00)80760-6، PMID 10338214.
- "DNA enables nanoscale control of the structure of matter"، Quarterly Reviews of Biophysics، 38 (4): 363–71، نوفمبر 2005، doi:10.1017/S0033583505004087، PMC 3478329، PMID 16515737.
- Palmer, Jason (02 ديسمبر 2010)، "Arsenic-loving bacteria may help in hunt for alien life"، BBC News، مؤرشف من الأصل في 03 ديسمبر 2010، اطلع عليه بتاريخ 02 ديسمبر 2010.
- Bortman, Henry (02 ديسمبر 2010)، "Arsenic-Eating Bacteria Opens New Possibilities for Alien Life"، Space.com، مؤرشف من الأصل في 04 ديسمبر 2010، اطلع عليه بتاريخ 02 ديسمبر 2010.
- Katsnelson, Alla (02 ديسمبر 2010)، "Arsenic-eating microbe may redefine chemistry of life"، Nature News، doi:10.1038/news.2010.645، مؤرشف من الأصل في 24 فبراير 2012.
- Cressey, Daniel (03 أكتوبر 2012)، "'Arsenic-life' Bacterium Prefers Phosphorus after all"، Nature News، doi:10.1038/nature.2012.11520.
- "Epigenetic regulation of human embryonic stem cells"، Frontiers in Genetics، 3: 238، 2012، doi:10.3389/fgene.2012.00238، PMC 3488762، PMID 23133442.
- "Genomic DNA methylation: the mark and its mediators"، Trends in Biochemical Sciences، 31 (2): 89–97، فبراير 2006، doi:10.1016/j.tibs.2005.12.008، PMID 16403636.
- "DNA methylation patterns and epigenetic memory"، Genes & Development، 16 (1): 6–21، يناير 2002، doi:10.1101/gad.947102، PMID 11782440.
- "Cytosine methylation and DNA repair"، Current Topics in Microbiology and Immunology، Current Topics in Microbiology and Immunology، 301: 283–315، 2006، doi:10.1007/3-540-31390-7_11، ISBN 3-540-29114-8، PMID 16570853.
- "The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain"، Science، 324 (5929): 929–30، مايو 2009، Bibcode:2009Sci...324..929K، doi:10.1126/science.1169786، PMC 3263819، PMID 19372393.
- "N6-methyladenine: the other methylated base of DNA"، BioEssays، 28 (3): 309–15، مارس 2006، doi:10.1002/bies.20342، PMC 2754416، PMID 16479578.
- "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei"، Cell، 75 (6): 1129–36، ديسمبر 1993، doi:10.1016/0092-8674(93)90322-H، PMID 8261512.
- Henri G.A.M. van Luenen, Carol Farris, Sabrina Jan, Paul-Andre Genest, Pankaj Tripathi, Arno Velds, Ron M. Kerkhoven, Marja Nieuwland, Andrew Haydock, Gowthaman Ramasamy, Saara Vainio, Tatjana Heidebrecht, Anastassis Perrakis, Ludo Pagie, Bas van Steensel, Peter J. Myler et Piet Borst, « Glucosylated Hydroxymethyluracil, DNA Base J, Prevents Transcriptional Readthrough in Leishmania », في Cellvolume = 150, no 5, 31 août 2012, ص. 909-921 [النص الكامل, lien PMID, lien DOI (pages consultées le 12 mars 2015)]
- Dane Z. Hazelbaker et Stephen Buratowski, « Transcription: Base J Blocks the Way », في Current Biology, vol. 22, no 22, 2012, ص. R960-R962 [النص الكامل, lien PMID, lien DOI (pages consultées le 12 mars 2015)]
- Created from PDB 1JDG نسخة محفوظة 22 سبتمبر 2008 على موقع واي باك مشين.
- "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation"، Biochemistry، 42 (30): 9221–6، أغسطس 2003، doi:10.1021/bi034593c، PMID 12885257.
- "Hydroxyl radicals and DNA base damage"، Mutation Research، 424 (1–2): 9–21، مارس 1999، doi:10.1016/S0027-5107(99)00004-4، PMID 10064846.
- "Oxidative decay of DNA"، The Journal of Biological Chemistry، 272 (32): 19633–6، أغسطس 1997، doi:10.1074/jbc.272.32.19633، PMID 9289489.
- "Regulation and mechanisms of mammalian double-strand break repair"، Oncogene، 22 (37): 5792–812، سبتمبر 2003، doi:10.1038/sj.onc.1206679، PMID 12947387.
- Johnson, George (28 ديسمبر 2010)، "Unearthing Prehistoric Tumors, and Debate"، نيويورك تايمز، مؤرشف من الأصل في 24 يونيو 2017،
If we lived long enough, sooner or later we all would get cancer.
- "The Preventable Causes of Cancer"، Molecular biology of the cell (ط. 4th)، New York: Garland Science، 2002، ISBN 0-8153-4072-9، مؤرشف من الأصل في 02 يناير 2016،
هناك كمية معينة غير قابلة للخفض متوقعة من السرطان بغض النظر عن الظروف: الطفرات لا يمكن تجنبها نهائيا لأنها نتائج لا مفر منها لحدود طبيعية أساسية في دقة تضاعف الدنا كما هو موضح في العنوان 5: إعادة تركيب الجينات. لو عاش الإنسان مدة كافية فلا مفر من أن إحدى خلاياه على الأقل ستتراكم لديها في النهاية طفرات كافية للتسبب بالسرطان.
- Kimura, Honoka؛ Suzuki, Aoi, المحررون (2008)، "Cancer and aging as consequences of un-repaired DNA damage"، New Research on DNA Damage، New York: Nova Science Publishers، ص. 1–47.، ISBN 978-1-60456-581-2، مؤرشف من الأصل في 25 أكتوبر 2014.
- "DNA damage, aging, and cancer"، The New England Journal of Medicine، 361 (15): 1475–85، أكتوبر 2009، doi:10.1056/NEJMra0804615، PMID 19812404.
- "A review and appraisal of the DNA damage theory of ageing"، Mutation Research، 728 (1–2): 12–22، 2011، doi:10.1016/j.mrrev.2011.05.001، PMID 21600302.
- "The genetic toxicology of acridines"، Mutation Research، 258 (2): 123–60، سبتمبر 1991، doi:10.1016/0165-1110(91)90006-H، PMID 1881402.
- "Mechanism of action in thalidomide teratogenesis"، Biochemical Pharmacology، 59 (12): 1489–99، يونيو 2000، doi:10.1016/S0006-2952(99)00388-3، PMID 10799645.
- "DNA modification by chemical carcinogens"، Pharmacology & Therapeutics، 28 (2): 237–72، 1985، doi:10.1016/0163-7258(85)90013-0، PMID 3936066.
- "Intercalators as anticancer drugs"، Current Pharmaceutical Design، 7 (17): 1745–80، نوفمبر 2001، doi:10.2174/1381612013397113، PMID 11562309.
- "The sequence of the human genome"، Science، 291 (5507): 1304–51، فبراير 2001، Bibcode:2001Sci...291.1304V، doi:10.1126/science.1058040، PMID 11181995.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "The bacterial nucleoid: a highly organized and dynamic structure"، Journal of Cellular Biochemistry، 96 (3): 506–21، أكتوبر 2005، doi:10.1002/jcb.20519، PMID 15988757.
- "Guide to the draft human genome"، Nature، 409 (6822): 824–6، فبراير 2001، Bibcode:2001Natur.409..824W، doi:10.1038/35057000، PMID 11236998.
- "The C-value enigma in plants and animals: a review of parallels and an appeal for partnership"، Annals of Botany، 95 (1): 133–46، يناير 2005، doi:10.1093/aob/mci009، PMID 15596463.
- "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project"، Nature، 447 (7146): 799–816، يونيو 2007، Bibcode:2007Natur.447..799B، doi:10.1038/nature05874، PMC 2212820، PMID 17571346.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - "The role of heterochromatin in centromere function"، Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences، 360 (1455): 569–79، مارس 2005، doi:10.1098/rstb.2004.1611، PMC 1569473، PMID 15905142.
- "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22"، Genome Research، 12 (2): 272–80، فبراير 2002، doi:10.1101/gr.207102، PMC 155275، PMID 11827946.
- "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution"، Journal of Molecular Biology، 318 (5): 1155–74، مايو 2002، doi:10.1016/S0022-2836(02)00109-2، PMID 12083509.
- "Replicative DNA polymerases"، Genome Biology، 2 (1): REVIEWS3002، 2001، doi:10.1186/gb-2001-2-1-reviews3002، PMC 150442، PMID 11178285.
- "Structure–function studies of the RNA polymerase II elongation complex"، Acta Crystallogr. D، 65 (Pt 2): 112–20، أفريل 2018، doi:10.1107/S0907444908039875، PMC 2631633، PMID 19171965.
{{استشهاد بدورية محكمة}}: تحقق من التاريخ في:|تاريخ=(مساعدة) - Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2007), Biochemistry (6 ed.), New York: WH Freeman & Co., ISBN 0-7167-6766-X. p 836
- Shu, Jian-Jun (2017)، "A new integrated symmetrical table for genetic codes"، BioSystems، 151: 21–26، doi:10.1016/j.biosystems.2016.11.004.
- Biology، McGraw hill education، ص. 249، ISBN 978-981-4581-85-1.
- Molecular Biology of the Cell (ط. 5th)، New York: WH Freeman، 2004، ص. 963.
- Acharya, PV (1971)، "The isolation and partial characterization of age-correlated oligo-deoxyribo-ribonucleotides with covalently linked aspartyl-glutamyl polypeptides."، Johns Hopkins medical journal. Supplement (1): 254–60، PMID 5055816.
- Bjorksten, J؛ Acharya, PVN؛ Ashman, S؛ Wetlaufer, DB (1971)، "Gerogenic fractions in the tritiated rat"، Journal of the American Geriatrics Society، 19 (7): 561–74، doi:10.1111/j.1532-5415.1971.tb02577.x، PMID 5106728.
- Browner, WS؛ Kahn, AJ؛ Ziv, E؛ Reiner, AP؛ Oshima, J؛ Cawthon, RM؛ Hsueh, WC؛ Cummings, SR. (2004)، "The genetics of human longevity"، Am J Med، 117 (11): 851–60، doi:10.1016/j.amjmed.2004.06.033، PMID 15589490.
- Tani, Katsuji؛ Nasu, Masao (2010)، "Roles of Extracellular DNA in Bacterial Ecosystems"، في Kikuchi, Yo؛ Rykova, Elena Y. (المحررون)، Extracellular Nucleic Acids، Springer، ص. 25–38، ISBN 978-3-642-12616-1.
- "Extracellular nucleic acids"، BioEssays، 29 (7): 654–67، يوليو 2007، doi:10.1002/bies.20604، PMID 17563084.
- "DNA as a nutrient: novel role for bacterial competence gene homologs"، Journal of Bacteriology، 183 (21): 6288–93، نوفمبر 2001، doi:10.1128/JB.183.21.6288-6293.2001، PMC 100116، PMID 11591672.
- "Extracellular DNA chelates cations and induces antibiotic resistance in Pseudomonas aeruginosa biofilms"، PLoS Pathogens، 4 (11): e1000213، نوفمبر 2008، doi:10.1371/journal.ppat.1000213، PMC 2581603، PMID 19023416.
- "A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm"، Molecular Microbiology، 77 (4): 815–29، أغسطس 2010، doi:10.1111/j.1365-2958.2010.07267.x، PMC 2962764، PMID 20598083.
- "Extracellular DNA required for bacterial biofilm formation"، Science، 295 (5559): 1487، فبراير 2002، doi:10.1126/science.295.5559.1487، PMID 11859186.
- "DNA builds and strengthens the extracellular matrix in Myxococcus xanthus biofilms by interacting with exopolysaccharides"، PLoS One، 7 (12): e51905، 2012، Bibcode:2012PLoSO...751905H، doi:10.1371/journal.pone.0051905، PMC 3530553، PMID 23300576.
- "Recent advances in the prenatal interrogation of the human fetal genome"، Trends in Genetics، 29 (2): 84–91، فبراير 2013، doi:10.1016/j.tig.2012.10.013، PMC 4378900، PMID 23158400.
- "Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome"، Cellular and Molecular Life Sciences، 54 (12): 1350–64، ديسمبر 1998، doi:10.1007/s000180050259، PMID 9893710.
- "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin"، Molecular Microbiology، 56 (4): 858–70، مايو 2005، doi:10.1111/j.1365-2958.2005.04598.x، PMID 15853876.
- "Crystal structure of the nucleosome core particle at 2.8 A resolution"، Nature، 389 (6648): 251–60، سبتمبر 1997، Bibcode:1997Natur.389..251L، doi:10.1038/38444، PMID 9305837.
- "Translating the histone code" (PDF)، Science، 293 (5532): 1074–80، أغسطس 2001، doi:10.1126/science.1063127، PMID 11498575، مؤرشف من الأصل (PDF) في 08 أغسطس 2017.
- "Nucleosome assembly and remodeling"، Current Topics in Microbiology and Immunology، Current Topics in Microbiology and Immunology، 274: 1–22، 2003، doi:10.1007/978-3-642-55747-7_1، ISBN 978-3-540-44208-0، PMID 12596902.
- "HMG1 and 2: architectural DNA-binding proteins"، Biochemical Society Transactions، 29 (Pt 4): 395–401، أغسطس 2001، doi:10.1042/BST0290395، PMID 11497996.
- "HMG domain proteins: architectural elements in the assembly of nucleoprotein structures"، Trends in Genetics، 10 (3): 94–100، مارس 1994، doi:10.1016/0168-9525(94)90232-1، PMID 8178371.
- Created from PDB 1LMB نسخة محفوظة 6 January 2008 على موقع واي باك مشين.
- "Replication protein A (RPA): the eukaryotic SSB"، Critical Reviews in Biochemistry and Molecular Biology، 34 (3): 141–80، 1999، doi:10.1080/10409239991209255، PMID 10473346.
- "Mediator of transcriptional regulation"، Annual Review of Biochemistry، 69: 729–49، 2000، doi:10.1146/annurev.biochem.69.1.729، PMID 10966474.
- "Biological control through regulated transcriptional coactivators"، Cell، 119 (2): 157–67، أكتوبر 2004، doi:10.1016/j.cell.2004.09.037، PMID 15479634.
- "A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells"، Proceedings of the National Academy of Sciences of the United States of America، 100 (14): 8164–9، يوليو 2003، Bibcode:2003PNAS..100.8164L، doi:10.1073/pnas.1332764100، PMC 166200، PMID 12808131.
- Created from PDB 1RVA نسخة محفوظة 6 January 2008 على موقع واي باك مشين.
- "Biology of DNA restriction"، Microbiological Reviews، 57 (2): 434–50، يونيو 1993، PMC 372918، PMID 8336674.
- "Structural and mechanistic conservation in DNA ligases"، Nucleic Acids Research، 28 (21): 4051–8، نوفمبر 2000، doi:10.1093/nar/28.21.4051، PMC 113121، PMID 11058099.
- "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism"، Biochemical Society Transactions، 33 (Pt 6): 1465–70، ديسمبر 2005، doi:10.1042/BST20051465، PMID 16246147.
- "Unraveling DNA helicases. Motif, structure, mechanism and function"، European Journal of Biochemistry، 271 (10): 1849–63، مايو 2004، doi:10.1111/j.1432-1033.2004.04094.x، PMID 15128295.
- "Polymerase structures and function: variations on a theme?"، Journal of Bacteriology، 177 (22): 6321–9، نوفمبر 1995، doi:10.1128/jb.177.22.6321-6329.1995، PMC 177480، PMID 7592405.
- "Eukaryotic DNA polymerases"، Annual Review of Biochemistry، 71: 133–63، 2002، doi:10.1146/annurev.biochem.71.090501.150041، PMID 12045093.
- "Cellular DNA replicases: components and dynamics at the replication fork"، Annual Review of Biochemistry، 74: 283–315، 2005، doi:10.1146/annurev.biochem.73.011303.073859، PMID 15952889.
- "The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention"، FASEB Journal، 8 (8): 497–503، مايو 1994، PMID 7514143.
- "Multi-protein complexes in eukaryotic gene transcription"، Plant Molecular Biology، 50 (6): 925–47، ديسمبر 2002، doi:10.1023/A:1021258713850، PMID 12516863.
- "Homology and enzymatic requirements of microhomology-dependent alternative end joining"، Cell Death & Disease، 6 (3): e1697، مارس 2015، doi:10.1038/cddis.2015.58، PMC 4385936، PMID 25789972.
- "Repair of naturally occurring mismatches can induce mutations in flanking DNA"، eLife، 3: e02001، أبريل 2014، doi:10.7554/elife.02001، PMC 3999860، PMID 24843013.
- "Error-Prone Repair of DNA Double-Strand Breaks"، Journal of Cellular Physiology، 231 (1): 15–24، يناير 2016، doi:10.1002/jcp.25053، PMC 4586358، PMID 26033759.
- "The molecular biology of cancer"، Molecular Aspects of Medicine، إلزيفير، 21 (6): 167–223، ديسمبر 2000، doi:10.1016/S0098-2997(00)00007-8، PMID 11173079.
- "Pesticide resistance via transposition-mediated adaptive gene truncation in Drosophila"، Science، الجمعية الأمريكية لتقدم العلوم، 309 (5735): 764–7، يوليو 2005، Bibcode:2005Sci...309..764A، doi:10.1126/science.1112699، PMID 16051794.
- "Shaping bacterial genomes with integrative and conjugative elements"، Research in Microbiology، Elsevier، 155 (5): 376–86، يونيو 2004، doi:10.1016/j.resmic.2004.01.012، PMID 15207870.
- "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila"، Proceedings of the National Academy of Sciences of the United States of America، الأكاديمية الوطنية للعلوم، 104 (16): 6504–10، أبريل 2007، Bibcode:2007PNAS..104.6504S، doi:10.1073/pnas.0701572104، PMC 1871816، PMID 17409186.
- Created from PDB 1M6G نسخة محفوظة 10 January 2010 على موقع واي باك مشين.
- "Chromosome territories, nuclear architecture and gene regulation in mammalian cells"، Nature Reviews. Genetics، 2 (4): 292–301، أبريل 2001، doi:10.1038/35066075، PMID 11283701.
- "An integrated view of protein evolution"، Nature Reviews. Genetics، 7 (5): 337–48، مايو 2006، doi:10.1038/nrg1838، PMID 16619049.
- "The role of double-strand break repair - insights from human genetics"، Nature Reviews. Genetics، 7 (1): 45–54، يناير 2006، doi:10.1038/nrg1746، PMID 16369571.
- "Mammalian Rad51 protein: a RecA homologue with pleiotropic functions"، Biochimie، 79 (9–10): 587–92، أكتوبر 1997، doi:10.1016/S0300-9084(97)82007-X، PMID 9466696.
- "Clarifying the mechanics of DNA strand exchange in meiotic recombination"، Nature، 442 (7099): 153–8، يوليو 2006، Bibcode:2006Natur.442..153N، doi:10.1038/nature04885، PMC 5607947، PMID 16838012.
- "The RuvABC resolvasome"، European Journal of Biochemistry، 269 (22): 5492–501، نوفمبر 2002، doi:10.1046/j.1432-1033.2002.03250.x، PMID 12423347.
- "Transposase" (PDF)، 2006، اطلع عليه بتاريخ 23 mars 2015.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغ:|citation=(مساعدة)، الوسيط غير المعروف|month=تم تجاهله (مساعدة) نسخة محفوظة 24 سبتمبر 2015 على موقع واي باك مشين. دُوِي:10.2210/rcsb_pdb/mom_2006_12 - Phillip Sanmiguel et Jeffrey L. Bennetzen, « Evidence that a Recent Increase in Maize Genome Size was Caused by the Massive Amplification of Intergene Retrotransposons », في Annals of Botany, vol. 82, no Supplement A, décembre 1998, ص. 37-44 [النص الكامل, lien DOI (pages consultées le 23 mars 2015)]
- Wanlong Li,Peng Zhang, John P. Fellers, Bernd Friebe et Bikram S. Gill, « Sequence composition, organization, and evolution of the core Triticeae genome », في The Plant Journal, vol. 40, no 4, novembre 2004, ص. 500-511 [النص الكامل, lien PMID, lien DOI (pages consultées le 23 mars 2015)]
- International Human Genome Sequencing Consortium, « Initial sequencing and analysis of the human genome », في Nature, vol. 409, no 6822, 1الأول {{{1}}} février 2001, ص. 860-921 [النص الكامل, lien PMID, lien DOI (pages consultées le 23 mars 2015)]
- Etienne Bucher, Jon Reinders et Marie Mirouze, « Epigenetic control of transposon transcription and mobility in Arabidopsis », في Current Opinion in Plant Biology, vol. 15, no 6, novembre 2012, ص. 503-510 [النص الكامل, lien PMID, lien DOI (pages consultées le 23 mars 2015)]
- David R. Edgel Marlene Belfort et David A. Shub, « Barriers to Intron Promiscuity in Bacteria », في Journal of Bacteriology, vol. 182, no 19, octobre 2000, ص. 5281-5289 [النص الكامل, lien PMID, lien DOI (pages consultées le 24 mars 2015)]
- Linus Sandegren et Britt-Marie Sjöberg, « Distribution, sequence homology, and homing of group I introns among T-even-like bacteriophages: evidence for recent transfer of old introns », في Journal of Biological Chemistry, vol. 279, no 21, 21 mai 2004, ص. 22218-22227 [النص الكامل, lien PMID, lien DOI (pages consultées le 24 mars 2015)]
- Richard P. Bonocora et David A. Shub, « A Self-Splicing Group I Intron in DNA Polymerase Genes of T7-Like Bacteriophages », في Journal of Bacteriology, vol. 186, no 23, décembre 2004, ص. 8153-8155 [النص الكامل, lien PMID, lien DOI (pages consultées le 24 mars 2015)]
- Chia-Ni Lee, Juey-Wen Lin, Shu-Fen Weng et Yi-Hsiung Tseng, « Genomic Characterization of the Intron-Containing T7-Like Phage phiL7 of Xanthomonas campestris », في Applied and Environmental Microbiology, vol. 75, no 24, décembre 2009, ص. 7828-7837 [النص الكامل, lien PMID, lien DOI (pages consultées le 24 mars 2015)]
- Keeling, P. J., & Palmer, J.D. (أغسطس 2008)، "Horizontal gene transfer in eukaryotic evolution"، Nature Reviews Genetics، 9 (8): 605–618، doi:10.1038/nrg2386، PMID 18591983، مؤرشف من الأصل في 22 أبريل 2016.
{{استشهاد بدورية محكمة}}: صيانة CS1: أسماء متعددة: قائمة المؤلفون (link) - Gyles, C.؛ Boerlin, P. (مارس 2014)، "Horizontally transferred genetic elements and their role in pathogenesis of bacterial disease"، Veterinary Pathology، 51 (2): 328–340، doi:10.1177/0300985813511131، PMID 24318976، مؤرشف من الأصل في 12 أغسطس 2016.
- Vaux, Felix؛ Trewick, Steven A.؛ Morgan-Richards, Mary (2017)، "Speciation through the looking-glass"، Biological Journal of the Linnean Society، 120 (2): 480–488، doi:10.1111/bij.12872.
- OECD, Safety Assessment of Transgenic Organisms, Volume 4: OECD Consensus Documents, 2010, pp.171-174
- "In situ transfer of antibiotic resistance genes from transgenic (transplastomic) tobacco plants to bacteria"، Appl. Environ. Microbiol.، 68 (7): 3345–51، يوليو 2002، doi:10.1128/aem.68.7.3345-3351.2002، PMC 126776، PMID 12089013.
- "Horizontal gene transfer in prokaryotes: quantification and classification"، Annu. Rev. Microbiol.، 55 (1): 709–42، 2001، doi:10.1146/annurev.micro.55.1.709، PMID 11544372.
- Nielsen KM (1998)، "Barriers to horizontal gene transfer by natural transformation in soil bacteria"، APMIS Suppl.، 84: 77–84، PMID 9850687.
- "Evidence for interspecies gene transfer in the evolution of 2,4-dichlorophenoxyacetic acid degraders"، Appl. Environ. Microbiol.، 64 (10): 4089–92، أكتوبر 1998، PMC 106609، PMID 9758850.
- Keen, E. C. (ديسمبر 2012)، "Paradigms of pathogenesis: Targeting the mobile genetic elements of disease"، Frontiers in Cellular and Infection Microbiology، 2: 161، doi:10.3389/fcimb.2012.00161، PMC 3522046، PMID 23248780.
- "Transfer of broad-host-range antibiotic resistance plasmids in soil microcosms"، Curr. Microbiol.، 28 (4): 209–215، 1994، doi:10.1007/BF01575963.
- Varga M, Kuntova L, Pantucek R, Maslanova I, Ruzickova V, Doskar J (2012)، "Efficient transfer of antibiotic resistance plasmids by transduction within methicillin-resistant Staphylococcus aureus USA300 clone"، FEMS Microbiol. Lett.، 332 (2): 146–152، doi:10.1111/j.1574-6968.2012.02589.x، PMID 22553940.
- Varga M, Pantucek R, Ruzickova V, Doskar J (2016)، "Molecular characterization of a new efficiently transducing bacteriophage identified in meticillin-resistant Staphylococcus aureus"، Journal of General Virology، 97 (1): 258–268، doi:10.1099/jgv.0.000329، PMID 26537974.
- Kubyshkin, V.؛ Acevedo-Rocha, C. G.؛ Budisa, N. (2017)، "On universal coding events in protein biogenesis"، Biosystems، doi:10.1016/j.biosystems.2017.10.004.
- Lin Edwards (4 أكتوبر 2010)، "Horizontal gene transfer in microbes much more frequent than previously thought"، PhysOrg.com، مؤرشف من الأصل في 15 سبتمبر 2011، اطلع عليه بتاريخ 06 يناير 2012.
- Carrie Arnold (18 أبريل 2011)، "To Share and Share Alike: Bacteria swap genes with their neighbors more frequently than researchers have realized"، Scientific American، مؤرشف من الأصل في 02 نوفمبر 2012، اطلع عليه بتاريخ 06 يناير 2012.
- "The antiquity of RNA-based evolution"، Nature، 418 (6894): 214–21، يوليو 2002، Bibcode:2002Natur.418..214J، doi:10.1038/418214a، PMID 12110897.
- "Prebiotic chemistry and the origin of the RNA world"، Critical Reviews in Biochemistry and Molecular Biology، 39 (2): 99–123، 2004، CiteSeerX 10.1.1.537.7679، doi:10.1080/10409230490460765، PMID 15217990.
- "Ribozymes. Making copies in the RNA world"، Science، 292 (5520): 1278، مايو 2001، doi:10.1126/science.292.5520.1278a، PMID 11360970.
- "What is the optimum size for the genetic alphabet?"، Proceedings of the National Academy of Sciences of the United States of America، 89 (7): 2614–8، أبريل 1992، Bibcode:1992PNAS...89.2614S، doi:10.1073/pnas.89.7.2614، PMC 48712، PMID 1372984.
- "Instability and decay of the primary structure of DNA"، Nature، 362 (6422): 709–15، أبريل 1993، Bibcode:1993Natur.362..709L، doi:10.1038/362709a0، PMID 8469282.
- "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal"، Nature، 407 (6806): 897–900، أكتوبر 2000، Bibcode:2000Natur.407..897V، doi:10.1038/35038060، PMID 11057666.
- "Geologically ancient DNA: fact or artefact?"، Trends in Microbiology، 13 (5): 212–20، مايو 2005، doi:10.1016/j.tim.2005.03.010، PMID 15866038.
- "Curiously modern DNA for a "250 million-year-old" bacterium"، Journal of Molecular Evolution، 54 (1): 134–7، يناير 2002، Bibcode:2002JMolE..54..134N، doi:10.1007/s00239-001-0025-x، PMID 11734907.
- "Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases"، Proceedings of the National Academy of Sciences of the United States of America، 108 (34): 13995–8، أغسطس 2011، Bibcode:2011PNAS..10813995C، doi:10.1073/pnas.1106493108، PMC 3161613، PMID 21836052.
- Steigerwald, John (08 أغسطس 2011)، "NASA Researchers: DNA Building Blocks Can Be Made in Space"، ناسا، مؤرشف من الأصل في 23 يونيو 2015، اطلع عليه بتاريخ 10 أغسطس 2011.
- ScienceDaily Staff (09 أغسطس 2011)، "DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests"، علم يوميا، مؤرشف من الأصل في 05 سبتمبر 2011، اطلع عليه بتاريخ 09 أغسطس 2011.
- Marlaire, Ruth (03 مارس 2015)، "NASA Ames Reproduces the Building Blocks of Life in Laboratory"، ناسا، مؤرشف من الأصل في 05 مارس 2015، اطلع عليه بتاريخ 05 مارس 2015.
- Yukihisa Katsumoto, Masako Fukuchi-Mizutani, Yuko Fukui, Filippa Brugliera, Timothy A. Holton, Mirko Karan, Noriko Nakamura, Keiko Yonekura-Sakakibara, Junichi Togami, Alix Pigeaire, Guo-Qing Tao, Narender S. Nehra, Chin-Yi Lu, Barry K. Dyson, Shinzo Tsuda, Toshihiko Ashikari, Takaaki Kusumi, John G. Mason et Yoshikazu Tanaka, « Engineering of the Rose Flavonoid Biosynthetic Pathway Successfully Generated Blue-Hued Flowers Accumulating Delphinidin », في Plant & Cell Physiology, vol. 48, no 11, novembre 2007, ص. 1589-1600 [النص الكامل, lien PMID, lien DOI (pages consultées le 2 avril 2015)]
- "Plant gene replacement results in the world's only blue rose"، 2005، اطلع عليه بتاريخ 2 avril 2015.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغ:|citation=(مساعدة)، الوسيط غير المعروف|month=تم تجاهله (مساعدة) - "Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells"، Cell، 9 (4 PT 2): 695–705، ديسمبر 1976، doi:10.1016/0092-8674(76)90133-1، PMID 189942.
- "Transgenic animal models in biomedical research"، Methods in Molecular Biology، 360: 163–202، 2007، doi:10.1385/1-59745-165-7:163، ISBN 1-59745-165-7، PMID 17172731.
- "Multigene engineering: dawn of an exciting new era in biotechnology"، Current Opinion in Biotechnology، 13 (2): 136–41، أبريل 2002، doi:10.1016/S0958-1669(02)00297-5، PMC 3481857، PMID 11950565.
- "Plant biotechnology in agriculture"، Biochimie، 84 (11): 1105–10، نوفمبر 2002، doi:10.1016/S0300-9084(02)00013-5، PMID 12595138.
- "GM traits list"، International Service for the Acquisition of Agri-Biotech Applications، مؤرشف من الأصل في 08 يونيو 2020.
- "ISAAA Brief 43-2011: Executive Summary"، International Service for the Acquisition of Agri-Biotech Applications، مؤرشف من الأصل في 13 يونيو 2020.
- Connor, Steve (02 نوفمبر 2007)، "The mouse that shook the world"، The Independent، مؤرشف من الأصل في 08 مارس 2018.
- "A DNA enzyme that cleaves RNA"، Chemistry & Biology، 1 (4): 223–9، ديسمبر 1994، doi:10.1016/1074-5521(94)90014-0، PMID 9383394.
- "DNA-catalyzed sequence-specific hydrolysis of DNA"، Nature Chemical Biology، 5 (10): 718–20، أكتوبر 2009، doi:10.1038/nchembio.201، PMC 2746877، PMID 19684594.
- "In vitro selection of self-cleaving DNAs"، Chemistry & Biology، 3 (12): 1039–46، ديسمبر 1996، doi:10.1016/S1074-5521(96)90170-2، PMID 9000012.
- "In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing"، Proceedings of the National Academy of Sciences of the United States of America، 112 (19): 5903–8، مايو 2015، Bibcode:2015PNAS..112.5903T، doi:10.1073/pnas.1420361112، PMC 4434688، PMID 25918425.
- Curtis, Caitlin؛ Hereward, James (29 أغسطس 2017)، "From the crime scene to the courtroom: the journey of a DNA sample"، The Conversation، مؤرشف من الأصل في 22 أكتوبر 2017، اطلع عليه بتاريخ 22 أكتوبر 2017.
- "Likelihood ratios for DNA identification"، Proceedings of the National Academy of Sciences of the United States of America، 91 (13): 6007–11، يونيو 1994، Bibcode:1994PNAS...91.6007C، doi:10.1073/pnas.91.13.6007، PMC 44126، PMID 8016106.
- "Interpreting DNA mixtures"، Journal of Forensic Sciences، 42 (2): 213–22، مارس 1997، PMID 9068179.
- "Individual-specific 'fingerprints' of human DNA"، Nature، 316 (6023): 76–9، 1985، Bibcode:1985Natur.316...76J، doi:10.1038/316076a0، PMID 2989708.
- Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspect Forensic Science Service Accessed 23 December 2006 نسخة محفوظة 18 مايو 2016 على موقع واي باك مشين.
- "Paternity Blood Tests That Work Early in a Pregnancy" New York Times June 20, 2012 نسخة محفوظة 24 June 2017 على موقع واي باك مشين.
- Baldi, Pierre؛ Brunak, Soren (2001)، Bioinformatics: The Machine Learning Approach، MIT Press، ISBN 978-0-262-02506-5، OCLC 45951728.
- Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. مطبعة جامعة كامبريج, 15 January 1997. (ردمك 978-0-521-58519-4).
- "Phylogenomic inference of protein molecular function: advances and challenges"، Bioinformatics، 20 (2): 170–9، يناير 2004، doi:10.1093/bioinformatics/bth021، PMID 14734307.
- Bioinformatics: Sequence and Genome Analysis (ط. 2nd)، Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press، 2004، ISBN 0-87969-712-1، OCLC 55106399.
- "Folding DNA to create nanoscale shapes and patterns"، Nature، 440 (7082): 297–302، مارس 2006، Bibcode:2006Natur.440..297R، doi:10.1038/nature04586، PMID 16541064.
- "Self-assembly of a nanoscale DNA box with a controllable lid"، Nature، 459 (7243): 73–6، مايو 2009، Bibcode:2009Natur.459...73A، doi:10.1038/nature07971، PMID 19424153.
- "DNA nanotechnology: a nanomachine goes live"، Nature Nanotechnology، 4 (5): 281–2، مايو 2009، Bibcode:2009NatNa...4..281I، doi:10.1038/nnano.2009.101، PMID 19421208.
- "Assembling materials with DNA as the guide"، Science، 321 (5897): 1795–9، سبتمبر 2008، Bibcode:2008Sci...321.1795A، doi:10.1126/science.1154533، PMID 18818351.
- "Programming DNA circuits"، 2015، اطلع عليه بتاريخ 28 mars 2015.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغs:|month=و|citation=(مساعدة) - Yaniv Amir, Eldad Ben-Ishay, Daniel Levner, Shmulik Ittah, Almogit Abu-Horowitz et Ido Bachelet, « Universal computing by DNA origami robots in a living animal », في Nature Nanotechnology, vol. 9, no 5, mai 2014, ص. 353-357 [النص الكامل, lien PMID, lien DOI (pages consultées le 28 mars 2015)]
- Lulu Qian, Erik Winfree et Jehoshua Bruck, « Neural network computation with DNA strand displacement cascades », في Nature, vol. 475, no 7356, 21 juillet 2011, ص. 368-372 [النص الكامل, lien PMID, lien DOI (pages consultées le 28 mars 2015)]
- "Dating branches on the tree of life using DNA"، Genome Biology، 3 (1): REVIEWS0001، 2002، doi:10.1046/j.1525-142X.1999.99010.x، PMC 150454، PMID 11806830.
- Mannis van Oven et Manfred Kayser, « Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation », في Human Mutation, vol. 30, no 2, février 2009, ص. E386-E394 [النص الكامل, lien PMID, lien DOI (pages consultées le 27 mars 2015)]
- (بالإنجليزية) Francesca D. Ciccarelli, Tobias Doerks, Christian von Mering, Christopher J. Creevey, Berend Snel et Peer Bork, « Toward automatic reconstruction of a highly resolved tree of life », في Science, vol. 311, no 5765, 2006, ص. 1283–1287 [النص الكامل, lien PMID, lien DOI]
- "Human mtDNA Migrations" (PDF)، 2002، اطلع عليه بتاريخ 27 mars 2015.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغs:|month=و|citation=(مساعدة)، روابط خارجية في|site= - George M. Church, Yuan Gao et Sriram Kosuri, « Next-Generation Digital Information Storage in DNA », في Science, vol. 337, no 6102, 28 septembre 2012, ص. 1628 [النص الكامل, lien PMID, lien DOI (pages consultées le 28 mars 2015)]
- "Researchers Store Computer Operating System and Short Movie on DNA. New Coding Strategy Maximizes Data-Storage Capacity of DNA Molecules."، 2017، اطلع عليه بتاريخ 26 janvier 2018.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغ:|citation=(مساعدة)، الوسيط غير المعروف|month=تم تجاهله (مساعدة) نسخة محفوظة 14 مارس 2017 على موقع واي باك مشين. - Nick Goldman, Paul Bertone, Siyuan Chen, Christophe Dessimoz, Emily M. LeProust, Botond Sipos et Ewan Birney, « Towards practical, high-capacity, low-maintenance information storage in synthesized DNA », في Nature, vol. 494, no 7435, 7 février 2013, ص. 77-80 [النص الكامل, lien PMID, lien DOI (pages consultées le 28 mars 2015)]
- "Towards practical, high-capacity, low-maintenance information storage in synthesized DNA – Supplemental Information 1: Human-Designed Information Stored in DNA" (PDF)، 2013، اطلع عليه بتاريخ 28 mars 2015.
{{استشهاد ويب}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة)، يحتوي الاستشهاد على وسيط غير معروف وفارغ:|citation=(مساعدة)، الوسيط غير المعروف|month=تم تجاهله (مساعدة) We recovered 757,051 bytes of information from 337 pg of DNA, giving an information storage density of 2,2 PB/g (= 757 051 / 337×10−12). We note that this information density is enough to store the US National Archives and Records Administration’s Electronic Records Archives’ 2011 total of ~100 TB in < 0.05 g of DNA, the Internet Archive Wayback Machines’s 2 PB archive of web sites in ~1 g of DNA, and CERN’s 80 PB CASTOR system for LHC data in ~35 g of DNA.(النص الأصلي){{{2}}} - Robert N. Grass, Reinhard Heckel, Michela Puddu, Daniela Paunescu et Wendelin J. Stark, « Robust Chemical Preservation of Digital Information on DNA in Silica with Error-Correcting Codes », في Angewandte Chemie International Edition, vol. 54, no 8, 16 février 2015, ص. 2552-2555 [النص الكامل, lien DOI (pages consultées le 28 mars 2015)]
- Jerome Bonnet, Pakpoom Subsoontorn et Drew Endy, « Rewritable digital data storage in live cells via engineered control of recombination directionality », في Proceedings of the National Academy of Sciences of the United States of America, vol. 109, no 23, 5 juin 2012, ص. 8884-8889 [النص الكامل, lien PMID, lien DOI (pages consultées le 28 mars 2015)]
- Miescher, Friedrich (1871) "Ueber die chemische Zusammensetzung der Eiterzellen" نسخة محفوظة 22 October 2015 على موقع واي باك مشين. (On the chemical composition of pus cells), Medicinisch-chemische Untersuchungen, 4 : 441–460. From p. 456: نسخة محفوظة 25 September 2015 على موقع واي باك مشين. "Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend." (Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ("Nuclein").)
- "Discovering DNA: Friedrich Miescher and the early years of nucleic acid research"، Human Genetics، 122 (6): 565–81، يناير 2008، doi:10.1007/s00439-007-0433-0، PMID 17901982.
- See:
- Albrect Kossel (1879) "Ueber Nucleïn der Hefe" نسخة محفوظة 17 November 2017 على موقع واي باك مشين. (On nuclein in yeast) Zeitschrift für physiologische Chemie, 3 : 284–291.
- Albrect Kossel (1880) "Ueber Nucleïn der Hefe II" نسخة محفوظة 17 November 2017 على موقع واي باك مشين. (On nuclein in yeast, Part 2) Zeitschrift für physiologische Chemie, 4 : 290–295.
- Albrect Kossel (1881) "Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich" نسخة محفوظة 17 November 2017 على موقع واي باك مشين. (On the distribution of hypoxanthins in the animal and plant kingdoms) Zeitschrift für physiologische Chemie, 5 : 267–271.
- Albrect Kossel, Untersuchungen über die Nucleine und ihre Spaltungsprodukte [Investigations into nuclein and its cleavage products] (Strassburg, Germany: K.J. Trübne, 1881), 19 pages.
- Albrect Kossel (1882) "Ueber Xanthin und Hypoxanthin" نسخة محفوظة 17 November 2017 على موقع واي باك مشين. (On xanthin and hypoxanthin), Zeitschrift für physiologische Chemie, 6 : 422–431.
- Albrect Kossel (1883) "Zur Chemie des Zellkerns" نسخة محفوظة 17 November 2017 على موقع واي باك مشين. (On the chemistry of the cell nucleus), Zeitschrift für physiologische Chemie, 7 : 7–22.
- Albrect Kossel (1886) "Weitere Beiträge zur Chemie des Zellkerns" (Further contributions to the chemistry of the cell nucleus), Zeitschrift für Physiologische Chemie, 10 : 248–264. Available on-line at: Max Planck Institute for the History of Science, Berlin, Germany نسخة محفوظة 14 July 2014 على موقع واي باك مشين.. On p. 264, Kossel remarked presciently: "Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge." (The study of the quantitative relations of the four nitrogenous bases — [and] of the dependence of their quantity on the physiological states of the cell — promises important insights into the fundamental physiological-chemical processes.)
- "Albrecht Kossel, a biographical sketch"، The Yale Journal of Biology and Medicine، المركز الوطني لمعلومات التقانة الحيوية، 26 (1): 80–97، سبتمبر 1953، PMC 2599350، PMID 13103145.
- "The structure of yeast nucleic acid"، J Biol Chem، 40 (2): 415–24، 1919.
- Koltsov proposed that a cell's genetic information was encoded in a long chain of amino acids. See:
- Н. К. Кольцов, "Физико-химические основы морфологии" (The physical-chemical basis of morphology) – speech given at the 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists at Leningrad, U.S.S.R., 12 December 1927.
- Reprinted in: Успехи экспериментальной биологии (Advances in Experimental Biology), series B, 7 (1) : ?-? (1928).
- Reprinted in German as: Nikolaj K. Koltzoff (1928) "Physikalisch-chemische Grundlagen der Morphologie" (The physical-chemical basis of morphology), Biologisches Zentralblatt, 48 (6) : 345–369.
- In 1934, Koltsov contended that the proteins that contain a cell's genetic information replicate. See: N. K. Koltzoff (5 October 1934) "The structure of the chromosomes in the salivary glands of Drosophila," Science, 80 (2075) : 312–313. From page 313: "I think that the size of the chromosomes in the salivary glands [of Drosophila] is determined through the multiplication of genonemes. By this term I designate the axial thread of the chromosome, in which the geneticists locate the linear combination of genes; … In the normal chromosome there is usually only one genoneme; before cell-division this genoneme has become divided into two strands."
- "The consequences of political dictatorship for Russian science"، Nature Reviews. Genetics، 2 (9): 723–9، سبتمبر 2001، doi:10.1038/35088598، PMID 11533721.
- "The Significance of Pneumococcal Types"، The Journal of Hygiene، 27 (2): 113–59، يناير 1928، doi:10.1017/S0022172400031879، PMC 2167760، PMID 20474956.
- "Bacterial gene transfer by natural genetic transformation in the environment"، Microbiological Reviews، 58 (3): 563–602، سبتمبر 1994، PMC 372978، PMID 7968924.
- See:
- "Some recent developments in the X-ray study of proteins and related structures" (PDF)، Cold Spring Harbor Symposia on Quantitative Biology، 6: 109–121، 1938، مؤرشف من الأصل (PDF) في 14 يوليو 2014.
- "X-ray studies of nucleic acids"، Symposia of the Society for Experimental Biology (1): 66–76، 1947، PMID 20257017، مؤرشف من الأصل في 05 يوليو 2014.
- "Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III"، The Journal of Experimental Medicine، 79 (2): 137–58، فبراير 1944، doi:10.1084/jem.79.2.137، PMC 2135445، PMID 19871359.
- "Independent functions of viral protein and nucleic acid in growth of bacteriophage"، The Journal of General Physiology، 36 (1): 39–56، مايو 1952، doi:10.1085/jgp.36.1.39، PMC 2147348، PMID 12981234.
- The B-DNA X-ray pattern on the right of this linked image نسخة محفوظة 25 May 2012 at Archive.is was obtained by روزاليند فرانكلين and ريموند غوسلين in May 1952 at high hydration levels of DNA and it has been labeled as "Photo 51"
- Regis, Ed (2009) What Is Life?: investigating the nature of life in the age of synthetic biology. Oxford: دار نشر جامعة أكسفورد (ردمك 0-19-538341-9); p. 52
- Pauling, L.; Corey, R. B. (1953). "A proposed structure for the nucleic acids." Proc. Natl. Acad. Sci. 39 (February 1953): 84-97. link. نسخة محفوظة 11 ديسمبر 2015 على موقع واي باك مشين.
- Nature Archives Double Helix of DNA: 50 Years نسخة محفوظة 5 April 2015 على موقع واي باك مشين.
- "Original X-ray diffraction image"، Oregon State Library، مؤرشف من الأصل في 30 يناير 2009، اطلع عليه بتاريخ 06 فبراير 2011.
- "The Nobel Prize in Physiology or Medicine 1962"، Nobelprize.org، مؤرشف من الأصل في 27 يوليو 2018.
- "The double helix and the 'wronged heroine'" (PDF)، Nature، 421 (6921): 407–8، يناير 2003، Bibcode:2003Natur.421..407M، doi:10.1038/nature01399، PMID 12540909، مؤرشف من الأصل (PDF) في 17 أكتوبر 2016.
- Crick, F.H.C. (1955)، A Note for the RNA Tie Club (PDF) (Speech)، Cambridge, England، مؤرشف من الأصل (PDF) في 01 أكتوبر 2008.
- "THE REPLICATION OF DNA IN ESCHERICHIA COLI"، Proceedings of the National Academy of Sciences of the United States of America، 44 (7): 671–82، يوليو 1958، Bibcode:1958PNAS...44..671M، doi:10.1073/pnas.44.7.671، PMC 528642، PMID 16590258.
- "The Nobel Prize in Physiology or Medicine 1968"، Nobelprize.org، مؤرشف من الأصل في 12 يونيو 2018.
- "Discovery of DNA structure and function: Watson and Crick."، Nature Education، 1 (1): 100، 2008.
- "ترجمة (Deoxyribonucleic acid)"، معجم مرعشي الطبي الكبير، مؤرشف من الأصل في 09 يونيو 2019، اطلع عليه بتاريخ 13 يونيو 2018.
- "ترجمة (Deoxyribonucleic acid)"، المعجم الطبي الموحد، مؤرشف من الأصل في 09 يونيو 2019، اطلع عليه بتاريخ 13 يونيو 2018.
- "ترجمة (Deoxyribonucleic acid)"، المعجم الطبي الموحد، مؤرشف من الأصل في 09 يونيو 2019، اطلع عليه بتاريخ 13 يونيو 2018.
- "ترجمة و معنى deoxyribonucleic acid بالعربي في قاموس المعاني. قاموس عربي انجليزي مصطلحات صفحة 1"، www.almaany.com، مؤرشف من الأصل في 09 يونيو 2019، اطلع عليه بتاريخ 13 يونيو 2018.
- "Al-Qamoos القاموس - English Arabic dictionary / قاموس إنجليزي عربي"، www.alqamoos.org، مؤرشف من الأصل في 09 يونيو 2019، اطلع عليه بتاريخ 13 يونيو 2018.
مصادر إضافية
- Berry, Andrew؛ Watson, James (2003)، DNA: the secret of life، New York: Alfred A. Knopf، ISBN 0-375-41546-7.
- Calladine, Chris R.؛ Drew, Horace R.؛ Luisi, Ben F.؛ Travers, Andrew A. (2003)، Understanding DNA: the molecule & how it works، Amsterdam: Elsevier Academic Press، ISBN 0-12-155089-3.
- Carina, Dennis؛ Clayton, Julie (2003)، 50 years of DNA، Basingstoke: Palgrave Macmillan، ISBN 1-4039-1479-6.
- Judson, Horace F (1979)، The Eighth Day of Creation: Makers of the Revolution in Biology (ط. 2nd)، Cold Spring Harbor Laboratory Press، ISBN 0-671-22540-5.
- Olby, Robert C. (1994)، The path to the double helix: the discovery of DNA، New York: Dover Publications، ISBN 0-486-68117-3., first published in October 1974 by MacMillan, with foreword by Francis Crick; the definitive DNA textbook, revised in 1994 with a 9-page postscript
- Micklas, David (2003)، DNA Science: A First Course، Cold Spring Harbor Press، ISBN 978-0-87969-636-8.
- Ridley, Matt (2006)، Francis Crick: discoverer of the genetic code، Ashland, OH: Eminent Lives, Atlas Books، ISBN 0-06-082333-X.
- Olby, Robert C. (2009)، Francis Crick: A Biography، Plainview, N.Y: Cold Spring Harbor Laboratory Press، ISBN 0-87969-798-9.
- Rosenfeld, Israel (2010)، DNA: A Graphic Guide to the Molecule that Shook the World، Columbia University Press، ISBN 978-0-231-14271-7.
- Schultz, Mark؛ Cannon, Zander (2009)، The Stuff of Life: A Graphic Guide to Genetics and DNA، Hill and Wang، ISBN 0-8090-8947-5.
- Stent, Gunther Siegmund؛ Watson, James (1980)، The Double Helix: A Personal Account of the Discovery of the Structure of DNA، New York: Norton، ISBN 0-393-95075-1.
- Watson, James (2004)، DNA: The Secret of Life، Random House، ISBN 978-0-09-945184-6.
- Wilkins, Maurice (2003)، The third man of the double helix the autobiography of Maurice Wilkins، Cambridge, England: University Press، ISBN 0-19-860665-6.
وصلات خارجية
- الدنـا على مشروع الدليل المفتوح
- الدنــا على موقع كيرلـي.
- التنبؤ بموقع ارتباط البروتين بالدنـا.
- لعبة لولب الدنا المزدوج، من الموقع الرسمي لجائزة نوبل.
- الدنـا من البداية موقع آخر لتعلم الدنا والوراثة من مندل حتى مشروع الجينوم البشري.
- مركز دولان لتعلم الدنــا.
- الدنا تحت المجهر الإلكتروني
- اللولب المزدوج: 50 سنة من الدنا.، مجلة نيتشر
- اللولب المزدوج 1953-2003 المركز الوطني لتعليم التقنية الحيوية.
- وحدات دراسة جينية للأساتذة الدنـا من البداية، دليل دراسة.
- دليل لاستنساخ الدنا.
- تم العثور على مفتاح لكيمياء الوراثة. نيويورك تايمز جوان 1953. أول جريدة أمريكية تغطي اكتشاف بنية الدنا.
- رسالة مكتوبة باليد من سبع صفحات أرسلها كريك لابنه ذو الـ 12 ربيعا مايكل سنة 1953 يشرح له فيها بنية الدنا. انظر ميدالية كريك تذهب تحت المطرقة مجلة نيتشر 5 أبريل 2013.
فيديوهات
- بوابة علم الأحياء
- بوابة علم الأحياء الخلوي والجزيئي
- بوابة الكيمياء
- بوابة الكيمياء الحيوية