البروتين الشوكي لفيروس كورونا
البروتين السكري الخاص بالشوكة (بالإنجليزية: Spike glycoprotein) (ويُسمى أحيانا بروتين الشوكة،[2] وسابقا E2[3]) هو أكبر البروتينات البنيوية الأربعة المتواجدة في فيروسات كورونا.[4] يتجمع بروتين الشوكة إلى مثلوثات تشكل بُنًى كبيرة تسمى الشوكات أو القسيمات الفولفية،[3] والتي تبرز على سطح الفيروس.[4][5] المظهر المميز لهذه الشوكات حين تُرى باستخدام المجهر الإلكتروني النافذ والتلوين السلبي على شكل «هالة»[6] والتي تسمى «كورونا» وهو مصطلح لاتيني يعني «التاج»، هو ما أعطى الفيروس اسم العائلة الخاص به.[2]
| البروتين الشوكي لفيروس كورونا S1 | |
|---|---|
 | |
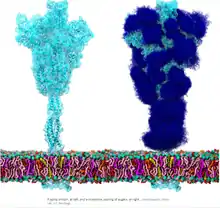
| نموذج للهيكل الخارجي لسارس-كوف-2.[1] ● الأزرق: غلاف الفيروس ● الفيروزي: البروتين الشوكي لفيروس كورونا (S) ● الأحمر: البروتين الغلافي لفيروس كورونا (E) ● الأخضر: البروتين الغشائي لفيروس كورونا (M) ● البرتقالي: جليكان | |
| معرف | |
| رمز | CoV_S1 |
| قاعدة بيانات عوائل البروتينات | PF01600 |
| إنتربرو | IPR002551 |
| البروتين الشوكي لفيروس كورونا S2 | |
|---|---|
| معرف | |
| رمز | CoV_S2 |
| قاعدة بيانات عوائل البروتينات | PF01601 |
| إنتربرو | IPR002552 |
وظيفة البروتين السكري الخاص بالشوكة هي توسط دخول الفيروس إلى الخلية المضيفة عبر التآثر مع جزيئات على السطح الخارجي للخلية وبعدها دمج الغشائين الفيروسي والخلوي. بروتين الشوكة هو بروتين دمج من الصنف 1 ويحتوي على منطقتين تعرفان باسم S1 وS2. تحتوي المنطقة S1 على نطاق الارتباط بالمستقبل الذي يرتبط بالمستقبلات على سطح الخلية. تستخدم فيروسات كورونا مجالا متنوعا من المستقبلات: يتآثر كل من سارس-كوف (الذي يسبب السارس) وسارس-كوف-2 (الذي يسبب كوفيد 19) مع المستقبل إنزيم محول للأنجيوتنسين 2 (ACE2). تحتوي المنطقة S2 على نطاق الدمج وبعض العناصر الأخرى الضرورية للاندماج مع الخلية المضيفة، واندماج الغشائين خطوة مطلوبة من أجل إصابة الخلية وتضاعف الفيروس. يحدد بروتين الشوكة مجال المضيف (الكائنات التي يمكن للفيروس إصابتها) والانتحاء الخلوي (ماهي الخلايا والأنسجة التي يمكنه إصابتها داخل الكائن).[4][5][7][8]
البروتين السكري الخاص بالشوكة شديد توليد المناعة، ووجدت أجسام مضادة لبروتين الشوكة لدى المرضى المتعافين من السارس ومن كوفيد 19. تستهدف الأجسام المضادة المحيِّدة الحواتم الموجودة في نطاق الارتباط بالمستقبل.[9] معظم المجهودات لتطوير لقاحٍ لكوفيد 19 نتيجة جائحة كوفيد 19 تهدف إلى تنشيط جهاز المناعة ضد بروتين الشوكة.[10][11][12]
البنية

بروتين الشوكة كبير جدا ويبلغ طوله عادة حوالي 1200-1400 حمض أميني،[8] وطوله في سارس-كوف-2 هو 1276 حمض أميني.[5] وهو بروتين غشائي أحادي العبور مكون من ذيل نهاية كربوكسيلية قصير في داخل الفيروس ولولب عبر غشائي ونطاق خارجي كبير بالنهاية الأمينية يبرز على السطح الخارجي للفيروس.[5][7]
يتكون البروتين السكري للشوكة من مثلوثات متجانسة، تتآثر فيها ثلاث نسخ متماثلة من البروتين عبر نطاقاتها الخارجية.[5][7] وُصف شكل بنية المثلوث على أنه شبيه «بالنفل ![]() أو الإجاصة أو البتلة».[3] يحتوي كل بروتين على منطقتين تعرفان باسم S1 وS2، وفي المثلوث المتجمع يكون جزء النهاية الأمينية للمنطقة S1 الأبعد عن سطح الفيروس في حين تشكل منطقة S2 «ساقا أو جذعا» مرنا يحتوي معظم تآثرات البروتين-بروتين التي تجعل المثلوث متماسكا في مكانه.[7]
أو الإجاصة أو البتلة».[3] يحتوي كل بروتين على منطقتين تعرفان باسم S1 وS2، وفي المثلوث المتجمع يكون جزء النهاية الأمينية للمنطقة S1 الأبعد عن سطح الفيروس في حين تشكل منطقة S2 «ساقا أو جذعا» مرنا يحتوي معظم تآثرات البروتين-بروتين التي تجعل المثلوث متماسكا في مكانه.[7]
المنطقة S1
المنطقة S1 من بروتين الشوكة السكري مسؤولة على التآثر مع الجزيئات المستقبِلة على سطح الخلية المضيفة كخطوة أولى من دخول الفيروس.[4][7] تحتوي S1 على نطاقين: نطاق النهاية الأمينية (NTD) ونطاق النهاية الكربوكسيلية (CTD)،[2][7] ويُعرفان أحيانا بالنطاقين A وB.[14] حسب نوع فيروس كورونا قد يُستخدم أحد أو كلا النطاقين كنطاق ارتباط بالمستقبل (RBD). تتنوع المستقبلات المستهدفة كثيرا، ومنها بروتينات مستقبلات سطح الخلية والسكريات مثل حمض السياليك التي تعمل كمستقبلات أو مستقبلات شريكة.[2][7] بشكل عامل يرتبط نطاق النهاية الأمينية بجزيئات السكر في حين يرتبط نطاق النهاية الكربوكسيلية بالبروتينات، باستثناء فيروس كورونا الفأري الذي يستخدم النطاق الأميني الخاص به للتآثر مع مستقبل بروتيني يسمى CEACAM1.[7] يملك نطاق النهاية الأمينية تطويا مشابها لتطوي الجالكتين لكنه يرتبط بجزيئات السكرر بطريقة مختلفة عن الجالكتينات.[7]

نطاق النهاية الكربوكسيلية مسؤول على تآثرات ميرس-كوف مع مستقبل الببتيداز نازع الببتيديل 4 (DPP-4)،[7] وتآثرات سارس-كوف[7] وسارس-كوف-2[5] مع مستقبلهما الإنزيم المحول للأنجيوتنسين 2 (ACE2). النطاق الكربوكسيلي لهذه الفيروسات يمكن تقسيمه أكثر إلى نطاقين فرعيين يعرفان: باللب والحلقة الممتدة أو نمط الارتباط بالمستقبل (RBM) أين تتموضع معظم الوحدات التي تتآثر مباشرة مع المستقبل المستهدف.[5][7] توجد اختلافات صغيرة -بشكل أساسي في نمط الارتباط بالمستقبل- بين تآثرات بروتينات الشوكة الخاصة بسارس-كوف وسارس-كوف-2 مع مستقبل ACE2.[5] تشير مقارنات البروتينات الشوكية لمختلف أنواع فيروسات كورونا إلى أن تباعدا [الإنجليزية] في منطقة نمط الارتباط بالمستقبل قد يكون مسؤولا على الاختلافات في المستقبلات المستهدفة، حتى عندما يكون لب نطاق الارتباط بالمستقل الخاص بـS1 متماثل جدا بنيويا.[7]
داخل أنساب فيروسات كورونا وكذلك عبر أجناس كورونا الرئيسية الأربعة، المنطقة S1 أقل انحفاظا من المنطقة S2، وهو ما يتوافق مع دورها في التآثر مع مستقبلات الخلية المضيفة المتخصصة بنوع الفيروس.[4][5][7] داخل المنطقة S1، نطاق النهاية الأمينية أكثر انحفاظا من نطاق النهاية الكربوكسيلية.[7]
المنطقة S2
المنطقة S2 من بروتين الشوكة السكري مسؤولة على الاندماج الغشائي [الإنجليزية] بين غلاف الفيروس والغشاء البلازمي للخلية المضيفة، وهو ما يمكن جينوم الفيروس من الدخول إلى الخلية.[5][7][8] تحتوي المنطقة S2 على ببتيد الدمج وهو سلسلة من الأحماض الأمينية معظمها كارهة للماء وظيفتها دخول غشاء الخلية المضيفة والإخلال باستقراره.[5][8] تحتوي S2 أيضا على نطاقين فرعيين يحتويان على تكرار سباعي [الإنجليزية] للأحماض الأمينية ويُعرفان باسم HR1 وHR2 وأحيانا يسميان «منطقة لب الاندماج».[5] تخضع هذه النطاقات الفرعية إلى تغيرات هيئية كبيرة أثناء عملية الاندماج لتشكيل حزمة ستة لوالب وهي ميزة خاصة ببروتينات الدمج من الصنف 1.[5][8] يُعتبر أن المنطقة S2 تشمل كذلك اللولب عبر الغشائي وذيل النهاية الكربوكسيلية المتواجد داخل الفيروس.[5]
المنطقة S2 منحفظة جدا مقارنة بالمنطقة S1 بين فيروسات كورونا.[5][7]
تعديلات ما بعد الترجمة
يُغلكز بروتين الشوكة بشكل كثيف عبر غلكزة N.[4] أبلغت دراسات بروتين شوكة سارس-كوف-2 كذلك عن غلكزة O في المنطقة S1.[17] ذيل النهاية الكربوكسيلية المتواجد داخل الفيروس غني بوحدات السيستئين وتتم إضافة البالميتيك [الإنجليزية] له.[5][18]
تُنشّط بروتينات الشوكة عبر قص حال للبروتين. تقوم بروتيازات الخلية المضيفة مثل الفيورين بقص بروتينات الشوكة في حدود المنطقتين S1-S2 ولاحقا في موقع يُعرف بـS2' في النهاية الأمينية لببتيد الدمج.[4][5][7][8]
تغير هيئي
مثل بقية بروتينات الدمج من الصنف 1، يخضع بروتين الشوكة إلى تغير هيئي كبير جدا أثناء عملية الاندماج.[4][5][7][8] تمت دراسة كلا هيئتي بنية بروتين الشوكة (هيئة قبل الاندماج وبعده) الخاصتين بعدة فيروسات كورونا وخاصة سارس-كوف-2 بواسطة المجهر الإلكتروني فائق البرودة.[5][19][20][21] لوحظت ديناميكا البروتين [الإنجليزية] أيضا داخل هيئة قبل الاندماج، والتي يمكن أن تختلف فيها التوجهات النسبية لبعض مناطق S1 بالنسبة إلى S2 في المثلوث. في الهيئة المغلقة، جميع مناطق S1 مكدسة ومتقاربة من بعضها والمنطقة التي تقوم بالاتصال والتآثر مع مستقبلات الخلية لا يمكن الوصول إليها فراغيا بواسطة المستقبل، بينما في الهيئات المفتوحة يمكن الوصول لواحد أو كلا نطاقي الارتباط بالمستقل الخاصين بـS1 وبإمكان المستقبل الارتباط بهما في هيئة الانفتاح.[5]
_(cropped).jpg.webp)
التعبير والتموقع
يتواجد الجين المشفر لبروتين الشوكة في النهاية 3' لجينوم الرنا الفيروسي موجب الدلالة إلى جانب جينات البروتينات البنيوية الثلاث الأخرى والعديد من البروتينات الملحقة المتنوعة حسب الفيروس.[4][5] يبدو أن نقل البروتين الخاص ببروتينات الشوكة يعتمد على جنس الفيروس: حين يُعبر عنها بمعزل عن البروتينات الفيروسية الأخرى، بروتينات الشوكة الخاصة بفيروسات بيتا قادرة على الوصول إلى سطح الخلية، في حين أن نظائرها في فيروسات ألفا وغاما تبقى داخل الخلية. عند تواجد بروتين الغشاء (M) يتغير نقل بروتين الشوكة وبدل خروجه إلى السطح يبقى في العنقود الحويصلي الأنبوبي [الإنجليزية] (ERGIC) وهو الموقع الذي يحدث فيه تجميع الفيروس.[18] في سارس-كوف-2، يعدل كلا بروتيني الغشاء والغلاف عملية نقل بروتين الشوكة عبر آليات مختلفة.[22]
بروتين الشوكة غير مطلوب من أجل تجميع الفيروس أو تجميع الجسيمات الشبيهة بالفيروس،[18] لكن تواجد الشوكة يمكن أن يؤثر على حجم الغلاف.[23] يعتمد إدراج بروتين الشوكة في الفيروسات أثناء التجميع والتبرعم على تآثرات البروتين بروتين مع بروتين الغشاء عبر ذيل النهاية الكربوكسيلية.[18][22] يقترح فحص الفيروسات باستخدام المجهر الإلكتروني فائق البرودة وجود حوالي 25[24] إلى 100 مثلوث شوكة في كل فيروس.[20][23]
الوظيفة

بروتين الشوكة مسؤول على دخول الفيروس إلى الخلية المضيفة، كخطوة أولية مطلوبة لتضاعف الفيروس، وهي خطوة لازمة لا يمكن الاستغناء عنها.[2] تتم هذه الخطوة على مرحلتين: أولا الارتباط بمستقبلٍ على سطح الخلية المضيفة عبر تآثرات مع المنطقة S1، وبعدها دمج الغشائين الخلوي والفيروسي بواسطة المنطقة S2.[7][8][9] يختلف مكان الاندماج حسب نوع الفيروس، إذ تستطيع بعض الأنواع الدخول من الغشاء الخلوي مباشرة بينما تدخل أنواع أخرى بواسطة الجسيمات الداخلية بعد الإدخال الخلوي.[8]
الارتباط
تسبب تآثرات نطاق الارتباط بالمستقبل الموجود في المنطقة S1 مع مستقبله الذي يستهدفه على سطح الخلية بدء عملية دخول لفيروس. تستهدف مختلف أنواع فيروسات كورونا مستقبلات سطح خلية مختلفة، وتستخدم في بعض الأحيان جزيئات سكرية مثل حمض السياليك، أو تشكل تآثرات بروتين بروتين مع بروتينات متجلية على سطح الخلية.[7][9] تختلف فيروسات كورونا كثيرا في المستقبلات التي تستهدفها وتستخدمها للدخول، ويحدد تواجد المستقبل المستهدف الذي يمكنه الارتباط مع S1 مجال الكائنات التي يمكن للفيروس إصابتها وانتحاءه الخلوي.[7][9][26]
القص الحال للبروتين
القص الحال لبروتين الشوكة -يسمى أحيانا «بالتحضير»- ضروري من أجل اندماج الغشائين. مقارنة ببروتينات الدمج من الصنف 1 الأخرى، عملية الدمج معقدة وتتطلب قصين في موقعين مختلفين: الأول في حدود المنطقين S1-S2 والثاني قي موقع S2' وذلك لتحرير ببتيد الدمج.[5][7][9] تختلف فيروسات كورونا في مرحلة دورة حياة الفيروس التي يحدث فيها القص، وبشكل محدد القص في موقع S1-S2. تقص العديد من فيروسات كورونا بروتين الشوكة في S1-S2 قبل أن تغادر الخلية بواسطة الفيورين ومحولات البروتين الطليعي [الإنجليزية] الأخرى.[7] في سارس-كوف-2، يتواجد موقع قص متعدد القواعد (PRRAR) خاص بالفيورين في الموضع S1-S2.[5][9] يمكن أن تُقص فيروسات أخرى بواسطة: بروتيازات خارج خلوية مثل الإيلاستاز أو بواسطة بروتيازات متواجدة على سطح الخلية بعد الارتباط بالمستقبل، أو بواسطة بروتيازات تتواجد في اليحلولات بعد الإدخال الخلوي.[7] يعتمد نوع البروتياز الذي يقوم بالقص على: نوع الفيروس، نوع الخلية، والبيئة المحلية التي يتواجد فيها الفيروس.[8] في سارس-كوف، بروتياز السيرين بروتياز السيرين عبر الغشائي 2 مهم من أجل هذه العملية، مع مساهمات إضافية من بروتيازي السيستئين: كاثبسين B وكاثبسين L [الإنجليزية] في اليحلولات.[8][9][33] أبلغ أن التريبسين والبروتيازات المماثلة له تساهم كذلك في القص.[8] في سارس-كوف-2، TMPRSS2 هو البروتياز الرئيسي الذي يقوم بالقص في الموقع S2' وأُبلغ أن تواجده أساسي للإصابة بالفيروس،[5][9] مع وجود دور وظيفي للكاثبسين L لكنه ليس دورا أساسيا.[33]
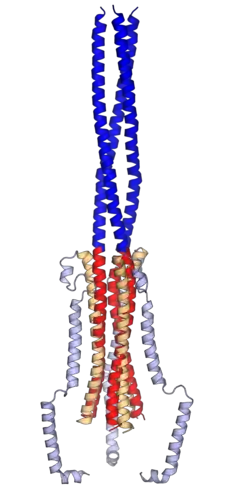
اندماج الغشاء
مثل بقية بروتينات الدمج من الصنف 1، يكون بروتين الشوكة في هيئة قبل الدمج في حالة شبه استقرار.[7] تثير عوامل متعددة تغيرا هيئيا يجعل التكرار السباعي [الإنجليزية] في المنطقة S2 يعيد التطوي إلى حزمة ستة لوالب [الإنجليزية] ممتدة وهو ما يتسبب في تآثر ببتيد الدمج مع غشاء الخلية وجلب الغشاء الفيروسي إلى مقربة منه.[5][7] الارتباط بالمستقبل والقص البروتيني الحال هما من العوامل الأساسية لإثارة التغير الهيئي، لكن توجد عوامل إثارة أخرى وهي مختلفة حسب نوع الفيروس والبيئة المحلية.[36] كمثال تقترح الدراسات المخبرية لسارس-كوف أن التغير الهيئي يعتمد على تركيز الكالسيوم.[8] على غير العادة بالنسبة لفيروسات كورونا، في فيروس كورونا الطيري الذي يصيب الطيور يمكن أن يُثار التغير الهيئي بأس هيدروجيني (pH) منخفض لوحده، بالنسبة لفيروسات أخرى الأس الهيدروجيني المنخفض ليس مثيرا في حد ذاته لكن قد يكون مطلوبا لعمل البروتيازات التي هي بدورها ضرورية من أجل الاندماج الغشائي.[8][36] يمكن أن يختلف موضع الاندماج الغشائي -في الغشاء البلازمي أو في الجسيمات الداخلية- حسب توافر هذه العوامل المثيرة للتغير الهيئي.[36] يسمح اندماج الغشائين الخلوي والفيروسي بدخول جينوم الفيروس الرنوي موجب الدلالة إلى العصارة الخلوية، والذي يبدأ بعده التعبير عن البروتينات الفيروسية.[2][4][9]
فضلا عن دمج الغشائين الفيروسي والخلوي، يمكن لبروتينات الشوكة في بعض فيروسات كورونا بدء عملية دمج الغشاء بين الخلايا المصابة وتشكيل ملتحم خلوي.[37] يمكن أن يلاحظ هذا التصرف في الخلايا المصابة في المزارع الخلوية.[38] لوحظ تشكل الملتحمات الخلوية في عيناتٍ نسيجيةٍ من المرضى المصابين بسارس-كوف، ميرس-كوف وسارس-كوف-2،[38] تشير بعض التقارير إلى اختلافٍ في تشكُّل الملتحمات الخلوية بين سارس-كوف وسارس-كوف-2 وتوعزه إلى اختلاف في التسلسل بالقرب من موقع القص S1-S2.[39][40][41]
توليد المناعة
بروتين الشوكة مولد كبير للضد لأنه مكشوف على سطح الفيروس، ويقوم الجهاز المناعي بتطوير أجسام مضادة محيِّدة له.[2][9] تعمل الغلكزة الكثيفة لبرروتين الشوكة كدرع جليكان يُخفي الحواتم عن الجهاز المناعي.[9][16] بسبب تفشي السارس في وجائحة كوفيد 19، تمت دراسة الأجسام المضادة لبروتينات الشوكة الخاصة بسارس-كوف وسارس-كوف-2 بكثافة. اكتُشف أن الأجسام المضادة لبروتينات الشوكة الخاصة بسارس-كوف وسارس-كوف-2 تستهدف حواتما تقع في انطاق الارتباط بالمستقبل[9][42] أو تتدخل في عملية التغير الهيئي.[9] تستهدف معظم الأجسام المضادة المستخلصة من الأفراد المصابين بالفيروس نطاق الارتباط بالمستقبل.[43]
الاستجابة لكوفيد 19
اللقاحات
ردا على جائحة كوفيد 19، طُورت العديد من اللقاحات لكوفيد 19 باستخدام تقنيات متنوعة بما في ذلك لقاح الرنا الرسول ولقاحات النواقل الفيروسية. معظم اللقاحات المطورة تستهدف بروتين الشوكة.[10][11][12] بناء على تقنيات استُخدمت سابقا في أبحاث اللقاحات التي استهدفت الفيروس التنفسي المخلوي وسارس-كوف، تستخدم مجهودات تطوير اللقاح فكرة إضافة طفرات لضمان استقرار هيئة قبل التطوي لبروتين الشوكة، وهو ما يسهل تطوير الأجسام المضادة للحواتم المكشوفة في هذه الهيئة.[44][45]
الأجسام المضادة وحيدة النسيلة
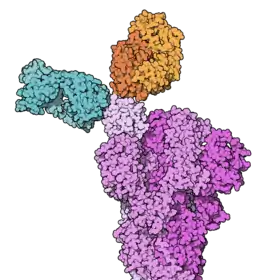
طُورت الأجسام المضادة وحيدة النسيلة التي تستهدف بروتين الشوكة كعلاجات لكوفيد 19. اعتبارا من 8 يوليو 2021، حصلت ثلاث منتجات للأجسام المضادة وحيدة النسيلة على ترخيص للاستخدام الطارئ في الولايات المتحدة:[47] بملانيفيماب/إتيسيفيماب [الإنجليزية]،[48][49] كاسيريفيماب/إيميفيماب،[50] وسوتروفيماب.[51] لا يوصى باستعمال بملانيفيماب/إتيسيفيماب في الولايات المتحدة بسبب زيادة متحورات سارس-كوف-2 التي قل تأثرها بهذه الأجسام المضادة.[47]
متحورات سارس-كوف-2
خلال جائحة كوفيد 19، تم تحديد تسلسل [الإنجليزية] جينوم فيروس سارس-كوف-2 مرات كثيرة، ونتج عن ذلك تحديد الآلاف من المتحورات المختلفة.[52] تملك العديد من هذه المتحورات طفرات استبدال لاترادفي تُغير تسلسل الأحماض الأمينية في بروتين الشوكة. في تحليل لمنظمة الصحة العالمية بتاريخ يوليو 2020، كان جين الشوكة ثاني أكثر الجينات تعرضا للطفرات بعد جين ORF1ab (الذي يشفر بروتينات فيروسية لابنيوية).[52] معدل التطور [الإنجليزية] في جين الشوكة أكبر من المعدل الإجمالي الملاحظ في كامل الجينوم.[53] تقترح تحاليل لجينومات سارس-كوف-2 أن لبعض المواقع في تسلسل بروتين الشوكة -وبالتحديد نطاق الارتباط بالمستقبل- أهمية تطورية[54] وأنها تخضع لاصطفاء إيجابي.[43][55]
الطفرات التي تحدث في بروتين الشوكة تدعو إلى القلق لأنها قد تؤثر على شدة الإعداء والانتقال، أو تسهل تجنب الجهاز المناعي.[43] حدثت الطفرة D614G بشكل مستقل في عدة سلالات فيروسية وأصبحت سائدة بين الجينومات المحدد تسلسلها،[56][57] ويمكن أن يكون لها مزايا في الإعداء والانتقال[43] ربما بسبب زيادة كثافة الشوكات على سطح الفيروس،[58] وهو ما يزيد نسبة الارتباط الجيد لهيئات البروتين أو يحسن الاستقرار،[59] لكنه لا يؤثر على اللقاحات.[60] الطفرة N614Y شائعة في متحورات ألفا وبيتا وغاما وأوميكرون الخاصة بسارس-كوف-2 وساهمت في تحسين الإعداء والانتقال،[61] وتخفيض فعالية اللقاحات،[62] وقدرة سارس-كوف-2 في إصابة أنواع قوارض جديدة.[63] تُغير الطفرة P681R موقع قص الفيورين، وهي مسؤولة على زيادة الإعداء والانتقال، والتأثير العالمي الذي أحدثه متحور دلتا.[64][65] الطفرات في الموضع E484 -بالتحديد E484k- لها صلة بتجنب المناعة وانخفاض فعالية ارتباط الأجسام المضادة.[43][53]
يتميز متحور أوميكرون الخاص بسارس-كوف-2 باحتوائه على عدد كبير غير مألوف من الطفرات في بروتين الشوكة.[66] تسبب طفرة الحذف 69-70 (Δ69-70) عدم ارتباط مسبار اختبار تفاعل البوليمراز المتسلسل (PCR) الخاص بشركة ثيرمو فيشر العلمية بجين الشوكة المستهدف وهو ما يؤدي إلى فشل في اكتشاف الجين (SGTF) في عينات سارس-كوف-2 الموجبة. استُخدم هذا التأثير كوسم لتتبع ومراقبة انتشار متحوري ألفا[67][68] وأوميكرون.[69]
معلومات خاطئة
خلال جائحة كوفيد 19، انتشرت شائعات ومعلومات خاطئة ضد التطعيم في منصات التواصل الاجتماعي تتعلق بدور بروتين الشوكة في لقاحات كوفيد 19. قيل بأن بروتينات الشوكة تملك «سمية خطيرة للخلايا» وأن لقاحات الرنا الرسول التي تحتوي على هذه البروتينات خطيرة في حد ذاتها. بروتينات الشوكة ليس لها سمية خلوية وليست خطيرة.[70][71] وقيل كذلك أن بروتينات الشوكة يتم «ذرفها وطرحها» من قبل الأفراد المطعَّمين، كتلميح خاطئ لظاهرة الذرف الفيروسي الذي يُحدثه اللقاح [الإنجليزية] وهو تأثير نادر للقاحات الفيروساات الحية الموهنة على عكس تلك المستخدمة ضد كوفيد 19. ذرف وطرح بروتينات الشوكة غير ممكن.[72][73]
التطور، الانحفاظ والتأشب
من أمثلة بروتينات الدمج من الصنف 1 التي تم تحديد خصائصها جيدا: بروتين شوكة فيروس كورونا، الراصة الدموية لفيروس الإنفلونزا، البروتين السكري gp41 لفيروس الإيدز، ويُعتقد أنها ذات صلة تطوريا.[7][74] المنطقة S2 من بروتين الشوكة المسؤولة على اندماج الغشاء أكثر انحفاظا من المنطقة S1 المسؤولة على التآثرات مع المستقبل.[4][5][7] يبدو أن المنطقة S1 خضعت لاصطفاء تنويعي معتبر.[75]
داخل المنطقة S1، نطاق النهاية الأمينية أكثر انحفاظا من نطاق النهاية الكربوكسلية. يقترح تطوي نطاق النهاية الأمينية المشابه لتطوي الجالكتين وجود صلة للنطاق الأميني مع بروتينات خلوية مماثلة له في البنية، إذ من الممكن أن يكون تطور منها عبر أخذ الجينات من المضيف.[7] اقتُرح أن نطاق النهاية الكربوكسيلية يمكن أن يكون تطور من نطاق النهاية الأمينية عبر تضاعف الجين.[7] يمكن أن تضع وضعية النطاق الكربوكسيلي المكشوفة على السطح -والتي تجعله عرضة لجهاز المناعة- هذه المنطقة تحت ضغط تطوري مرتفع.[7] تقترح مقارنة مختلف بُنى نطاقات النهاية الكربوكسيلية لمختلف فيروسات كورونا إمكانية كونها تحت اصطفاء تنويعي[76] وفي بعض حالات فيروسات كورونا ذات الصلة البعيدة التي تستخدم نفس مستقبل سطح الخلية، قد يكون استخدامها لنفس المستقبل نتيجة تطور تقاربي.[14]
المنطقة الجينومية التي تشفر الشوكة هي منطقة ساخنة للتأشيب المتماثل وغير المتماثل كذلك.[77][78][79] مناطق التعابر ليست مفردة في أحداث التأشيب -خاصة تلك المتعلقة بالشوكة- بين جنيسات فيروسات كورونا لكنها تميل لتكون مزدوجة، ما يعني أن الشوكة تنتقل إلى جُنيس آخر في وضع مماثل للكاسيت [الإنجليزية].[77] لا يوجد لحد الآن دليل على أن الشوكة انتقلت (عبر التأشيب) من جُنيس إلى آخر في جنس بيتا من فيروسات كورونا (بما في ذلك الفيروسات البشرية: سارس-كوف، سارس-كوف-2، ميرس-كوف، HKU1 وOC43).[77][78] مع ذلك، حدثت العديد من عمليات التأشيب بين مختلف جُنيسات أجناس ألفا وغاما ودلتا من فيروسات كورونا، وهي سبب معتبر يدعو للقلق واليقطة فيما يخص تطور جائحة كوفيد 19.[77]
مراجع
- Solodovnikov؛ Arkhipova (29 يوليو 2021)، "Достоверно красиво: как мы сделали 3D-модель SARS-CoV-2" [Truly beautiful: how we made the SARS-CoV-2 3D model] (باللغة الروسية)، N+1، مؤرشف من الأصل في 30 يوليو 2021، اطلع عليه بتاريخ 30 يوليو 2021.
- Deng؛ Baker (2021)، "Coronaviruses: Molecular Biology (Coronaviridae)"، Encyclopedia of Virology: 198–207، doi:10.1016/B978-0-12-814515-9.02550-9، ISBN 9780128145166.
- Masters (2006)، "The Molecular Biology of Coronaviruses"، Advances in Virus Research، 66: 193–292، doi:10.1016/S0065-3527(06)66005-3، ISBN 9780120398690، PMC 7112330، PMID 16877062.
- Wang؛ Grunewald؛ Perlman (2020)، Coronaviruses: An Updated Overview of Their Replication and Pathogenesis، Coronaviruses، Methods in Molecular Biology، ج. 2203، ص. 1–29، doi:10.1007/978-1-0716-0900-2_1، ISBN 978-1-0716-0899-9، PMC 7682345، PMID 32833200.
- Zhu؛ He؛ Yin؛ Zeng؛ Ye؛ Shi؛ Xu (14 يونيو 2021)، "Molecular biology of the SARs‐CoV‐2 spike protein: A review of current knowledge"، Journal of Medical Virology، 93 (10): 5729–5741، doi:10.1002/jmv.27132، PMC 8427004، PMID 34125455.
- "Virology: Coronaviruses"، Nature، 220 (5168): 650، نوفمبر 1968، Bibcode:1968Natur.220..650.، doi:10.1038/220650b0، PMC 7086490.
- Li (29 سبتمبر 2016)، "Structure, Function, and Evolution of Coronavirus Spike Proteins"، Annual Review of Virology، 3 (1): 237–261، doi:10.1146/annurev-virology-110615-042301، PMC 5457962، PMID 27578435.
- Millet؛ Whittaker (أبريل 2018)، "Physiological and molecular triggers for SARS-CoV membrane fusion and entry into host cells"، Virology، 517: 3–8، doi:10.1016/j.virol.2017.12.015، PMC 7112017، PMID 29275820.
- V’kovski؛ Kratzel؛ Steiner؛ Stalder؛ Thiel (مارس 2021)، "Coronavirus biology and replication: implications for SARS-CoV-2"، Nature Reviews Microbiology، 19 (3): 155–170، doi:10.1038/s41579-020-00468-6، PMC 7592455، PMID 33116300.
- Flanagan؛ Best؛ Crawford؛ Giles؛ Koirala؛ Macartney؛ Russell؛ Teh؛ Wen (02 أكتوبر 2020)، "Progress and Pitfalls in the Quest for Effective SARS-CoV-2 (COVID-19) Vaccines"، Frontiers in Immunology، 11: 579250، doi:10.3389/fimmu.2020.579250، hdl:11343/251733، PMC 7566192، PMID 33123165.
- Le؛ Cramer؛ Chen؛ Mayhew (أكتوبر 2020)، "Evolution of the COVID-19 vaccine development landscape"، Nature Reviews Drug Discovery، 19 (10): 667–668، doi:10.1038/d41573-020-00151-8، PMID 32887942، S2CID 221503034.
- Kyriakidis؛ López-Cortés؛ González؛ Grimaldos؛ Prado (ديسمبر 2021)، "SARS-CoV-2 vaccines strategies: a comprehensive review of phase 3 candidates"، NPJ Vaccines، 6 (1): 28، doi:10.1038/s41541-021-00292-w، PMC 7900244، PMID 33619260.
- Wrapp؛ Wang؛ Corbett؛ Goldsmith؛ Hsieh؛ Abiona؛ Graham؛ McLellan (13 مارس 2020)، "Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation"، Science، 367 (6483): 1260–1263، Bibcode:2020Sci...367.1260W، doi:10.1126/science.abb2507، PMC 7164637، PMID 32075877.
- Hulswit؛ de Haan؛ Bosch (2016)، "Coronavirus Spike Protein and Tropism Changes"، Advances in Virus Research، 96: 29–57، doi:10.1016/bs.aivir.2016.08.004، ISBN 9780128047361، PMC 7112277، PMID 27712627.
- Zimmer (09 أكتوبر 2020)، "The Coronavirus Unveiled"، The New York Times، مؤرشف من الأصل في 4 ديسمبر 2021، اطلع عليه بتاريخ 12 أغسطس 2021.
- Casalino؛ Gaieb؛ Goldsmith؛ Hjorth؛ Dommer؛ Harbison؛ Fogarty؛ Barros؛ Taylor؛ McLellan, Jason S.؛ Fadda, Elisa؛ Amaro, Rommie E. (28 أكتوبر 2020)، "Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein"، ACS Central Science، 6 (10): 1722–1734، doi:10.1021/acscentsci.0c01056، PMC 7523240، PMID 33140034.
- Shajahan؛ Supekar؛ Gleinich؛ Azadi (09 ديسمبر 2020)، "Deducing the N- and O-glycosylation profile of the spike protein of novel coronavirus SARS-CoV-2"، Glycobiology، 30 (12): 981–988، doi:10.1093/glycob/cwaa042، PMC 7239183، PMID 32363391.
- Ujike؛ Taguchi (03 أبريل 2015)، "Incorporation of Spike and Membrane Glycoproteins into Coronavirus Virions"، Viruses، 7 (4): 1700–1725، doi:10.3390/v7041700، PMC 4411675، PMID 25855243.
- Walls؛ Park؛ Tortorici؛ Wall؛ McGuire؛ Veesler (أبريل 2020)، "Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein"، Cell، 181 (2): 281–292.e6، doi:10.1016/j.cell.2020.02.058، PMC 7102599، PMID 32155444.
- Klein؛ Cortese؛ Winter؛ Wachsmuth-Melm؛ Neufeldt؛ Cerikan؛ Stanifer؛ Boulant؛ Bartenschlager؛ Chlanda, Petr (ديسمبر 2020)، "SARS-CoV-2 structure and replication characterized by in situ cryo-electron tomography"، Nature Communications، 11 (1): 5885، Bibcode:2020NatCo..11.5885K، doi:10.1038/s41467-020-19619-7، PMC 7676268، PMID 33208793.
- Cai؛ Zhang؛ Xiao؛ Peng؛ Sterling؛ Walsh؛ Rawson؛ Rits-Volloch؛ Chen (25 سبتمبر 2020)، "Distinct conformational states of SARS-CoV-2 spike protein"، Science، 369 (6511): 1586–1592، Bibcode:2020Sci...369.1586C، doi:10.1126/science.abd4251، PMC 7464562، PMID 32694201.
- Boson؛ Legros؛ Zhou؛ Siret؛ Mathieu؛ Cosset؛ Lavillette؛ Denolly (يناير 2021)، "The SARS-CoV-2 envelope and membrane proteins modulate maturation and retention of the spike protein, allowing assembly of virus-like particles"، Journal of Biological Chemistry، 296: 100111، doi:10.1074/jbc.RA120.016175، PMC 7833635، PMID 33229438.
- Neuman؛ Kiss؛ Kunding؛ Bhella؛ Baksh؛ Connelly؛ Droese؛ Klaus؛ Makino؛ Sawicki, Stanley G.؛ Siddell, Stuart G.؛ Stamou, Dimitrios G.؛ Wilson, Ian A.؛ Kuhn, Peter؛ Buchmeier, Michael J. (أبريل 2011)، "A structural analysis of M protein in coronavirus assembly and morphology"، Journal of Structural Biology، 174 (1): 11–22، doi:10.1016/j.jsb.2010.11.021، PMC 4486061، PMID 21130884.
- Ke؛ Oton؛ Qu؛ Cortese؛ Zila؛ McKeane؛ Nakane؛ Zivanov؛ Neufeldt؛ Cerikan, Berati؛ Lu, John M.؛ Peukes, Julia؛ Xiong, Xiaoli؛ Kräusslich, Hans-Georg؛ Scheres, Sjors H. W.؛ Bartenschlager, Ralf؛ Briggs, John A. G. (17 ديسمبر 2020)، "Structures and distributions of SARS-CoV-2 spike proteins on intact virions"، Nature، 588 (7838): 498–502، Bibcode:2020Natur.588..498K، doi:10.1038/s41586-020-2665-2، PMC 7116492، PMID 32805734.
- Goodsell؛ Voigt؛ Zardecki؛ Burley (06 أغسطس 2020)، "Integrative illustration for coronavirus outreach"، PLOS Biology، 18 (8): e3000815، doi:10.1371/journal.pbio.3000815، PMC 7433897، PMID 32760062.
- Lim؛ Ng؛ Tam؛ Liu (25 يوليو 2016)، "Human Coronaviruses: A Review of Virus–Host Interactions"، Diseases، 4 (4): 26، doi:10.3390/diseases4030026، PMC 5456285، PMID 28933406.
- Yeager؛ Ashmun؛ Williams؛ Cardellichio؛ Shapiro؛ Look؛ Holmes (يونيو 1992)، "Human aminopeptidase N is a receptor for human coronavirus 229E"، Nature، 357 (6377): 420–422، Bibcode:1992Natur.357..420Y، doi:10.1038/357420a0، PMC 7095410، PMID 1350662.
- Hofmann؛ Pyrc؛ van der Hoek؛ Geier؛ Berkhout؛ Pohlmann (31 مايو 2005)، "Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry"، Proceedings of the National Academy of Sciences، 102 (22): 7988–7993، Bibcode:2005PNAS..102.7988H، doi:10.1073/pnas.0409465102، PMC 1142358، PMID 15897467.
- Huang؛ Dong؛ Milewska؛ Golda؛ Qi؛ Zhu؛ Marasco؛ Baric؛ Sims؛ Pyrc, Krzysztof؛ Li, Wenhui؛ Sui, Jianhua (15 يوليو 2015)، "Human Coronavirus HKU1 Spike Protein Uses O -Acetylated Sialic Acid as an Attachment Receptor Determinant and Employs Hemagglutinin-Esterase Protein as a Receptor-Destroying Enzyme"، Journal of Virology، 89 (14): 7202–7213، doi:10.1128/JVI.00854-15، PMC 4473545، PMID 25926653.
- Künkel؛ Herrler (يوليو 1993)، "Structural and Functional Analysis of the Surface Protein of Human Coronavirus OC43"، Virology، 195 (1): 195–202، doi:10.1006/viro.1993.1360، PMC 7130786، PMID 8317096.
- Raj؛ Mou؛ Smits؛ Dekkers؛ Müller؛ Dijkman؛ Muth؛ Demmers؛ Zaki؛ Fouchier, Ron A. M.؛ Thiel, Volker؛ Drosten, Christian؛ Rottier, Peter J. M.؛ Osterhaus, Albert D. M. E.؛ Bosch, Berend Jan؛ Haagmans, Bart L. (مارس 2013)، "Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC"، Nature، 495 (7440): 251–254، Bibcode:2013Natur.495..251R، doi:10.1038/nature12005، PMC 7095326، PMID 23486063.
- Li؛ Moore؛ Vasilieva؛ Sui؛ Wong؛ Berne؛ Somasundaran؛ Sullivan؛ Luzuriaga؛ Greenough, Thomas C.؛ Choe, Hyeryun؛ Farzan, Michael (نوفمبر 2003)، "Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus"، Nature، 426 (6965): 450–454، Bibcode:2003Natur.426..450L، doi:10.1038/nature02145، PMC 7095016، PMID 14647384.
- "Mechanisms of SARS-CoV-2 entry into cells"، Nature Reviews Molecular Cell Biology، 23 (1): 3–20، 2022، doi:10.1038/s41580-021-00418-x، PMC 8491763، PMID 34611326.
- Walls؛ Xiong؛ Park؛ Tortorici؛ Snijder؛ Quispe؛ Cameroni؛ Gopal؛ Dai؛ Lanzavecchia, Antonio؛ Zambon, Maria؛ Rey, Félix A.؛ Corti, Davide؛ Veesler, David (فبراير 2019)، "Unexpected Receptor Functional Mimicry Elucidates Activation of Coronavirus Fusion"، Cell، 176 (5): 1026–1039.e15، doi:10.1016/j.cell.2018.12.028، PMC 6751136، PMID 30712865.
- Fan؛ Cao؛ Kong؛ Zhang (ديسمبر 2020)، "Cryo-EM analysis of the post-fusion structure of the SARS-CoV spike glycoprotein"، Nature Communications، 11 (1): 3618، Bibcode:2020NatCo..11.3618F، doi:10.1038/s41467-020-17371-6، PMC 7367865، PMID 32681106.
- White؛ Whittaker (يونيو 2016)، "Fusion of Enveloped Viruses in Endosomes"، Traffic، 17 (6): 593–614، doi:10.1111/tra.12389، PMC 4866878، PMID 26935856.
- Belouzard؛ Millet؛ Licitra؛ Whittaker (20 يونيو 2012)، "Mechanisms of Coronavirus Cell Entry Mediated by the Viral Spike Protein"، Viruses، 4 (6): 1011–1033، doi:10.3390/v4061011، PMC 3397359، PMID 22816037.
- Buchrieser؛ Dufloo؛ Hubert؛ Monel؛ Planas؛ Rajah؛ Planchais؛ Porrot؛ Guivel‐Benhassine؛ Van der Werf, Sylvie؛ Casartelli, Nicoletta؛ Mouquet, Hugo؛ Bruel, Timothée؛ Schwartz, Olivier (ديسمبر 2020)، "Syncytia formation by SARS‐CoV‐2‐infected cells"، The EMBO Journal، 39 (23): e106267، doi:10.15252/embj.2020106267، PMC 7646020، PMID 33051876.
- Zhang؛ Zheng؛ Niu؛ Zhang؛ Wang؛ Yao؛ Peng؛ Franca؛ Wang؛ Zhu, Yichao؛ Su, Yan؛ Tang, Meng؛ Jiang, Xiaoyi؛ Ren, He؛ He, Meifang؛ Wang, Yuqi؛ Gao, Lihua؛ Zhao, Ping؛ Shi, Hanping؛ Chen, Zhaolie؛ Wang, Xiaoning؛ Piacentini, Mauro؛ Bian, Xiuwu؛ Melino, Gerry؛ Liu, Liang؛ Huang, Hongyan؛ Sun, Qiang (20 أبريل 2021)، "SARS-CoV-2 spike protein dictates syncytium-mediated lymphocyte elimination"، Cell Death & Differentiation، 28 (9): 2765–2777، doi:10.1038/s41418-021-00782-3، PMC 8056997، PMID 33879858.
- Braga؛ Ali؛ Secco؛ Chiavacci؛ Neves؛ Goldhill؛ Penn؛ Jimenez-Guardeño؛ Ortega-Prieto؛ Bussani, Rossana؛ Cannatà, Antonio؛ Rizzari, Giorgia؛ Collesi, Chiara؛ Schneider, Edoardo؛ Arosio, Daniele؛ Shah, Ajay M.؛ Barclay, Wendy S.؛ Malim, Michael H.؛ Burrone, Juan؛ Giacca, Mauro (03 يونيو 2021)، "Drugs that inhibit TMEM16 proteins block SARS-CoV-2 spike-induced syncytia"، Nature، 594 (7861): 88–93، Bibcode:2021Natur.594...88B، doi:10.1038/s41586-021-03491-6، PMC 7611055، PMID 33827113.
- Lin؛ Li؛ Wang؛ Shi (يونيو 2021)، "Syncytia formation during SARS-CoV-2 lung infection: a disastrous unity to eliminate lymphocytes"، Cell Death & Differentiation، 28 (6): 2019–2021، doi:10.1038/s41418-021-00795-y، PMC 8114657، PMID 33981020.
- Premkumar؛ Segovia-Chumbez؛ Jadi؛ Martinez؛ Raut؛ Markmann؛ Cornaby؛ Bartelt؛ Weiss؛ Park, Yara؛ Edwards, Caitlin E.؛ Weimer, Eric؛ Scherer, Erin M.؛ Rouphael, Nadine؛ Edupuganti, Srilatha؛ Weiskopf, Daniela؛ Tse, Longping V.؛ Hou, Yixuan J.؛ Margolis, David؛ Sette, Alessandro؛ Collins, Matthew H.؛ Schmitz, John؛ Baric, Ralph S.؛ de Silva, Aravinda M. (11 يونيو 2020)، "The receptor binding domain of the viral spike protein is an immunodominant and highly specific target of antibodies in SARS-CoV-2 patients"، Science Immunology، 5 (48): eabc8413، doi:10.1126/sciimmunol.abc8413، PMC 7292505، PMID 32527802.
- Harvey؛ Carabelli؛ Jackson؛ Gupta؛ Thomson؛ Harrison؛ Ludden؛ Reeve؛ Rambaut؛ Peacock, Sharon J.؛ Robertson, David L. (يوليو 2021)، "SARS-CoV-2 variants, spike mutations and immune escape"، Nature Reviews Microbiology، 19 (7): 409–424، doi:10.1038/s41579-021-00573-0، PMC 8167834، PMID 34075212.
- Fauci (09 أبريل 2021)، "The story behind COVID-19 vaccines"، Science، 372 (6538): 109، Bibcode:2021Sci...372..109F، doi:10.1126/science.abi8397، PMID 33833099.
- Koenig؛ Schmidt (17 يونيو 2021)، "Spike D614G — A Candidate Vaccine Antigen Against Covid-19"، New England Journal of Medicine، 384 (24): 2349–2351، doi:10.1056/NEJMcibr2106054، PMID 34133867.
- Hansen؛ Baum؛ Pascal؛ Russo؛ Giordano؛ Wloga؛ Fulton؛ Yan؛ Koon؛ Patel, Krunal؛ Chung, Kyung Min؛ Hermann, Aynur؛ Ullman, Erica؛ Cruz, Jonathan؛ Rafique, Ashique؛ Huang, Tammy؛ Fairhurst, Jeanette؛ Libertiny, Christen؛ Malbec, Marine؛ Lee, Wen-yi؛ Welsh, Richard؛ Farr, Glen؛ Pennington, Seth؛ Deshpande, Dipali؛ Cheng, Jemmie؛ Watty, Anke؛ Bouffard, Pascal؛ Babb, Robert؛ Levenkova, Natasha؛ Chen, Calvin؛ Zhang, Bojie؛ Romero Hernandez, Annabel؛ Saotome, Kei؛ Zhou, Yi؛ Franklin, Matthew؛ Sivapalasingam, Sumathi؛ Lye, David Chien؛ Weston, Stuart؛ Logue, James؛ Haupt, Robert؛ Frieman, Matthew؛ Chen, Gang؛ Olson, William؛ Murphy, Andrew J.؛ Stahl, Neil؛ Yancopoulos, George D.؛ Kyratsous, Christos A. (21 أغسطس 2020)، "Studies in humanized mice and convalescent humans yield a SARS-CoV-2 antibody cocktail"، Science، 369 (6506): 1010–1014، Bibcode:2020Sci...369.1010H، doi:10.1126/science.abd0827، PMC 7299284، PMID 32540901.
- "Therapeutic Management of Nonhospitalized Adults With COVID-19"، Covid-19 Treatment Guidelines، National Institutes of Health، مؤرشف من الأصل في 4 ديسمبر 2021.
- "etesevimab"، IUPHAR/BPS Guide to Pharmacology، مؤرشف من الأصل في 4 ديسمبر 2021، اطلع عليه بتاريخ 10 فبراير 2021.
- "Lilly announces agreement with U.S. government to supply 300,000 vials of investigational neutralizing antibody bamlanivimab (LY-CoV555) in an effort to fight COVID-19"، Eli Lilly and Company (Press release)، 28 أكتوبر 2020، مؤرشف من الأصل في 10 ديسمبر 2021.
- "Casirivimab injection, solution, concentrate Imdevimab injection, solution, concentrate REGEN-COV- casirivimab and imdevimab kit"، DailyMed، مؤرشف من الأصل في 4 يناير 2021، اطلع عليه بتاريخ 18 مارس 2021.
- "Sotrovimab injection, solution, concentrate"، DailyMed، مؤرشف من الأصل في 4 نوفمبر 2021، اطلع عليه بتاريخ 15 يونيو 2021.
- Koyama؛ Platt؛ Parida (01 يوليو 2020)، "Variant analysis of SARS-CoV-2 genomes"، Bulletin of the World Health Organization، 98 (7): 495–504، doi:10.2471/BLT.20.253591، PMC 7375210، PMID 32742035.
- Winger؛ Caspari (27 مايو 2021)، "The Spike of Concern—The Novel Variants of SARS-CoV-2"، Viruses، 13 (6): 1002، doi:10.3390/v13061002، PMC 8229995، PMID 34071984.
- Saputri؛ Li؛ van Eerden؛ Rozewicki؛ Xu؛ Ismanto؛ Davila؛ Teraguchi؛ Katoh؛ Standley, Daron M. (17 سبتمبر 2020)، "Flexible, Functional, and Familiar: Characteristics of SARS-CoV-2 Spike Protein Evolution"، Frontiers in Microbiology، 11: 2112، doi:10.3389/fmicb.2020.02112، PMC 7527407، PMID 33042039.
- Cagliani؛ Forni؛ Clerici؛ Sironi (يونيو 2020)، "Computational Inference of Selection Underlying the Evolution of the Novel Coronavirus, Severe Acute Respiratory Syndrome Coronavirus 2"، Journal of Virology، 94 (12): e00411-20، doi:10.1128/JVI.00411-20، PMC 7307108، PMID 32238584.
- Isabel؛ Graña-Miraglia؛ Gutierrez؛ Bundalovic-Torma؛ Groves؛ Isabel؛ Eshaghi؛ Patel؛ Gubbay؛ Poutanen, Tomi؛ Guttman, David S.؛ Poutanen, Susan M. (ديسمبر 2020)، "Evolutionary and structural analyses of SARS-CoV-2 D614G spike protein mutation now documented worldwide"، Scientific Reports، 10 (1): 14031، Bibcode:2020NatSR..1014031I، doi:10.1038/s41598-020-70827-z، PMC 7441380، PMID 32820179.
- Korber؛ Fischer؛ Gnanakaran؛ Yoon؛ Theiler؛ Abfalterer؛ Hengartner؛ Giorgi؛ Bhattacharya؛ Foley, Brian؛ Hastie, Kathryn M.؛ Parker, Matthew D.؛ Partridge, David G.؛ Evans, Cariad M.؛ Freeman, Timothy M.؛ de Silva, Thushan I.؛ McDanal, Charlene؛ Perez, Lautaro G.؛ Tang, Haili؛ Moon-Walker, Alex؛ Whelan, Sean P.؛ LaBranche, Celia C.؛ Saphire, Erica O.؛ Montefiori, David C.؛ Angyal, Adrienne؛ Brown, Rebecca L.؛ Carrilero, Laura؛ Green, Luke R.؛ Groves, Danielle C.؛ Johnson, Katie J.؛ Keeley, Alexander J.؛ Lindsey, Benjamin B.؛ Parsons, Paul J.؛ Raza, Mohammad؛ Rowland-Jones, Sarah؛ Smith, Nikki؛ Tucker, Rachel M.؛ Wang, Dennis؛ Wyles, Matthew D. (أغسطس 2020)، "Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus"، Cell، 182 (4): 812–827.e19، doi:10.1016/j.cell.2020.06.043، PMC 7332439، PMID 32697968.
- Zhang؛ Jackson؛ Mou؛ Ojha؛ Peng؛ Quinlan؛ Rangarajan؛ Pan؛ Vanderheiden؛ Suthar, Mehul S.؛ Li, Wenhui؛ Izard, Tina؛ Rader, Christoph؛ Farzan, Michael؛ Choe, Hyeryun (ديسمبر 2020)، "SARS-CoV-2 spike-protein D614G mutation increases virion spike density and infectivity"، Nature Communications، 11 (1): 6013، doi:10.1038/s41467-020-19808-4، PMC 7693302، PMID 33243994.
- Jackson؛ Zhang؛ Farzan؛ Choe (يناير 2021)، "Functional importance of the D614G mutation in the SARS-CoV-2 spike protein"، Biochemical and Biophysical Research Communications، 538: 108–115، doi:10.1016/j.bbrc.2020.11.026، PMC 7664360، PMID 33220921.
- McAuley (أكتوبر 2020)، "Experimental and in silico evidence suggests vaccines are unlikely to be affected by D614G mutation in SARS-CoV-2 spike protein"، npj Vaccines، 5: 96، doi:10.1038/s41541-020-00246-8، PMC 7546614، PMID 33083031.
- Liu (نوفمبر 2021)، "The N501Y Spike substitution enhances SARS-CoV-2 infection and transmission"، Nature، doi:10.1038/s41586-021-04245-0.
- Abdool Karim (2021)، "New SARS-CoV-2 variants — clinical, public health, and vaccine implications"، New England Journal of Medicine، doi:10.1056/NEJMc2100362.
- Kuiper (2021)، "But Mouse, you are not alone: On some severe acute respiratory syndrome coronavirus 2 variants infecting mice"، ILAR Journal، doi:10.1093/ilar/ilab031.
- Callaway (2021)، "The mutation that helps Delta spread like wildfire"، Nature، doi:10.1038/d41586-021-02275-2.
- Peacock (2021)، "The SARS-CoV-2 variants associated with infections in India, B.1.617, show enhanced Spike cleavage by furin"، bioRxiv، doi:10.1101/2021.05.28.446163.
- "Classification of Omicron (B.1.1.529): SARS-CoV-2 Variant of Concern"، World Health Organization، 26 نوفمبر 2021، مؤرشف من الأصل في 26 نوفمبر 2021، اطلع عليه بتاريخ 26 نوفمبر 2021.
- "S-Gene Target Failure as a Marker of Variant B.1.1.7 Among SARS-CoV-2 Isolates in the Greater Toronto Area, December 2020 to March 2021"، JAMA، 325 (20): 2115–2116، 25 مايو 2021، doi:10.1001/jama.2021.5607، ISSN 0098-7484، PMC 8033504، PMID 33830171، مؤرشف من الأصل في 14 ديسمبر 2021.
- European Centre for Disease Prevention and Control, World Health Organization (03 مارس 2021)، Methods for the detection and identification of SARS-CoV-2 variants (Technical report)، Stockholm: European Centre for Disease Prevention and Control، Diagnostic screening assays of known VOCs، مؤرشف من الأصل في 16 ديسمبر 2021.
- SARS-CoV-2 variants of concern and variants under investigation in England Variant of concern: Omicron, VOC21NOV-01 (B.1.1.529), technical briefing 30 (PDF) (Briefing)، Public Health England، 03 ديسمبر 2021، GOV-10547، مؤرشف من الأصل (PDF) في 11 ديسمبر 2021، اطلع عليه بتاريخ 15 ديسمبر 2021.
- "COVID-19 vaccines are not 'cytotoxic'" (Fact check)، Reuters، 18 يونيو 2021، مؤرشف من الأصل في 13 يناير 2022.
- Gorski DH (24 مايو 2021)، "The 'deadly' coronavirus spike protein (according to antivaxxers)"، Science-Based Medicine، مؤرشف من الأصل في 15 يناير 2022.
- "Debunking the anti-vaccine hoax about 'vaccine shedding'"، PolitiFact، 05 مايو 2021، مؤرشف من الأصل في 3 ديسمبر 2021، اطلع عليه بتاريخ 11 مايو 2021.
- "The Latest Anti-Vax Myth: 'Vaccine Shedding'"، MedPage Today، 29 أبريل 2021، مؤرشف من الأصل في 6 ديسمبر 2021، اطلع عليه بتاريخ 11 مايو 2021.
- Vance؛ Lee (يوليو 2020)، "Virus and eukaryote fusogen superfamilies"، Current Biology، 30 (13): R750–R754، doi:10.1016/j.cub.2020.05.029، PMC 7336913، PMID 32634411.
- Li (01 مارس 2012)، "Evidence for a Common Evolutionary Origin of Coronavirus Spike Protein Receptor-Binding Subunits"، Journal of Virology، 86 (5): 2856–2858، doi:10.1128/jvi.06882-11، PMC 3302248، PMID 22205743.
- Shang؛ Zheng؛ Yang؛ Liu؛ Geng؛ Luo؛ Zhang؛ Li (23 أبريل 2018)، "Cryo-EM structure of infectious bronchitis coronavirus spike protein reveals structural and functional evolution of coronavirus spike proteins"، PLOS Pathogens، 14 (4): e1007009، doi:10.1371/journal.ppat.1007009، PMC 5933801، PMID 29684066.
- Nikolaidis؛ Markoulatos؛ Van de Peer؛ Oliver؛ Amoutzias (12 أكتوبر 2021)، Hepp, Crystal (المحرر)، "The neighborhood of the Spike gene is a hotspot for modular intertypic homologous and non-homologous recombination in Coronavirus genomes"، Molecular Biology and Evolution (باللغة الإنجليزية): msab292، doi:10.1093/molbev/msab292، ISSN 0737-4038، PMC 8549283، PMID 34638137، مؤرشف من الأصل في 23 أكتوبر 2021.
- Bobay؛ O’Donnell؛ Ochman (17 ديسمبر 2020)، Tang, Hua (المحرر)، "Recombination events are concentrated in the spike protein region of Betacoronaviruses"، PLOS Genetics (باللغة الإنجليزية)، 16 (12): e1009272، doi:10.1371/journal.pgen.1009272، ISSN 1553-7404، PMC 7775116، PMID 33332358.
- Yang؛ Yan؛ Hall؛ Jiang (13 أبريل 2021)، Rasmus, Nielsen (المحرر)، "Characterizing Transcriptional Regulatory Sequences in Coronaviruses and Their Role in Recombination"، Molecular Biology and Evolution (باللغة الإنجليزية)، 38 (4): 1241–1248، doi:10.1093/molbev/msaa281، ISSN 1537-1719، PMC 7665640، PMID 33146390، مؤرشف من الأصل في 23 ديسمبر 2021.
- بوابة طب
- بوابة الكيمياء الحيوية
- بوابة عقد 2020
- بوابة كوفيد-19
- بوابة علم الفيروسات